【论文笔记】《Virtual histological staining of unlabelled tissueautofluorescence images via deep learning》
摘要
使用生成性对抗网络模型训练的卷积神经网络,将未标记组织切片的宽场自体荧光图像,转换为相同样本的组织学染色版本的亮场图像。这些图像和标准组织学染色进行了盲对照,并涉及不同类型的染色,结果显示没有重大不一致。
背景
临床上确定组织切片的金标准图像是一个艰苦的过程,要使用多种试剂,并对组织产生不可逆转的影响。最近有人尝试使用不同的成像模式来改变这一工作流程,这将依赖光谱学的研究。病理学家和肿瘤分类软件包通常经过培训,用于检查组织染色的组织样本,出于这一原因,上述一些技术也得到了增强,以创建假苏木精和伊红(H&E)图像。
本文干的事如下图所示:

我们通过对包括唾液腺、甲状腺、肾脏、肝脏和肺在内的无标记人体组织样本进行成像,展示了这种基于深度学习的虚拟组织学染色方法。网络输出生成的等效图像与使用三种不同染色剂标记的相同样本的图像非常匹配,即H&E(唾液腺和甲状腺)、琼斯染色剂(肾脏)和马森三色染色剂(肝脏和肺)。病理学家能够在我们的虚拟染色技术生成的图像中识别组织病理学特征,并与相同样本的组织病理学染色图像高度一致。
结果
【组织样本的虚拟染色】作者展示了唾液腺的HE、肾癌的JS、肝癌的染色结果。这些结果表明,深层网络可以从无标签样本的单一自体荧光图像中推断不同组织类型所用的不同类型组织学染色方法的染色模式。由表1可看出,4个病理专家均对VS给出了比HS还高的评分,且使用VS的诊断所需时间不高于HS。
【WSIs染色效果的盲评估】我们的数据表明,病理学家能够使用两种染色技术识别组织病理学特征,并且两种技术之间高度一致,而无需明确首选的染色技术(虚拟与组织学)。
【染色标准化】在染色过程中,组织学染色的肝组织切片中的变化高于虚拟染色的组织图像。虚拟染色的着色完全是其训练的结果(和训练阶段使用的训练金标准有关),可以通过使用新染色金标准对网络进行重新训练,进一步根据病理学家的偏好进行调整,这种“改进”的培训可以从头开始,也可以通过迁移学习加速。
【迁移学习】迁移学习包含两个层面(其他组织或其他染色方式),可以使得模型更快收敛。
讨论
我们展示了使用有监督的深度学习技术对无标记组织切片进行虚拟染色的能力,它有可能为组织样本的组织学染色标准实践提供一种数字化替代方案。
未来的一个研究方向是:可以结合其他无标记成像技术和/或荧光通道,使用本文提出的基于深度学习的方法进一步增强对不同组织成分的推断(比如原文中列举的通过Cy5来凸显出黑色素瘤)。
在染色过程和相关步骤中,一些组织成分可能会丢失或变形,从而在训练阶段误导损失/成本函数,当然这对训练有素的神经网络没有太大影响。为了将这一挑战对网络性能的影响降至最低,具体做法是:在初始配准结束时,为组织染色过程前后之间图像的可接受相关性系数设置阈值(阈值大小因组织而异),并从我们的训练和验证集中消除不匹配的图像对,以确保网络学习到真实的风格转换,而不是组织染色过程对组织形态的干扰。这也更加促使人们是用虚拟染色代替原有染色(因为原有染色需要繁琐的切片制备和观察,甚至损失形态特征)。
未来的另一个研究方向是:使用一个更通用的CNN模型可以针对多种组织-染色组合进行训练。
还有一个研究方向,进一步开发自动聚焦算法,可以学会在成像阶段拒绝这些伪影。这是由于制备过程中的灰尘、杂质混入切片所导致的。
作为下一步,需要进行大规模随机临床研究,如果通过临床比较检测到诊断失败,它可以通过在发现此类失败时对其进行惩罚,来调整反馈,从而在统计学上改善其性能。
我们的对比机制源自具有单一激发带的组织自体荧光,可以探索其他用于虚拟染色无标签组织样本的对比生成方法,例如,多个激发和发射波长,以及其他成像方式,如偏振成像、定量相位显微镜、光学相干层析成像以及这些方式的组合。
方法
样本制备
为了测试我们的方法,我们使用了不同的组织-染色组合:唾液腺和甲状腺组织切片用H&E染色,肾组织切片用琼斯染色,肝和肺组织切片用马森三色染色。在WSI研究中,FFPE组织切片在自体荧光成像阶段未被覆盖。自体荧光成像后,组织样本进行如上所述的组织学染色。
数据获取
使用配备有机动台的常规荧光显微镜(IX83,Olympus)拍摄无标签组织自体荧光图像。
图像预处理和对齐
准确匹配输入图像和目标图像的FOV(Field Of View)至关重要。
第一步是找到“匹配未染色自体荧光图像和染色亮场图像”的候选特征。为此,对每个自体荧光图像(2048×2048像素)进行降采样,以匹配亮场显微镜图像的有效像素大小。这产生了1351×1351像素的未染色自体荧光组织图像,通过使得底部1%和顶部1%的像素值趋于饱和来增强对比度,并反转对比度,以更好地表示“灰度转换后WSI”的颜色图。这之后,通过将每个1351×1351像素块与从整个灰度图像中提取的相同大小的对应块相关联,计算归一化相关分数矩阵。利用这些信息,我们裁剪了原始整张亮场图像当中与之匹配的FOV,以创建目标图像。由于在两个不同的显微成像实验(自体荧光,然后是亮场)中样品放置的轻微不匹配,它们仍然无法在单个像素水平上高精度配准,这会随机导致同一样品的输入图像和目标图像之间出现轻微旋转角度。
第二步可校正自发荧光和亮场图像之间的轻微旋转角度。这是通过从图像对中提取特征向量(描述符)及其相应位置,并使用提取的描述符匹配特征来实现的。之后,使用使用“随机样本一致性(RANSAC)算法”的变种“M估计样本一致性算法”找到对应于匹配对的变换矩阵。把该变换矩阵乘在亮场图像上,得到角度校正后的图像。由于旋转角度校正,图像被进一步裁剪100个像素(每侧50个像素),以适应图像边界处未定义的像素值。
第三步,使用神经网络学习大致匹配图像之间的转换。该网络使用与下面和图2中描述的网络相同的结构。不过在图像匹配任务中,使用了较少的迭代次数,因此网络只学习颜色映射,而不学习输入图像和标签图像之间的任何空间变换。通过弹性图像配准算法,自体荧光图像通过该网络实现局部特征匹配。该算法通过从大到小的分层匹配相应的块来匹配两组图像的局部特征(自发荧光图像与亮场图像)。然后将该步骤计算出的变换图应用于每个亮场图像(角度校正)。【参见参考文献32】

综上所述,第一步是在找大体的匹配区域FOV,第二步是角度修正,第三步是精确配准。

网络结构
损失函数:
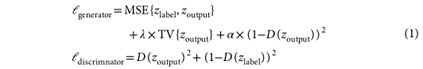
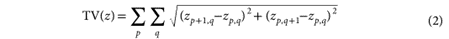
如果生成器和判别器都被成功训练,〖D(Z〗_output)最后应该是收敛到0.5附近。

上图就是生成器和判别器的结构。每一次下采样步骤都对应一个残差快,结构如下,一共有4个这种降采样结构。
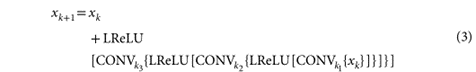
如果是WSI,那么要在激活函数后加上batch normalization。
这个是上采样过程的结构,一共也是4组这样的结构。

判别器结构在这里。
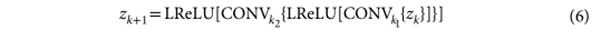
在判别器的最后,是这样的结构。
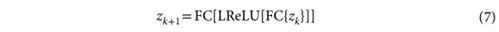
最后要把这个2048维向量通过sigmoid函数变成0到1的概率值。
做完Patch级别的分析之后,我们整合到WSI中,这就需要我们去除重叠。因此文章在此处还说明了如何整合。