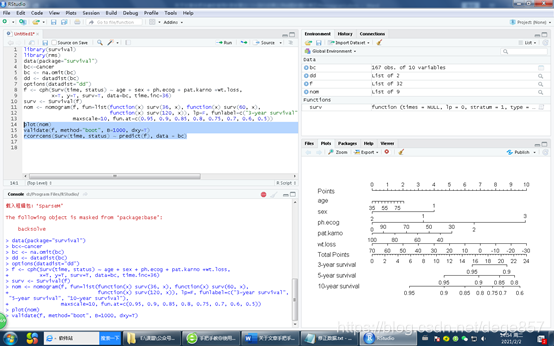
「R言語でのCOX回帰後にノモグラムを描くように教えてください」という記事が発表された後、多くの人から、検量線が醜く、古い顔が少し赤くなったというメッセージが届きました。

今日も改訂して、元のコードを確認しましょう。具体的な意味については、元の記事をご覧ください。
library(survival)
library(rms)
data(package="survival")
bc<-cancer
bc <- na.omit(bc)
dd <- datadist(bc)
options(datadist="dd")
f <- cph(Surv(time, status) ~ age + sex + ph.ecog + pat.karno +wt.loss,
x=T, y=T, surv=T, data=bc, time.inc=36)
surv <- Survival(f)
nom <- nomogram(f, fun=list(function(x) surv(36, x), function(x) surv(60, x),
function(x) surv(120, x)), lp=F, funlabel=c("3-year survival", "5-year survival", "10-year survival"),
maxscale=10, fun.at=c(0.95, 0.9, 0.85, 0.8, 0.75, 0.7, 0.6, 0.5))
plot(nom)
validate(f, method="boot", B=1000, dxy=T)
rcorrcens(Surv(time, status) ~ predict(f), data = bc)
このステップでは、内部検証と識別が完了し、キャリブレーションの準備が整います。元のコードは次のようになります。実際、画像の修正は簡単です。uの2つのパラメータを調整する必要があります。 uは生存時間を確認する期間を表し、f3のtime.incも同時に変更する必要があることに注意してください。また、mはサンプルのサンプルサイズを表します。
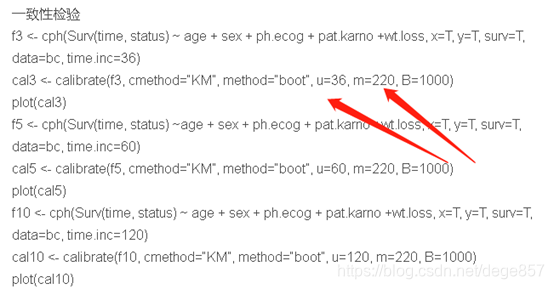
新しいコードは次のとおりです。
f3 <- cph(Surv(time, status) ~ age + sex + ph.ecog + pat.karno +wt.loss, x=T, y=T, surv=T,
data=bc, time.inc=720)
cal3 <- calibrate(f3, cmethod="KM", method="boot", u=720, m=50, B=1000)
plot(cal3)
f5<- cph(Surv(time, status) ~ age + sex + ph.ecog + pat.karno +wt.loss, x=T, y=T, surv=T,
data=bc, time.inc=360)
cal5 <- calibrate(f5, cmethod="KM", method="boot", u=360, m=50, B=1000)
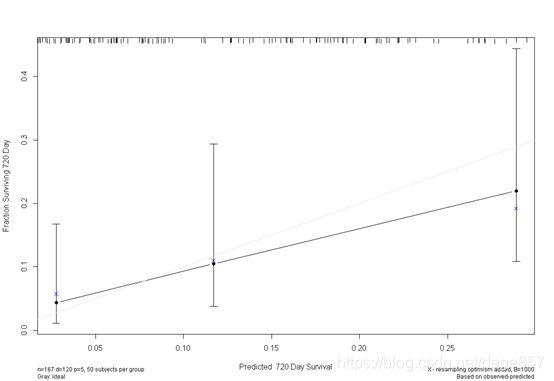
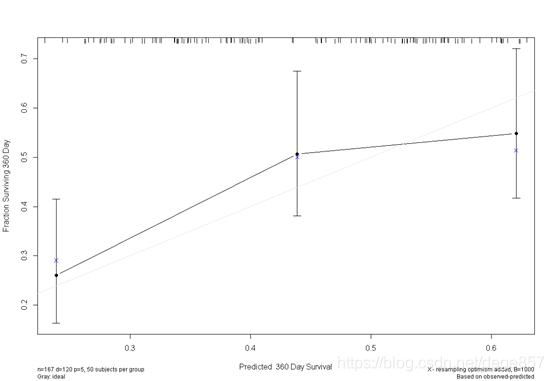
美しくして色を付ける
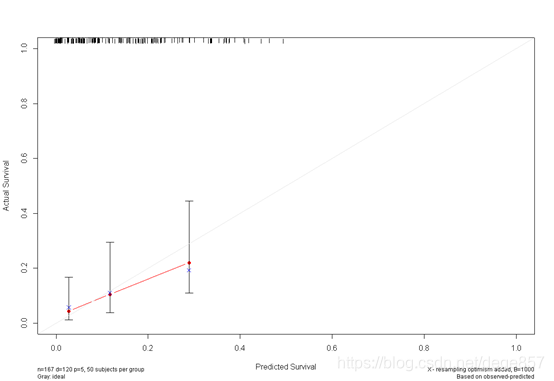
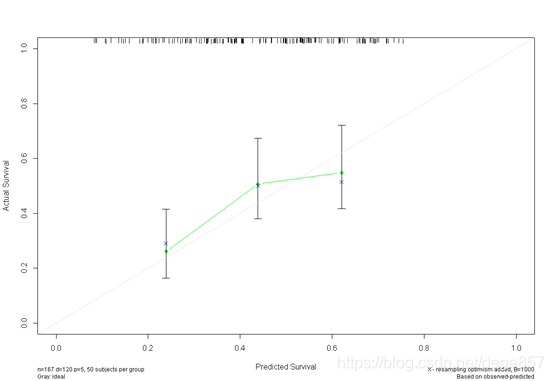
こともできます。2枚の写真を組み合わせることもできます。
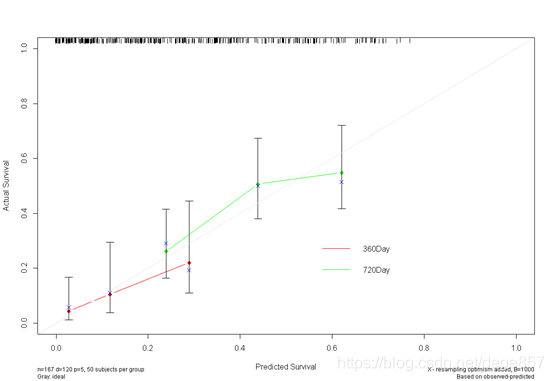
よりエキサイティングな記事については、公開アカウントに注意してください:ゼロベースの調査
