シェフィールド遺伝的アルゴリズムツールボックス
ツールボックスをインストールする
シェフィールド遺伝的アルゴリズムツールボックスは、MATLABの関数ライブラリとして、遺伝的アルゴリズムを簡単に実装できます。初めに、粒子群最適化やクジラ最適化アルゴリズムなどの群知能最適化アルゴリズムを整数領域で学びました。異なる演算子を使用して母集団内の各エージェントを反復し、微分進化などの遺伝学にも遭遇しましたアルゴリズムの改善。遺伝的アルゴリズムを最初に理解した後、私は遺伝的アルゴリズムの2進数と10進数の変換について考えてきました。これは、10進数の小数部分と2進数の数が関係するためです。シェフィールドツールボックスには、bs2rv()関数があります。組み込み関数には特定のアルゴリズムが含まれているため、アルゴリズムに必要なパラメーターに従って、特定の間隔でバイナリ配列を10進数に簡単に変換できます。原理は特別ではありません。分かりました
ツールボックスをダウンロード
オリジナルのダウンロードリンクは、オフィシャルによって無料で提供されましたが、現在は404です。必要な場合は、私に個人的に信頼してもらえます。オリジナルの公式Webサイトは次のとおりです。
http://codem.group.shef.ac.uk/index.php/ga-toolbox(1
)ネットワークディスクから取り出し、中国語の追加を記憶するために、ダウンロード後に以前の中国語を削除できます。
(2)解凍して、matlab管理ツールのフォルダー(ツールボックス)に入れます。PS私はmatlab2012aを使用しています

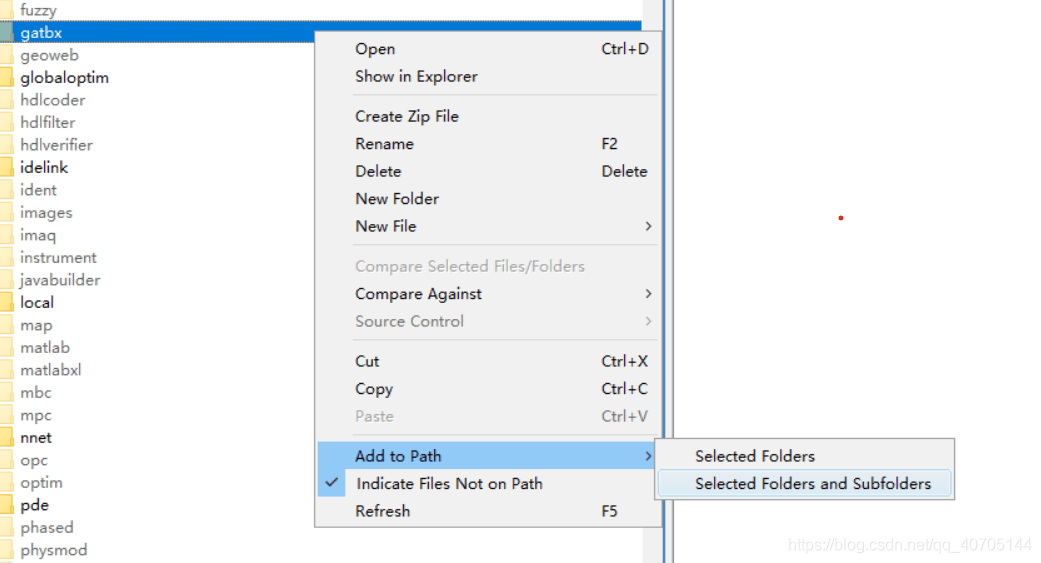
パスを追加
matlabで次のフォルダーを見つけて、すべてのファイルのパスにgatbxを追加します。私のmatlabは起動するたびに追加する必要があります。
遺伝的アルゴリズム関数
遺伝的アルゴリズムは、グループインテリジェンス最適化アルゴリズムです。母集団では、各粒子の選択、交差、突然変異の3つの操作を繰り返します。理解しにくい関数をいくつか紹介しますが、一部の関数については、理解に偏りがあるかもしれません。
ランキング()関数
ランキング()関数には3つの呼び出し形式があります
1.FitnV =ランキング(ObjV)
2.FitnV =ランキング(ObjV、RFun)
3.FitnV =ランキング(ObjV、RFun、SUBPOP)
最初の形式
ObjVは、オブジェクトの値であり、ベクトルを格納します。線形ソートと差圧= 2のシーケンスを返します。線形ソートと差圧2がデフォルトの属性です。マクロの観点からは、** [0,2] ベクトルが返されます。ObjVが大きいほど、戻り値は小さくなります。この戻り値は選択関数**で呼び出され
ます
。2 番目の形式Rfunは2桁の配列で渡す必要があります
。Rfun(1)では、線形ソートの場合、Rfun(1)は[1,2]で制御する必要があります私が理解している限り、圧力差は圧縮モードであり、Rfun(1)= 2、関数の戻り値は[0,2]で制御され、Rfun(1)= 1.5、戻り値は[0.5,1.5]で制御されます。
Rfun(2)では、Rfun(2)= 1は非線形ソートであり、Rfun(2)= 0は線形ソートです。線形ソートと非線形ソートについてはほとんど知られていませんが、ranking()関数のデフォルトは線形ソートです。
3番目の形式の
デフォルトはSUBPOP = 1で、SUBPOPはObjV中性子集団の数を定義します。このパラメーターはあまり研究されていません。
select()関数
母集団から個人を選択1.
SelCh =選択(SEL_F、Chrom、FitnV)
2. SelCh =選択(SEL_F、Chrom、FitnV、GGAP)
3. SelCh =選択(SEL_F、Chrom、FitnV、GGAP、SUBPOP)
文字列パラメーターで渡される最初の形式SEL_Fは、「rws」がルーレットであるルーレットホイール選択であり、「sus」がランダムユニバーサルサンプリングである確率的ユニバーサルサンプリングです。
2番目の形式
GGAPは、世代間のギャップ、つまり残りの確率を表します。デフォルトは1.0です。
bs2rv()関数
2進数から10進数への関数
bs2rv(Chrom、FieldD)
この関数は、遺伝的アルゴリズムツールボックスに組み込まれているアルゴリズムとして理解されています。遺伝的アルゴリズムの選択、交差、突然変異はすべてバイナリ文字列で完了するため、この関数を次のように変換する必要があります。 10進数。
FieldDは、変換が渡される必要があるフォーマットパラメータであることを理解しています。
FieldD = [len lb ub code scale lbin ubin]
len = size(Chrom、2)
lb、ubは、各変数の上限と下限です。
コードはエンコードモードで提供され、1は標準バイナリエンコードを意味し、0はグレイエンコードを意味します。
scale着信部分文字列に使用されるスケール。0はスケールが計算されることを意味します。1は対数目盛を意味します。
lbin ubinは、変換された範囲に境界が含まれるかどうかを示し、0は境界がないことを意味し、1は包含を意味します。
まとめ
この記事では、シェフィールドの遺伝的アルゴリズムツールボックスのごく一部を簡単に紹介します。不正確な点がある場合は、私的な手紙でそれらを話し合うことを歓迎します。この記事が私の学習の記録に役立つことに加えて、この記事が皆さんのお役に立てれば幸いです。
