Die Hessische Matrix hat ein breites Anwendungsspektrum in der Bildverarbeitung: im Bereich der Bildsegmentierung, einschließlich Kantenerkennung, Texturanalyse usw.
Prinzip:
Unter der Annahme, dass die Funktion des Bildpixelwerts in Bezug auf die Koordinaten (x, y) f(x, y) ist, erweitern Sie dann f(x+dx, y+dy) bei f(x0, y0), um Folgendes zu erhalten Formel

Wenn diese Formel durch eine Matrix dargestellt wird, wird der Rest verworfen
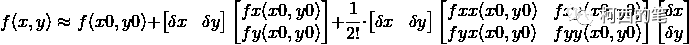
Die zweite Matrix im dritten Term auf der rechten Seite der obigen Gleichung ist die Hesse-Matrix im zweidimensionalen Raum.

Vereinfachtes 2D
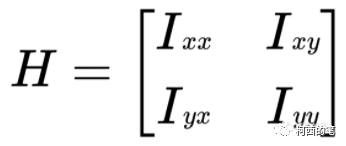
Am Beispiel eines zweidimensionalen Bildes ist die Punktstruktur im Bild isotrop, während die lineare Struktur anisotrop ist. Daher können wir die Hessische Matrix verwenden, um die lineare Struktur im Bild zu verbessern und die punktförmige Struktur und Rauschpunkte herauszufiltern. Ebenso kann es auch verwendet werden, um die Punktstruktur im Bild herauszufinden und andere Informationen herauszufiltern.
Im Allgemeinen wird die Ableitung zweiter Ordnung direkt an digitalen Grafiken durchgeführt, diese Methode weist jedoch eine geringe Robustheit auf und wird leicht durch Rauschen gestört. Es wird also die Gaußsche Funktion erhöhen.
Anwendung:
Als nächstes führen wir eine Gefäßverbesserung basierend auf dem annotierten Frangi-Filter durch.
def getkernel(kernel=7,sigma=1.0):
w = kernel//2
PI = 3.1415926
[Y,X]= np.mgrid[-w:w+1,-w:w+1]
xxGauKernel = (1-X**2/sigma**2)*np.exp(-1*(X**2+Y**2)/(2*sigma**2))*(-1/(2*PI*sigma**4))
yyGauKernel = (1-Y**2/sigma**2)*np.exp(-1*(X**2+Y**2)/(2*sigma**2))*(-1/(2*PI*sigma**4))
xyGauKernel = np.exp(-1*(X**2+Y**2)/(2*sigma**2))*((X*Y)/(2*PI*sigma**6))
return xxGauKernel,yyGauKernel,xyGauKernel
def Hessian2Dd(image,kernel=7,sigma=1.0):
xxGauKernel,yyGauKernel,xyGauKernel = getkernel(kernel,sigma)
xxDerivae = cv2.filter2D(image,-1,xxGauKernel,borderType =cv2.BORDER_CONSTANT)
yyDerivae = cv2.filter2D(image,-1,yyGauKernel,borderType =cv2.BORDER_CONSTANT)
xyDerivae = cv2.filter2D(image,-1,xyGauKernel,borderType =cv2.BORDER_CONSTANT)
return xxDerivae,xyDerivae,yyDerivae
def eig2image(Dxx,Dxy,Dyy):
Dxx=np.array(Dxx,dtype=float)
Dyy=np.array(Dyy,dtype=float)
Dxy=np.array(Dxy,dtype=float)
if (len(Dxx.shape)!=2):
print("len(Dxx.shape)!=2,不是二维数组!")
return 0
tmp = np.sqrt( (Dxx - Dyy)**2 + 4*Dxy**2)
v2x = 2*Dxy
v2y = Dyy - Dxx + tmp
mag = np.sqrt(v2x**2 + v2y**2)
i=np.array(mag!=0)
v2x[i==True] = v2x[i==True]/mag[i==True]
v2y[i==True] = v2y[i==True]/mag[i==True]
v1x = -v2y
v1y = v2x
mu1 = 0.5*(Dxx + Dyy + tmp)
mu2 = 0.5*(Dxx + Dyy - tmp)
check=abs(mu1)>abs(mu2)
Lambda1=mu1.copy()
Lambda1[check==True] = mu2[check==True]
Lambda2=mu2
Lambda2[check==True] = mu1[check==True]
Ix=v1x
Ix[check==True] = v2x[check==True]
Iy=v1y
Iy[check==True] = v2y[check==True]
return Lambda1,Lambda2,Ix,Iy
def FrangiFilter2D(I):
I=np.array(I,dtype=float)
defaultoptions = {'FrangiScaleRange':(1,10), 'FrangiScaleRatio':2, 'FrangiBetaOne':0.5, 'FrangiBetaTwo':15, 'verbose':True,'BlackWhite':True};
options=defaultoptions
sigmas=np.arange(options['FrangiScaleRange'][0],options['FrangiScaleRange'][1],options['FrangiScaleRatio'])
sigmas.sort()
beta = 2*pow(options['FrangiBetaOne'],2)
c = 2*pow(options['FrangiBetaTwo'],2)
shape=(I.shape[0],I.shape[1],len(sigmas))
ALLfiltered=np.zeros(shape)
ALLangles =np.zeros(shape)
Rb=0
S2=0
for i in range(len(sigmas)):
#Show progress
if(options['verbose']):
print('Current Frangi Filter Sigma: ',sigmas[i])
S_round=np.round(3*sigmas[i])*2+1
[Dxx,Dxy,Dyy] = Hessian2Dd(I,S_round,sigmas[i])
Dxx = pow(sigmas[i],2)*Dxx
Dxy = pow(sigmas[i],2)*Dxy
Dyy = pow(sigmas[i],2)*Dyy
[Lambda2,Lambda1,Ix,Iy]=eig2image(Dxx,Dxy,Dyy)
angles = np.arctan2(Ix,Iy)
Lambda1[Lambda1==0] = np.spacing(1)
Rb = (Lambda2/Lambda1)**2
S2 = Lambda1**2 + Lambda2**2
Ifiltered = np.exp(-Rb/beta) * (np.ones(I.shape)-np.exp(-S2/c))
if(options['BlackWhite']):
Ifiltered[Lambda1<0]=0
else:
Ifiltered[Lambda1>0]=0
ALLfiltered[:,:,i] = Ifiltered
ALLangles[:,:,i] = angles
if len(sigmas) > 1:
outIm=ALLfiltered.max(2)
else:
outIm = (outIm.transpose()).reshape(I.shape)
return outImTestergebnisse anzeigen:
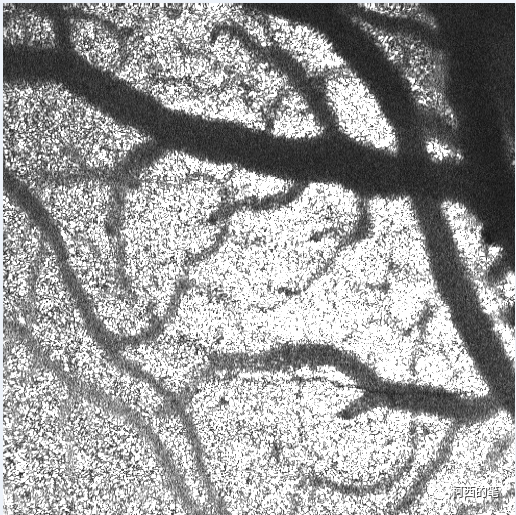

Das Prinzip der 3D-Version der Frangi-Filterung ist das gleiche wie das der 2D-Version. In diesem Artikel wird es nicht mehr direkt bereitgestellt. Leser, die es benötigen, können mir eine private Nachricht senden.