1. Información general
En la arquitectura general de RedisGraph, el modelo de almacenamiento de gráficos de RedisGraph se resume muy brevemente:
- RedisGraph usa DataBlock para almacenar atributos de nodos y bordes.
- RedisGraph utiliza una matriz dispersa para representar el gráfico, y el formato de almacenamiento de la matriz dispersa es una matriz dispersa comprimida (Compressed Sparse Row Matrix, CSR_Matrix).
¿Cómo almacena DataBlock las propiedades de nodo y borde? ¿Cuáles son los diseños específicos para representar gráficos usando matrices dispersas? Este artículo se centra en el modelo de almacenamiento de gráficos de RedisGraph.
2 Gráfico
La estructura de datos Graph en RedisGraph se define de la siguiente manera:
struct Graph {
DataBlock *nodes; // graph nodes stored in blocks
DataBlock *edges; // graph edges stored in blocks
RG_Matrix adjacency_matrix; // adjacency matrix, holds all graph connections
RG_Matrix *labels; // label matrices
RG_Matrix node_labels; // mapping of all node IDs to all labels possessed by each node
RG_Matrix *relations; // relation matrices
RG_Matrix _zero_matrix; // zero matrix
pthread_rwlock_t _rwlock; // read-write lock scoped to this specific graph
bool _writelocked; // true if the read-write lock was acquired by a writer
SyncMatrixFunc SynchronizeMatrix; // function pointer to matrix synchronization routine
GraphStatistics stats; // graph related statistics
};
-
Un gráfico de propiedades se almacena en RedisGraph.
Un gráfico de propiedades consta de nodos, relaciones y propiedades (atributos). La relación corresponde al borde en el gráfico, y tanto el nodo como el borde tienen atributos (pares clave-valor).
-
RedisGraph usa DataBlock para almacenar atributos de nodos y bordes.
-
RedisGraph usa RG_Matrix para representar gráficos.
3 bloque de datos
DataBlock es una estructura de datos contenedora para almacenar elementos del mismo tipo. De comprensión simple, DataBlock es una matriz que se puede consultar de acuerdo con el índice, pero el diseño de esta matriz es más complicado y necesita admitir operaciones eficientes de adición, eliminación, consulta y cambio de tamaño.
En RedisGraph, uno de los usos de DataBlock es almacenar atributos de nodo y borde (pares clave-valor clave-valor, por ejemplo, nombre: redis-server, ip: 127.0.0.1).
3.1 la implementación de DataBlock
Primero, veamos la definición de la estructura de datos DataBlock:
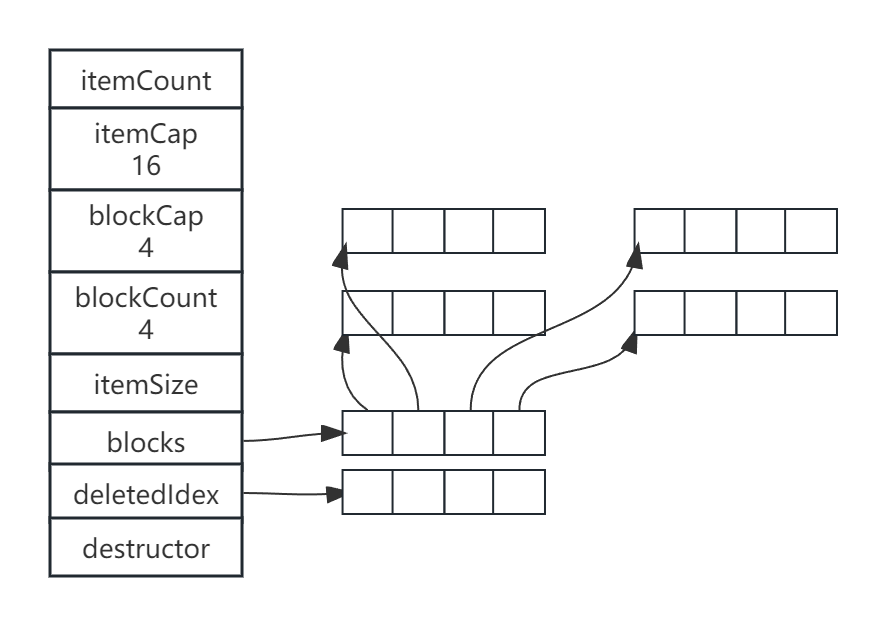
typedef struct {
uint64_t itemCount; // Number of items stored in datablock.
uint64_t itemCap; // Number of items datablock can hold.
uint64_t blockCap; // Number of items a single block can hold.
uint blockCount; // Number of blocks in datablock.
uint itemSize; // Size of a single item in bytes.
Block **blocks; // Array of blocks.
uint64_t *deletedIdx; // Array of free indicies.
fpDestructor destructor; // Function pointer to a clean-up function of an item.
} DataBlock;
typedef struct Block {
size_t itemSize; // Size of a single item in bytes.
struct Block *next; // Pointer to next block.
unsigned char data[]; // Item array. MUST BE LAST MEMBER OF THE STRUCT!
} Block;
- En la siguiente figura se muestra un DataBlock que contiene 16 elementos y 4 bloques:
-
DataBlock requiere que cada elemento tenga el mismo tamaño y, por lo general, solo almacena elementos del mismo tipo.
-
El primer bit de cada elemento se utiliza para indicar si el elemento se elimina, itemSize = sizeof(item)+1.
-
blockCount representa el número de bloques. blockCount = itemCap/blockCap.
-
Cuando la capacidad del DataBlock sea insuficiente, el DataBlock se ampliará para agregar un Bloque. En este punto, se agregará la matriz de bloques
realloc()y el puntero de bloque recién agregado se agregará al final de la matriz de bloques. Sólo se apoya la expansión, no la contracción. -
eliminadoIdx es una cola para almacenar temporalmente el índice del elemento publicado. Cuando es necesario asignar espacio para un elemento nuevo, el índice del elemento libre se obtiene preferentemente de eliminadoIdx.
-
Hay un puntero al siguiente bloque del bloque. El propósito es facilitar la operación transversal en el DataBlock.
-
La complejidad temporal de la consulta es O (1).
Para consultar el elemento idx en el DataBlock:
Block *block = idx / dataBlock->blockCap; idx = idx % dataBlock->blockCap; void* target_item = block->data + (idx * block->itemSize); -
En general, DataBlock es una estructura de datos contenedora muy eficiente para agregar, eliminar, verificar y modificar.
3.2 DataBlock para almacenamiento de gráficos
En RedisGraph, use DataBlock para almacenar atributos de nodo y borde.
Primero, echemos un vistazo a Graph_New()la declaración sobre la creación de un DataBlock.
#define NODE_CREATION_BUFFER_DEFAULT 16384
Graph *Graph_New
(
size_t node_cap,
size_t edge_cap
) {
.......
g->nodes = DataBlock_New(node_cap, node_cap, sizeof(AttributeSet), cb);
g->edges = DataBlock_New(edge_cap, edge_cap, sizeof(AttributeSet), cb);
.......
}
- En la inicialización de Graph, se crean dos bloques de datos para almacenar las propiedades del nodo y el borde respectivamente. Cada DataBlock contiene solo un Bloque. En la configuración predeterminada, cada bloque almacena hasta 16384 elementos, es decir, almacena hasta 16384 información de atributos de borde o nodo.
- Cuando el número de nodos y bordes exceda 16384, se activará la operación de expansión de DataBlock y se agregará un bloque.
3.3 Conjunto de atributos
Como se mencionó anteriormente, RedisGraph usa DataBlock para almacenar las propiedades del nodo y el borde. De hecho, esta afirmación es inexacta: DataBlock almacena punteros a atributos de nodo y borde. Después de todo, los atributos (pares clave-valor clave-valor) del nodo y el borde tienen una longitud variable, mientras que los punteros a los atributos del nodo y el borde tienen una longitud fija.
Veamos AttributeSetla definición:
typedef unsigned short Attribute_ID;
typedef struct {
Attribute_ID id; // attribute identifier
SIValue value; // attribute value
} Attribute;
typedef struct {
ushort attr_count; // number of attributes
Attribute attributes[]; // key value pair of attributes
} _AttributeSet;
typedef _AttributeSet* AttributeSet;
- Los atributos de nodo y borde son pares clave-valor clave-valor, y cada clave corresponde a un Attribute_ID (tipo corto sin firmar).
- AttributeSet es un puntero a la colección de atributos de nodos y bordes.
- Al agregar atributos al AttributeSet correspondiente al nodo o borde, primero, _AttributeSet reasigna el espacio de memoria y luego escribe el valor del atributo correspondiente.
4 RG_Matriz
RedisGraph utiliza una matriz dispersa para representar el gráfico, y el formato de almacenamiento de la matriz dispersa es una matriz dispersa comprimida (Compressed Sparse Row Matrix, CSR_Matrix). De hecho, RedisGraph no implementa completamente el código de almacenamiento de la matriz en sí, sino que encapsula GrB_Matrix en GraphBLAS.
4.1 la implementación de RG_Matrix
Comencemos con el código y analicemos RG_Matrix.
struct _RG_Matrix {
bool dirty; // Indicates if matrix requires sync
GrB_Matrix matrix; // Underlying GrB_Matrix
GrB_Matrix delta_plus; // Pending additions
GrB_Matrix delta_minus; // Pending deletions
RG_Matrix transposed; // Transposed matrix
pthread_mutex_t mutex; // Lock
};
typedef struct _RG_Matrix _RG_Matrix;
typedef _RG_Matrix *RG_Matrix;
-
RedisGraph usa GrB_Matrix en GraphBLAS para construir la matriz RG_Matrix. RG_Matrix utiliza dos formatos de almacenamiento de GrB_Matrix: matriz dispersa (GxB_SPARSE) y matriz ultra dispersa (GxB_HYPERSPARSE). Estos dos formatos de almacenamiento se presentarán en la Sección 3.2.
-
la matriz es la matriz base (primaria)
-
Su formato de almacenamiento es GxB_SPARSE o GxB_HYPERSPARSE.
-
Cuando el número de elementos distintos de cero en la matriz alcanza un cierto umbral, cambiará de GxB_HYPERSPARSE a GxB_SPARSE.
-
-
inserción y eliminación eficientes
- delta_plus y delta_minus son buffers para inserción y eliminación.
- Al insertar elementos en RG_Matrix, los elementos insertados se almacenarán temporalmente en delta_plus.
- Cuando los elementos distintos de cero en delta_plus alcancen un cierto umbral, se agruparán (utilizando el operador de suma de matrices) y se escribirán en la matriz. matriz=matriz+delta_plus.
- delta_minus es lo mismo que delta_plus, no hay más detalles.
- El formato de almacenamiento de delta_plus y delta_minus es GxB_HYPERSPARSE.
-
Para algunas matrices que almacenan relaciones de gráficos, RedisGraph almacenará sus matrices transpuestas para facilitar las consultas de gráficos.
(N0)-[A]->(N1)-[B]->(N2)<-[A]-(N3)Cuando se utiliza un patrón de búsqueda como parte de una consulta, se utiliza la matriz de transposición de A, Transpose(A).
-
Antes de que RG_Matrix realice las siguientes operaciones, es necesario adquirir el mutex de bloqueo
- Realizar operación de cambio de tamaño en RG_Matrix
- Matriz de actualización con procesamiento por lotes de delta_plus y delta_minus
4.2 GrB_Matrix en GraphBLAS
Como se mencionó en la Sección 4.1, RG_Matrix utiliza dos formatos de almacenamiento de GrB_Matrix: matriz dispersa (GxB_SPARSE) y matriz ultra dispersa (GxB_HYPERSPARSE).
-
Matriz dispersa (GxB_SPARSE)
- Almacenamiento CSR estándar
- Este formato requiere que los elementos de la matriz se almacenen en orden de filas y los elementos de cada fila se pueden almacenar desordenados.
index pointrsAlmacene la posición inicial de cada fila de elementos de datos.indicesEste es el número de columna donde se almacenan los datos de cada fila,dataen correspondencia uno a uno con los elementos de .- csr_matrix permite un acceso rápido a las filas de una matriz, pero un acceso muy lento a las columnas.
- La complejidad temporal de obtener una fila de elementos distintos de cero es O (1)

-
Matriz hiperdispersa (GxB_HYPERSPARSE)
- Variantes de la Matriz de RSE
- Usar la matriz CSR directamente es un desperdicio de almacenamiento cuando hay muy pocas filas distintas de cero.
- Agregar una matriz: se utiliza para almacenar los números de fila de filas distintas de cero y la matriz está ordenada.
- La complejidad temporal de obtener una fila de elementos distintos de cero es O (logN), donde N es el número de filas distintas de cero.
4.3 RG_Matrix para almacén de gráficos
Comencemos primero con el código:
struct Graph {
RG_Matrix adjacency_matrix; // adjacency matrix, holds all graph connections
RG_Matrix *labels; // label matrices
RG_Matrix node_labels; // mapping of all node IDs to all labels possessed by each node
RG_Matrix *relations; // relation matrices
};
- A modo de ilustración, tomamos la siguiente figura como ejemplo.

-
GraphLa estructura de datos mantiene 4 tipos de matrices:Las matrices son todas matrices cuadradas NxN, donde N es el número de vértices. La fila y la columna de la matriz corresponden al NodeID en la figura anterior.
-
matriz de adyacencia
-
La matriz de adyacencia marca todas las conexiones relacionales en el gráfico y el tipo de relación es agnóstico.
-
Adjacency_matrix Adjacency_matrix
A dj = [ 0 1 1 1 0 0 0 0 0 0 1 0 0 0 0 0 0 ] Adj = \begin{bmatrix} 0 & 1 & 1 & 1 & 0 & 0 & 1\\ 0 & 0 & 1 & 0 & 1 & 1 & 0\\ 0& 0 & 0 & 0 & 0 & 0 & 0 \\ 0 & 1 & 0 & 0 & 0 & 0 & 0 \\ 1 & 0 & 0 & 0 & 0 & 0 & 0 \\ 0 & 1 & 0 & & 0 & 0 & 0 \\ 1 & 0 & 0 & 0 & 0 & 0 & 0 \ end{bmatrix}adj _ _= 0000101100101011000001000000010000001000001000000
-
-
etiquetas
-
Para acomodar los nodos escritos, a cada etiqueta se le asigna una matriz adicional y la matriz de etiquetas es simétrica a la matriz a lo largo de la diagonal principal.
-
Hay 4 tipos de nodos en la figura anterior y hay 4 matrices de etiquetas: ciudad, persona, publicación y comentario. Sólo se muestra la matriz de comentarios.
comentario = [ 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 ] comentario = \begin{bmatrix} 0 & 0 & 0 & 0 & 0 & 0 & 0\\ 0 & 0 & 0 & 0 & 0 & 0 & 0\\ 0 & 0 & 0 & 0 & 0 & 0 y 0 \\ 0 y 0 y 0 y 1 y 0 y 0 y 0 \\ 0 y 0 y 0 y 0 y 1 y 0 y 0 \\ 0 y 0 y 0 y 0 y 0 y 1 y 0 \\ 0 y 0 y 0 y 0 y 0 y 0 y 0 \end{bmatrix}comentario _ _ _ _= 0000000000000000000000001000000010000000100000000 -
¿Por qué configurar la matriz de etiquetas?
Al hacer coincidir
(person)-[like]->(comment)esta consulta, no solo se consultará el comentario que le gusta a la persona, sino también la publicación. En este momento, se necesita la matriz de comentarios para filtrar el último nodo de comentario.
-
-
etiquetas_nodo
-
En algunos escenarios, un nodo puede corresponder a varias etiquetas. Cada etiqueta también tendrá un ID de etiqueta.
-
La matriz node_label es un mapeo: asigna todos los ID de nodo a todas las etiquetas que pertenecen a cada nodo
-
Un gráfico corresponde solo a una matriz node_label
-
Predeterminado: citylabel_ID1,personlabel_ID1,commentlabel_ID2,postlabel_ID3,node_labelvalue: comentario = [
0 1 0 0 0 0 0 0 1 0 0 0 0 1 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 1 0 0 0 0 0 0 1 0 0 0 0 0 0 1 0 0 0 ] comentario = \begin{bmatrix} 0&1&0&0&0&0; 0 & 0\\ 0 & 1 & 0 & 0 & 0 & 0 & 0\\ 1 & 0 & 0 & 0 & 0 y 0 y 0 \\ 0 y 0 y 1 y 0 y 0 y 0 y 0 \\ 0 y 0 y 1 y 0 y 0 y 0 y 0 \\ 0 y 0 y 1 y 0 y 0 y 0 y 0 \\ 0 y 0 y 0 y 1 y 0 y 0 y 0 \ end{bmatrix}comentario _ _ _ _= 0010000110000000011100000001000000000000000000000
-
-
relaciones
-
Cada tipo de relación tiene su propia matriz dedicada.
-
Hay 4 tipos de relaciones en la figura anterior y hay 4 matrices de relaciones: ubicado, amigo, hasCreator y me gusta. Solo se muestra la matriz similar.
me gusta = [ 0 0 0 0 0 0 1 0 0 0 1 1 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 ] me gusta = \begin{bmatrix} 0 & 0 & 0 & 0 & 0 & 0 & 1\\ 0 & 0 & 0 & 1 & 1 & 1 & 0\\ 0 & 0 & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & 0 & 0 & 0 & 0 \ \ 0 y 0 y 0 y 0 y 0 y 0 y 0 \end{bmatrix}me gusta _= 0000000000000000000000100000010000001000001000000
-
-