guía
La reducción de la dimensionalidad es un método común para que los profesionales del aprendizaje automático visualicen y comprendan grandes conjuntos de datos de alta dimensión. Una de las técnicas de visualización más utilizadas es t-SNE , pero su rendimiento se ve afectado por el tamaño del conjunto de datos, y usarlo correctamente puede tener un costo de aprendizaje.
UMAP es un nuevo algoritmo desarrollado por McInnes et al. Tiene muchas ventajas sobre t-SNE, sobre todo una mayor velocidad de cálculo y una mejor conservación de la estructura global de los datos. En este artículo , veremos UMAPla teoría detrás de esto para comprender mejor cómo funciona el algoritmo, cómo usarlo de manera correcta y efectiva, y t-SNEcómo se desempeña en comparación con
Entonces, ¿ UMAPqué llevar? Lo que es más importante, UMAPes rápido y escala bien con el tamaño y la dimensionalidad del conjunto de datos. Por ejemplo, el conjunto de datos MNISTUMAP de 70 000 puntos y 784 dimensiones se puede reducir en menos de 3 minutos , en comparación con los 45 minutos de . Además, tienden a conservar mejor la estructura global de los datos. Esto se puede atribuir a la sólida base teórica, que permite que el algoritmo logre un mejor equilibrio entre enfatizar la estructura local y la estructura global.scikit-learnt-SNEUMAPUMAP
1. UMAP frente a t-SNE
Antes de sumergirnos en UMAPla teoría detrás de esto, echemos un vistazo a cómo funciona en datos de alta dimensión del mundo real. La siguiente imagen muestra el uso UMAPy la reducción de un subconjunto del conjunto de datos MNIST de modat-SNE de 784 dimensiones a 3 dimensiones. Tenga en cuenta el grado en que cada clase distinta se agrupa (estructura local), mientras que clases similares (como sandalias, zapatillas y botines) tienden a agruparse (estructura global).
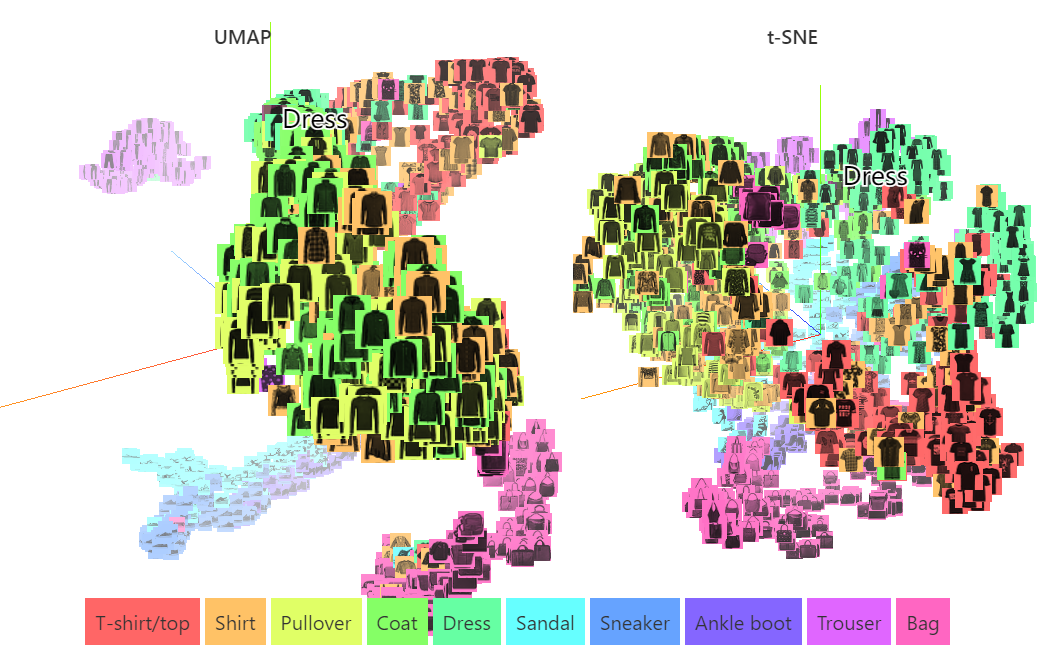
Si bien ambos algoritmos exhiben un fuerte agrupamiento local y reúnen categorías similares, UMAPsepara más claramente estos grupos de categorías similares entre sí. Vale la pena señalar que el tiempo de cálculo UMAPtarda 4 minutos, mientras que el multinúcleo t-SNEtarda 27 minutos.
2. Teoría
UMAPEl núcleo de y t-SNEes muy similar, ambos utilizan el algoritmo de diseño gráfico ( graph layout) para organizar los datos en un espacio de baja dimensión. En términos simples, UMAPprimero se construye una representación gráfica de los datos de alta dimensión y luego se optimiza el gráfico de baja dimensión para que sea lo más similar posible desde el punto de vista estructural. Aunque UMAPlas matemáticas utilizadas para construir gráficos de alta dimensión son complejas, la idea detrás de esto es muy simple.
Para construir el gráfico inicial de alta dimensión, una cosa UMAPllamada fuzzy simplicial complexEsto es realmente solo una representación de un gráfico ponderado, donde los pesos de los bordes indican la probabilidad de que dos puntos estén conectados. Para determinar la conectividad, UMAPse extiende un radio hacia afuera desde cada punto y los puntos se conectan cuando estos radios se superponen. La elección de este radio es fundamental: demasiado pequeño dará como resultado grupos pequeños y aislados, demasiado grande y todo estará completamente conectado. UMAPEsta dificultad se supera eligiendo localmente el radio en función de la distancia al enésimo vecino más cercano de cada punto. UMAPLuego, el gráfico se hace disminuyendo la probabilidad de conexiones a medida que crece el radio fuzzy. UMAPFinalmente, se asegura que la estructura local esté equilibrada con la estructura global especificando que cada punto debe estar conectado al menos con su vecino más cercano .
Una vez que se construye el gráfico de alta dimensión, UMAPel diseño de las simulaciones de baja dimensión se optimiza para que sea lo más similar posible. El proceso es t-SNEbásicamente el mismo que el , pero se usan algunos trucos para acelerar el proceso.
La clave para un uso efectivo UMAPradica en comprender la construcción del gráfico inicial de alta dimensión. Aunque la idea detrás del proceso es bastante intuitiva, el algoritmo se basa en algunas matemáticas avanzadas para proporcionar sólidas garantías teóricas sobre qué tan bien el gráfico representa realmente los datos. Los lectores interesados pueden dirigirse a: Comprensión profunda de la teoría UMAP .
3. Parámetros
Una vez que comprende UMAPla teoría detrás de esto, se vuelve mucho más fácil comprender los parámetros de un algoritmo, especialmente cuando se compara con los parámetros t-SNEen perplexity. Consideraremos dos de los parámetros más utilizados: n_neighborsy min_dist, que se utilizan efectivamente para controlar el equilibrio entre la estructura local y global en el resultado final de reducción de dimensionalidad.

n_neighbors
El parámetro más importante es n_neighborsel número de vecinos más cercanos aproximados utilizados para construir el gráfico inicial de alta dimensión. Controla efectivamente UMAPcómo equilibrar la estructura local versus la global: los valores más pequeños atraerán más atención a la estructura local al limitar la cantidad de puntos vecinos considerados al analizar datos de alta dimensión UMAP, mientras que los valores más grandes presionarán para UMAPrepresentar la estructura global mientras pierden. detalle.
min_dist
El segundo parámetro que estudiaremos es min_distla distancia mínima entre puntos en un espacio de baja dimensión. Este parámetro controla UMAPqué tan estrechamente se agrupan los puntos, los valores más bajos dan como resultado incrustaciones más estrechas. min_distLos valores más grandes UMAPempaquetarán los puntos de manera más flexible, enfocándose en cambio en preservar una topología amplia.
La siguiente visualización explora UMAPel efecto de los parámetros en la proyección 2D de datos 3D. Al cambiar los parámetros n_neighborsy min_dist, puede explorar su efecto en la proyección resultante.
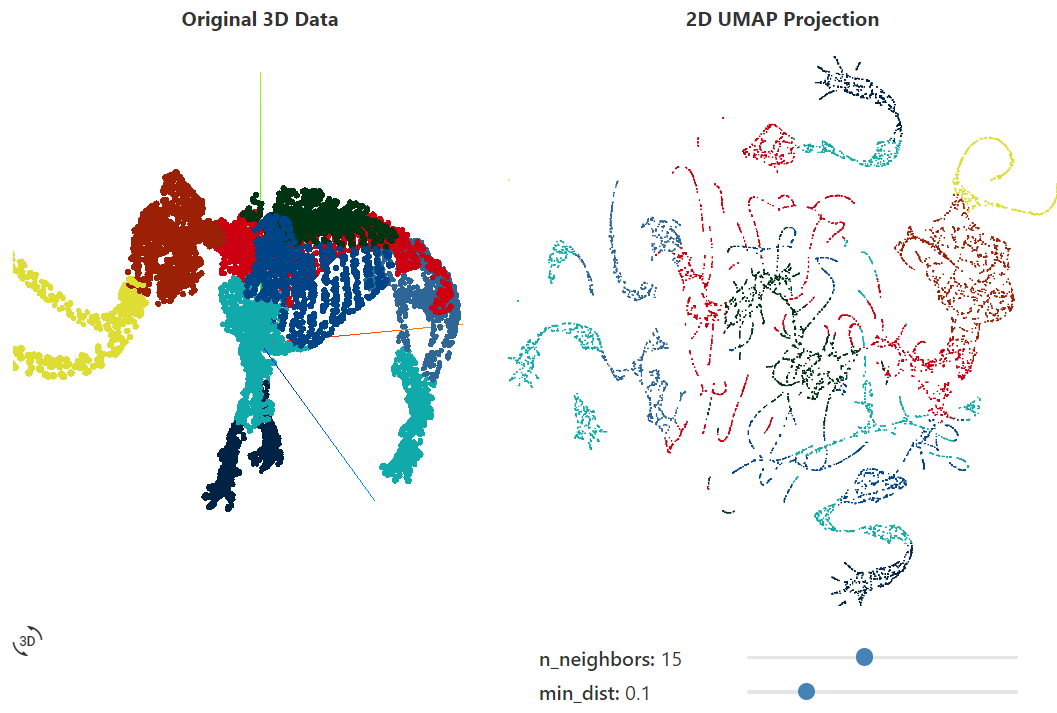
Si bien UMAPla mayoría de las aplicaciones de , involucran la proyección de datos de alta dimensión, la proyección de 3D puede servir como una analogía útil para comprender UMAPcómo priorizar la estructura global y local de acuerdo con sus parámetros. A medida que n_neighborsse UMAPconectan más y más puntos adyacentes al construir representaciones gráficas de datos de alta dimensión, se obtienen proyecciones que reflejan con mayor precisión la estructura global de los datos. A valores muy bajos, cualquier información sobre la estructura global se pierde casi por completo. A medida que min_distaumenta el parámetro, UMAPtiende a "esparcir" los puntos proyectados, lo que da como resultado una menor agrupación de los datos y menos énfasis en la estructura global.
4. UMAP frente a t-SNE 2.0
La mayor diferencia en el resultado de , en comparación con , es el equilibrio entre la estructura local y global, y generalmente es t-SNEmejor para preservar la estructura global en la proyección final. Esto significa que la relación entre clústeres puede ser más significativa que . Es importante destacar que cualquier eje o distancia dados en una dimensión inferior aún no se pueden interpretar directamente mediante técnicas como , ya que y σ necesariamente distorsionan la forma de dimensión superior de los datos cuando se proyectan a una dimensión inferior .UMAPUMAPt-SNEUMAPt-SNEPCA
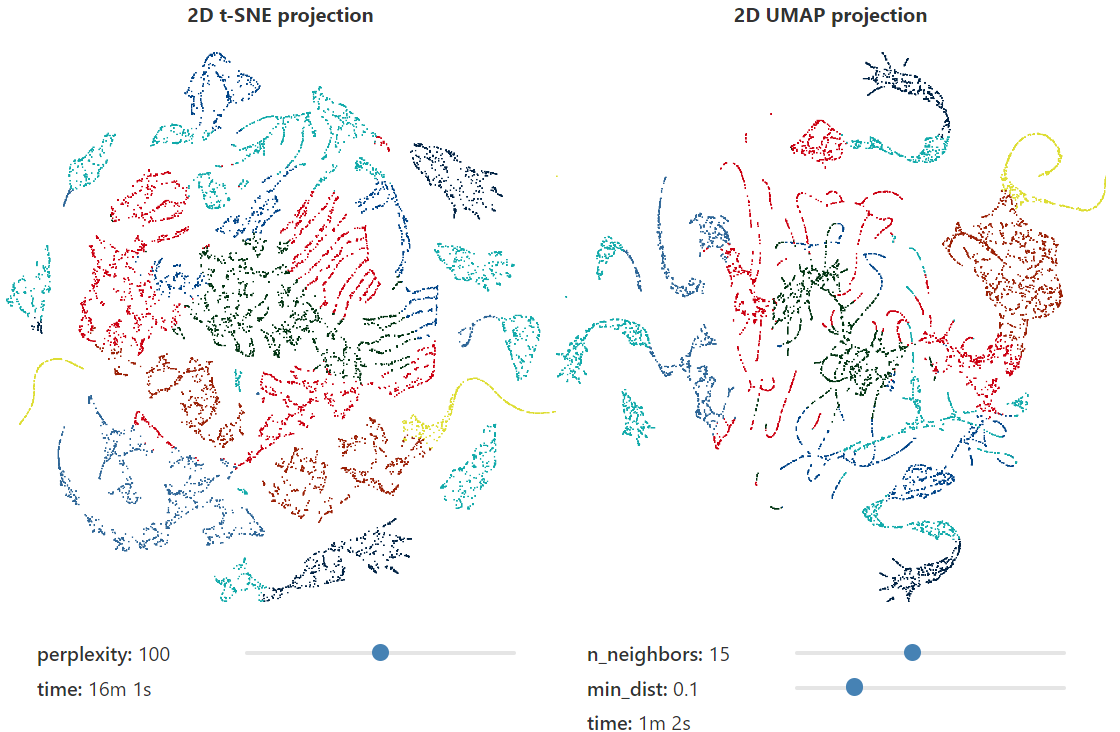
Volviendo al ejemplo del mamut 3D, podemos ver fácilmente algunas diferencias enormes entre los resultados de los dos algoritmos. Para perplexityvalores de parámetros más bajos, t-SNEexiste una tendencia a "desenrollar" los datos proyectados, conservando poca estructura global. Por el contrario, UMAPexiste una tendencia a agrupar partes adyacentes de estructuras de alta dimensión en dimensiones bajas, lo que refleja la estructura global. Tenga en cuenta que se requiere el uso t-SNEde valores extremadamente altos perplexity(~1000) para comenzar a ver la estructura global, y en perplexityvalores tan grandes, el tiempo de cálculo se extiende significativamente. También vale la pena señalar que las proyecciones varían ampliamente de una ejecución a otra t-SNE, con diferentes datos de alta dimensión que se proyectan en diferentes ubicaciones. Aunque UMAPtambién es un algoritmo estocástico, las proyecciones resultantes son sorprendentemente similares cada vez que se ejecuta y con diferentes parámetros.
Vale la pena señalar que t-SNEel UMAPrendimiento del ejemplo de juguete en la figura anterior es muy similar, excepto por el ejemplo a continuación. Curiosamente, UMAPno es posible separar dos clústeres anidados, especialmente en dimensiones altas.
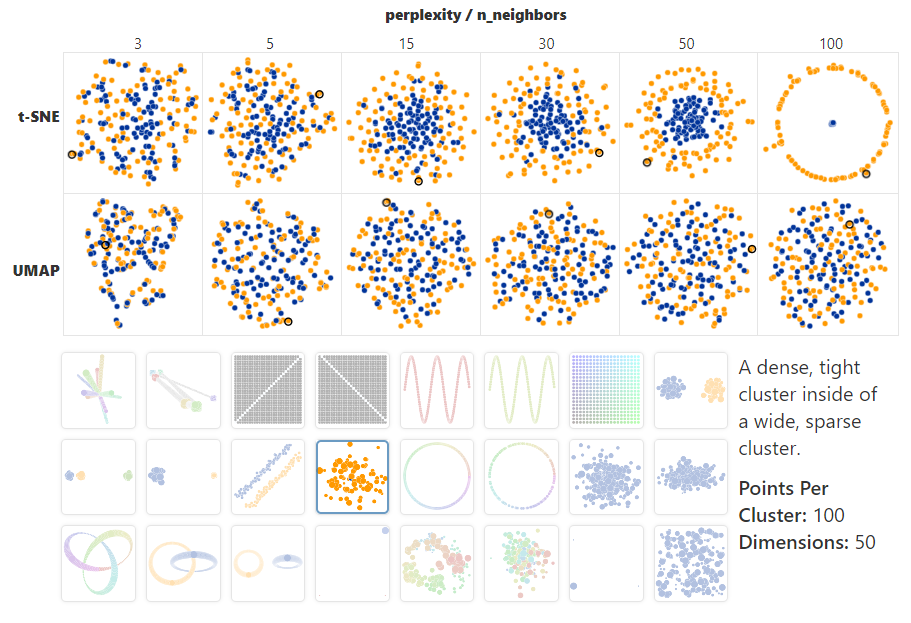
La falla del algoritmo para manejar este caso de inclusión puede deberse al UMAPuso de distancias locales en la construcción inicial del gráfico. Dado que las distancias entre los puntos de alta dimensión suelen ser muy similares (la maldición de la dimensionalidad), UMAPparece conectar los puntos exteriores de los grupos interiores con los puntos exteriores de los grupos exteriores. Esto realmente combina los dos grupos juntos.
5. entender
Si bien UMAPofrece muchas t-SNEventajas sobre los demás, de ninguna manera es una panacea, y la lectura e interpretación de sus resultados requiere precaución.
- Los hiperparámetros realmente importan
Elegir buenos hiperparámetros no es fácil y depende de los datos y objetivos. Esta es UMAPuna gran ventaja de la velocidad, y al ejecutarlo varias veces con varios hiperparámetros UMAP, puede tener una mejor idea de cómo la proyección se ve afectada por sus parámetros.
- el tamaño del grupo no tiene sentido
Como en t-SNE, el tamaño de los clústeres en relación con los demás es esencialmente sin sentido. Esto se debe al UMAPuso de un concepto de distancia local para construir su representación gráfica de alta dimensión.
- La distancia entre clústeres puede no tener ningún significado
Asimismo, la distancia entre los clústeres puede no tener sentido. Si bien es cierto que UMAPla posición global de los clústeres se conserva mejor en , la distancia entre ellos no tiene sentido. De nuevo, esto se debe al uso de distancias locales al construir el gráfico.
- El ruido aleatorio no siempre parece aleatorio
Especialmente a n_neighborsvalores bajos, se puede observar un agrupamiento espurio.
- Necesita visualizar los resultados varias veces
Dado que UMAPel algoritmo es aleatorio, diferentes ejecuciones con los mismos hiperparámetros pueden producir resultados diferentes. Además, dado que la elección de los hiperparámetros es tan importante, puede ser útil ejecutar la proyección varias veces con varios hiperparámetros.
Resumir
UMAPes una herramienta muy poderosa en el arsenal de un científico de datos, con t-SNEmuchas ventajas sobre Si bien es algo similar UMAPa t-SNEla salida de , la mayor velocidad, la mejor conservación de la estructura global y los parámetros más fáciles de entender lo convierten en una UMAPherramienta más efectiva para visualizar datos de alta dimensión. Finalmente, es importante recordar que ninguna técnica de reducción de dimensionalidad es perfecta, y esta UMAPno es la excepción. Sin embargo, al desarrollar una comprensión intuitiva de cómo funciona un algoritmo y cómo ajustar sus parámetros, podemos usar esta poderosa herramienta de manera más efectiva para visualizar y comprender grandes conjuntos de datos de alta dimensión.
Este artículo es publicado por mdnice multiplataforma