
Inhaltsverzeichnis
Missionsübersicht
Encoder-Decoder
Verbindung überspringen
Implementierungsdetails
verlustfunktion
Upsampling-Methode
Füllen oder nicht?
So funktioniert U-Net
Missionsübersicht
U-Net wurde für semantische Segmentierungsaufgaben entwickelt. Wenn ein neuronales Netzwerk ein Bild als Eingabe akzeptiert, können wir Objekte allgemein oder nach Instanz klassifizieren. Wir können die in einem Bild enthaltenen Objekte (Bildklassifizierung), die Position aller Objekte (Bildlokalisierung/semantische Segmentierung) oder die Position einzelner Objekte (Objekterkennung/Instanzsegmentierung) vorhersagen.
Die folgende Abbildung zeigt die Unterschiede zwischen diesen Computer-Vision-Aufgaben. Um das Problem zu vereinfachen, betrachten wir nur die Klassifizierung mit einer Kategorie und einem Label.

Bei einer Klassifizierungsaufgabe geben wir einen Vektor der Größe k aus, wobei k die Anzahl der Kategorien ist. In der Erkennungsaufgabe müssen wir die Vektoren x, y, Höhe, Breite und Kategorie ausgeben, die den Begrenzungsrahmen definieren.
Bei der Segmentierungsaufgabe müssen wir jedoch ein Bild mit denselben Abmessungen wie die ursprüngliche Eingabe ausgeben. Dies stellt eine erhebliche technische Herausforderung dar: Wie extrahieren neuronale Netze relevante Merkmale aus Eingabebildern und projizieren sie dann in Segmentierungsmasken?
Encoder-Decoder
Wenn Sie mit Encoder-Decoder nicht vertraut sind, empfehle ich Ihnen, diesen Artikel zu lesen:
https://towardsdatascience.com/understanding-latent-space-in-machine-learning-de5a7c687d8d
Encoder-Decoder sind verwandt, weil sie eine Ausgabe erzeugen, die dem entspricht, was wir wollen: eine Ausgabe mit den gleichen Abmessungen wie die Eingabe. Können wir das Encoder-Decoder-Konzept auf die Bildsegmentierung anwenden? Wir können eine 1D-Binärmaske generieren und Kreuzentropieverlust verwenden, um das Netzwerk zu trainieren.
Unser Netzwerk besteht aus zwei Teilen: Der Encoder extrahiert relevante Merkmale aus dem Bild, und der Decoderteil nimmt die extrahierten Merkmale und rekonstruiert die Segmentierungsmaske.
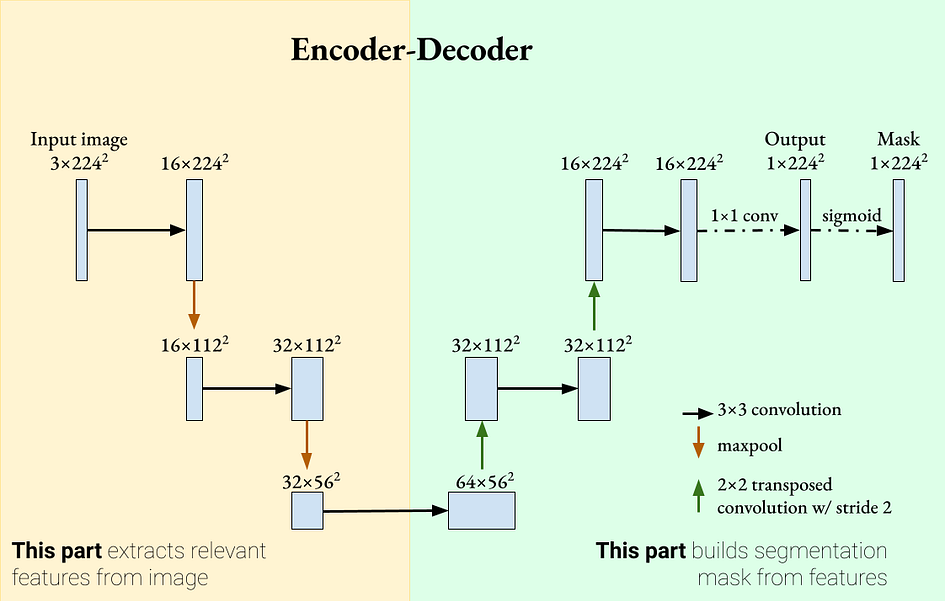
Im Encoder-Teil werden Faltungsschichten verwendet, gefolgt von ReLU und Max Pooling als Feature-Extraktoren. Im Decoderteil wird die transponierte Faltung verwendet, um die Größe der Feature-Map zu vergrößern und die Anzahl der Kanäle zu reduzieren. Padding wird verwendet, um die Größe der Feature-Maps nach der Faltungsoperation gleich zu halten.
Möglicherweise fällt Ihnen auf, dass dieses Netzwerk im Gegensatz zum Klassifizierungsnetzwerk keine vollständig verbundenen/linearen Schichten aufweist. Dies ist ein Beispiel für ein vollständig konvolutionelles Netzwerk (FCN). Es wurde gezeigt, dass FCN bei Segmentierungsaufgaben eine gute Leistung erbringt, beginnend mit dem Artikel „Fully Convolutional Networks for Semantic Segmentation“ von Shelhamer et al. [1].
Es gibt jedoch ein Problem mit diesem Netzwerk. Wenn wir weitere Encoder- und Decoder-Ebenen hinzufügen, „verkleinern“ wir die Feature-Map tatsächlich immer mehr. Daher kann der Encoder detailliertere Merkmale verwerfen, um allgemeinere Merkmale zu erhalten. Wenn es um die Segmentierung medizinischer Bilder geht, kann es wichtig sein, dass jedes Pixel als krank/normal klassifiziert wird. Wie stellen wir sicher, dass dieses Encoder-Decoder-Netzwerk sowohl allgemeine als auch detaillierte Funktionen akzeptiert?
Verbindung überspringen
https://towardsdatascience.com/introduction-to-resnets-c0a830a288a4
Da tiefe neuronale Netze beim Weiterleiten von Informationen durch aufeinanderfolgende Schichten möglicherweise bestimmte Merkmale „vergessen“, können Skip-Verbindungen diese Merkmale wieder einführen und so das Lernen leistungsfähiger machen. Skip-Verbindungen wurden in Residual-Netzwerken (ResNet) eingeführt und zeigten Klassifizierungsverbesserungen sowie gleichmäßigere Lerngradienten. Inspiriert durch diesen Mechanismus können wir Sprungverbindungen zu U-Net hinzufügen, sodass jeder Decoder die Feature-Map seines entsprechenden Encoders enthält. Dies ist ein entscheidendes Merkmal von U-Net.
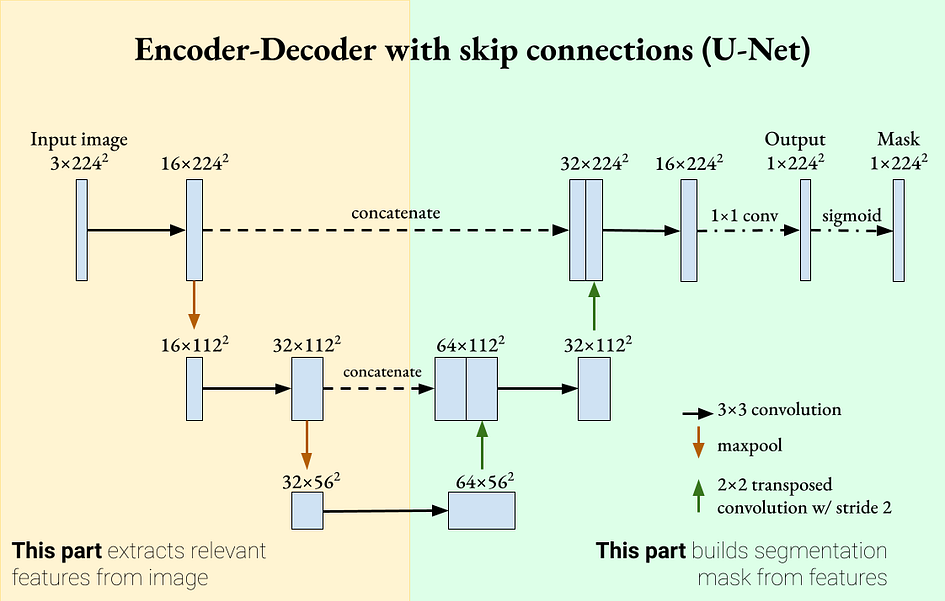
U-Net ist ein Encoder-Decoder-Segmentierungsnetzwerk mit Skip-Verbindungen. Bild vom Autor bereitgestellt. U-Net hat zwei definierende Eigenschaften:
Das Encoder-Decoder-Netzwerk extrahiert mit zunehmender Tiefe allgemeinere Merkmale.
Überspringen Sie Verbindungen und führen Sie detaillierte Funktionen im Decoder wieder ein. Diese beiden Eigenschaften bedeuten, dass U-Net sowohl detaillierte als auch allgemeine Funktionen zur Segmentierung verwenden kann. U-Net wurde ursprünglich für die biomedizinische Bildverarbeitung eingeführt, wo die Segmentierungsgenauigkeit sehr wichtig ist [2].
Implementierungsdetails

Die vorherigen Abschnitte lieferten einen sehr allgemeinen Überblick über U-Net und warum es funktioniert. Zwischen allgemeinem Verständnis und praktischer Umsetzung spielen jedoch Details eine wichtige Rolle. Hier werde ich einige Optionen für die U-Net-Implementierung skizzieren.
verlustfunktion
Da das Ziel eine binäre Maske ist (ein Pixelwert von 1 bedeutet, dass das Pixel ein Objekt enthält), ist eine häufige Verlustfunktion, die zum Vergleichen der Ausgabe mit der Grundwahrheit verwendet wird, ein kategorialer Kreuzentropieverlust (oder ein binärer Kreuzentropieverlust in im Single-Label-Fall).
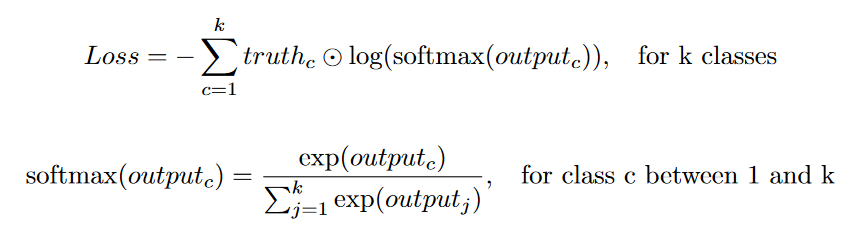
Im ursprünglichen U-Net-Artikel wurden der Verlustfunktion zusätzliche Gewichte hinzugefügt. Dieser Gewichtsparameter bewirkt zwei Dinge: Er gleicht Klassenungleichgewichte aus und verleiht den Segmentierungsgrenzen eine höhere Bedeutung. In vielen U-Net-Implementierungen, die ich gefunden habe, wird dieser zusätzliche Gewichtungsfaktor normalerweise nicht verwendet.
Eine weitere häufige Verlustfunktion ist der Würfelverlust. Der Würfelverlust misst die Ähnlichkeit zweier Bildsätze, indem deren Schnittfläche mit ihrer Gesamtfläche verglichen wird. Beachten Sie, dass Würfelverlust nicht dasselbe ist wie Schnittpunkt über Union (IOU). Sie messen ähnliche Dinge, aber mit unterschiedlichen Nennern. Je höher der Würfelkoeffizient ist, desto geringer ist der Würfelverlust.
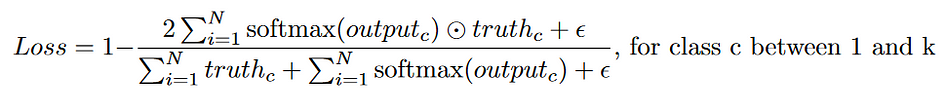
Hier wird ein Epsilon-Term hinzugefügt, um eine Division durch 0 zu vermeiden (Epsilon ist normalerweise 1). Einige Implementierungen, wie beispielsweise die von Milletari et al., quadrieren die Pixelwerte im Nenner vor der Summierung [3]. Im Vergleich zum Kreuzentropieverlust ist der Dice-Verlust sehr robust gegenüber unausgeglichenen Segmentierungsmasken, die bei biomedizinischen Bildsegmentierungsaufgaben häufig vorkommen.
Upsampling-Methode
Ein weiteres Detail ist die Wahl der Upsampling-Methode des Decoders. Hier sind einige gängige Methoden:
Bilineare Interpolation. Diese Methode verwendet lineare Interpolation, um Ausgabepixel vorherzusagen. Typischerweise folgt auf das Upsampling über diese Methode eine Faltungsschicht.
Maximales Anti-Pooling. Diese Methode ist die umgekehrte Operation des Max-Poolings. Es verwendet die Indizes der Max-Pooling-Operation und füllt diese Indizes auf den Maximalwert. Alle anderen Werte werden auf 0 gesetzt. Normalerweise folgt auf das maximale Unpooling eine Faltungsschicht, um fehlende Werte zu „glätten“.
Dekonvolution/transponierte Faltung. Es gibt viele Blogbeiträge zum Thema Dekonvolution. Ich empfehle die Lektüre dieses Artikels als guten visuellen Leitfaden.
https://towardsdatascience.com/types-of-convolutions-in-deep-learning-717013397f4d
Die Dekonvolution besteht aus zwei Schritten: Zuerst wird ein Abstand um jedes Pixel des Originalbilds hinzugefügt und dann wird eine Faltung angewendet. Im ursprünglichen U-Net wurde eine 2x2-transponierte Faltung mit einem Schritt von 2 verwendet, um die räumliche Auflösung und Kanaltiefe zu ändern.
Neuanordnung der Pixel. Diese Methode wird in hochauflösenden Netzwerken wie SRGAN verwendet. Zuerst verwenden wir die Faltung, um die C x H x W-Feature-Map in (Cr^2) x H x W umzuwandeln. Durch die Neuanordnung der Pixel werden diese Pixel dann in einem Mosaik „neu angeordnet“, um eine Ausgabe der Größe C x (Hr) x (Wr) zu erzeugen.
Nicht gefüllt oder gefüllt?
Wenn der Kernel größer als 1x1 und ohne Auffüllung ist, erzeugen Faltungsschichten eine Ausgabe, die kleiner als die Eingabe ist. Dies ist ein Problem für U-Net. Erinnern Sie sich an das U-Net-Diagramm im vorherigen Abschnitt, dass wir einen Teil des Bildes mit seinem dekodierten Teil verbunden haben. Wenn wir keine Auffüllung verwenden, hat das dekodierte Bild im Vergleich zum kodierten Bild kleinere räumliche Abmessungen.
Das ursprüngliche U-Net-Papier enthielt jedoch keine Polsterung. Obwohl kein Grund angegeben wurde, gehe ich davon aus, dass der Grund dafür darin liegt, dass die Autoren keine Segmentierungsfehler an den Bildrändern einführen wollten. Stattdessen führten sie vor der Verkettung einen Mittelzuschnitt des codierten Bildes durch. Bei einem Bild mit der Eingabegröße 572 x 572 beträgt die Ausgabe 388 x 388, mit einem Verlust von etwa 50 %. Wenn Sie U-Net ohne Padding ausführen möchten, müssen Sie es mehrmals auf überlappenden Kacheln ausführen, um ein vollständiges Segmentierungsbild zu erhalten.
So funktioniert U-Net
Hier implementieren wir ein sehr einfaches U-Net-ähnliches Netzwerk nur zur Segmentierung von Ellipsen. Dieses U-Net ist nur 3 Schichten tief, verwendet die gleiche Polsterung und den gleichen binären Kreuzentropieverlust. Komplexere Netzwerke können mehr Faltungsschichten pro Auflösung verwenden oder die Tiefe nach Bedarf erweitern.
import torch
import numpy as np
import torch.nn as nn
class EncoderBlock(nn.Module):
# Consists of Conv -> ReLU -> MaxPool
def __init__(self, in_chans, out_chans, layers=2, sampling_factor=2, padding="same"):
super().__init__()
self.encoder = nn.ModuleList()
self.encoder.append(nn.Conv2d(in_chans, out_chans, 3, 1, padding=padding))
self.encoder.append(nn.ReLU())
for _ in range(layers-1):
self.encoder.append(nn.Conv2d(out_chans, out_chans, 3, 1, padding=padding))
self.encoder.append(nn.ReLU())
self.mp = nn.MaxPool2d(sampling_factor)
def forward(self, x):
for enc in self.encoder:
x = enc(x)
mp_out = self.mp(x)
return mp_out, x
class DecoderBlock(nn.Module):
# Consists of 2x2 transposed convolution -> Conv -> relu
def __init__(self, in_chans, out_chans, layers=2, skip_connection=True, sampling_factor=2, padding="same"):
super().__init__()
skip_factor = 1 if skip_connection else 2
self.decoder = nn.ModuleList()
self.tconv = nn.ConvTranspose2d(in_chans, in_chans//2, sampling_factor, sampling_factor)
self.decoder.append(nn.Conv2d(in_chans//skip_factor, out_chans, 3, 1, padding=padding))
self.decoder.append(nn.ReLU())
for _ in range(layers-1):
self.decoder.append(nn.Conv2d(out_chans, out_chans, 3, 1, padding=padding))
self.decoder.append(nn.ReLU())
self.skip_connection = skip_connection
self.padding = padding
def forward(self, x, enc_features=None):
x = self.tconv(x)
if self.skip_connection:
if self.padding != "same":
# Crop the enc_features to the same size as input
w = x.size(-1)
c = (enc_features.size(-1) - w) // 2
enc_features = enc_features[:,:,c:c+w,c:c+w]
x = torch.cat((enc_features, x), dim=1)
for dec in self.decoder:
x = dec(x)
return x
class UNet(nn.Module):
def __init__(self, nclass=1, in_chans=1, depth=5, layers=2, sampling_factor=2, skip_connection=True, padding="same"):
super().__init__()
self.encoder = nn.ModuleList()
self.decoder = nn.ModuleList()
out_chans = 64
for _ in range(depth):
self.encoder.append(EncoderBlock(in_chans, out_chans, layers, sampling_factor, padding))
in_chans, out_chans = out_chans, out_chans*2
out_chans = in_chans // 2
for _ in range(depth-1):
self.decoder.append(DecoderBlock(in_chans, out_chans, layers, skip_connection, sampling_factor, padding))
in_chans, out_chans = out_chans, out_chans//2
# Add a 1x1 convolution to produce final classes
self.logits = nn.Conv2d(in_chans, nclass, 1, 1)
def forward(self, x):
encoded = []
for enc in self.encoder:
x, enc_output = enc(x)
encoded.append(enc_output)
x = encoded.pop()
for dec in self.decoder:
enc_output = encoded.pop()
x = dec(x, enc_output)
# Return the logits
return self.logits(x)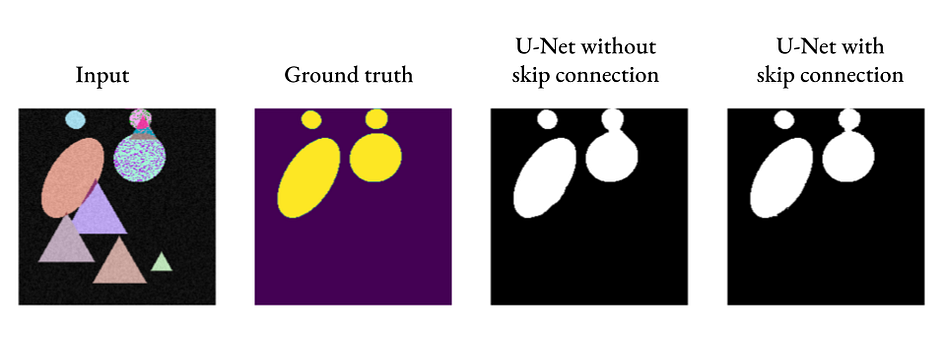
Wie wir sehen können, kann U-Net auch ohne Skip-Verbindungen akzeptable Segmentierungsergebnisse liefern, aber durch das Hinzufügen von Skip-Verbindungen können feinere Details eingeführt werden (siehe die Verbindung zwischen den beiden Ovalen rechts).
abschließend
Wenn ich U-Net in einem Satz erklären würde, wäre es so, dass U-Net wie ein Encoder-Decoder für Bilder ist, aber mit Skip-Verbindungen, um sicherzustellen, dass keine Details verloren gehen. U-Net wird häufig bei vielen Segmentierungsaufgaben eingesetzt und hat in den letzten Jahren auch bei Bildgenerierungsaufgaben Erfolge erzielt.
Verweise:
[1] Long, Jonathan, Evan Shelhamer und Trevor Darrell. „Vollständig Faltungsnetzwerke zur semantischen Segmentierung.“ Tagungsband der IEEE-Konferenz zu Computer Vision und Mustererkennung . 2015.
[2] Ronneberger, Olaf, Philipp Fischer und Thomas Brox. „U-net: Faltungsnetzwerke für die biomedizinische Bildsegmentierung.“ Internationale Konferenz über medizinische Bildverarbeitung und computergestützte Intervention . Springer, Cham, 2015.
[3] Milletari, Fausto, Nassir Navab und Seyed-Ahmad Ahmadi. „V-net: Vollständig faltende neuronale Netze für die volumetrische Segmentierung medizinischer Bilder.“ 2016 vierte internationale Konferenz zum Thema 3D-Vision (3DV) . IEEE, 2016.
☆ ENDE ☆
Wenn Sie dies sehen, bedeutet das, dass Ihnen dieser Artikel gefällt. Bitte leiten Sie ihn weiter und liken Sie ihn. Suchen Sie auf WeChat nach „uncle_pn“. Willkommen beim Hinzufügen des WeChat „woshicver“ des Herausgebers. Jeden Tag wird im Freundeskreis ein hochwertiger Blogbeitrag aktualisiert.
↓ Scannen Sie den QR-Code, um den Editor hinzuzufügen↓
