Die in dieser Ausgabe geteilte Literatur wird vom ARC Centre of Excellence in Plant Energy Biology, School of Molecular Sciences, University of Western Australia, in der berühmten Botanik-Rezensionszeitschrift „Current Opinion in Plant Biology“ mit dem Titel „ Triticeae- Genomsequenzen offenbaren enorme Erweiterungen“ veröffentlicht von Genfamilien, die an der Wiederherstellung der Fruchtbarkeit beteiligt sind“. Die Studie ergab, dass die RFL- und mTERF-Genfamilien im Zusammenhang mit der Wiederherstellung der Fruchtbarkeit eine große Expansion und Diversität aufwiesen, und enthüllte den Evolutionsmechanismus dieser Gene, was eine neue Perspektive für die Hybridproduktion von Weizen und verwandten Sorten eröffnete.

-
Veröffentlichte Zeitschrift: Current Opinion in Plant Biology
-
Erscheinungszeit: 2022
-
Schlagfaktor: 9,396
-
DOI: 10.1016/j.pbi.2021.102166
Artikel-Highlights
● Dieses Papier fasst aktuelle Forschungsergebnisse zur Entwicklung und Diversität der RFL- und mTERF-Genfamilien in Getreide zusammen.
● Diskussion des Forschungsfortschritts und zukünftiger Forschungstrends auf der molekularen Basis von Pflanzen-CMS und der Wiederherstellung der Pflanzenfruchtbarkeit.
Hauptinhalt
1. Genomweite Vergleichsstudie der RFL-Familie in Getreide
Die erste genomweite Vergleichsstudie von Reis- und Gersten-RFL-Familien (Restoration of Fertility) zeichnet sich durch ein hohes Maß an inter- und intraspezifischen Struktur- und Kopienzahlvariationen aus. Eine detaillierte Analyse der Kollinearität und Sequenzerhaltung ergab, dass RFL-Sequenzen, die sich innerhalb eines Clusters befinden, mit größerer Wahrscheinlichkeit diversifizieren als solche, die sich außerhalb des Clusters befinden. Die Diversität innerhalb der RFL-Gruppe wird durch genetische Interaktionen zwischen dem sterilitätsverursachenden CMS-Gen und dem Rf-Gen bestimmt.
Das Weizengenom enthält eine ungewöhnlich hohe Anzahl an RFL-Genen (207 in „Chinese Spring“), viel höher als bei anderen Angiospermen (ungefähr 20–30). Die Forscher entwickelten eine Methode zur RFL-Sequenzerfassung, die auf einem ähnlichen Ansatz für das R-Gen basiert. Unter Verwendung der RFL-Sequenzen von Brotweizen und seinen subgenomischen Spendern sowie eng verwandten Arten wurden RFL-spezifische Sonden für die Sequenzerfassung entwickelt. Kandidatengene für die Restorer-Gene Rf1 und Rf3 wurden durch Vergleich der in den Restorer- und Nicht-Restaurer-Genotypen vorhandenen Sequenzen ausgewählt und ihre Funktionen durch transgene Experimente verifiziert.
Die Verfügbarkeit von Roggen- und Weizen-Referenzgenomsequenzen erleichterte die Charakterisierung von Rfmulti multiloci ( Wiederherstellung der Fruchtbarkeit in mehreren CMS-Systemen ). Ein Kandidatengen für Rfmulti wurde im Weizengenom durch vergleichende Analyse kollinearer Regionen im Roggen- und Weizengenom identifiziert . Der nächste Schritt wird darin bestehen, diese Vorhersage experimentell zu überprüfen.
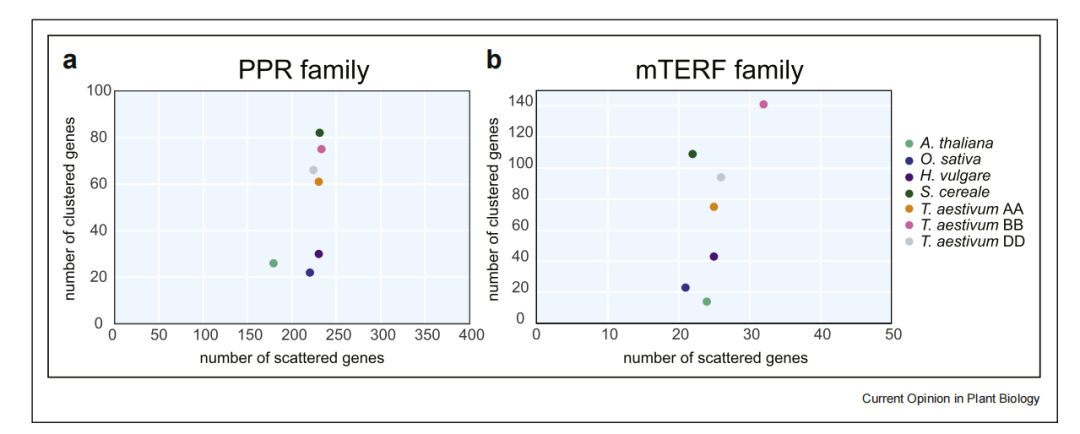
Abbildung 1 Erweiterung der RFL- und mTERF-Genfamilien bei Triticeae und ihren Verwandten
2. Identifizierung des mTERF-Gens vom RFL-Typ im Weizen-Pan-Genom
Studien an Roggen und Gerste legen nahe, dass auch Mitglieder der mTERF-Familie (mitochondrialer Transkriptionsterminationsfaktor) an der Wiederherstellung der Fruchtbarkeit in Getreide beteiligt sein könnten. Proteine, die zu dieser Familie gehören, bestehen aus einem wiederholten Motiv aus ca. 30–35 Aminosäuren (mTERF-Motiv), ähnlich wie PPR-Proteine. Bei Getreide besteht das gleiche Phänomen zwischen verteilten und aggregierten mTERF-Genen. Es gibt ungefähr 21–32 verstreute Gene, die den 24 verstreuten Genen in Arabidopsis entsprechen, von denen viele hoch konserviert und wahrscheinlich homolog sind. Geclusterte mTERF-Gene erfuhren in Getreide eine erhebliche Ausweitung, mit über 300 in Weizen und etwa 100 in Roggen (Abb. 1). Die geclusterten mTERFs zeigten ein ähnliches Evolutionsmuster wie die RFL-PPR-Genkladen, das heißt, die Ähnlichkeit zwischen den Kollaterallinien war hoch, die Ähnlichkeit zwischen den Ortholinien jedoch relativ gering. Bei Roggen überlappten die RFL- und mTERF-Cluster auf Chromosom 4R mit genomischen Regionen, die Rfp1- , Rfp2- und Rfp3- Restaurierungsloci enthielten. Bisher wurde nur die Sequenz von Rfp1 geklont und als mTERF-Gen identifiziert.
Fortschritte bei der Sequenzierung und den Assemblierungsstrategien, die zur Auflösung wiederholter Getreidegenome entwickelt wurden, haben die Genauigkeit der komplexen Assemblierung von RFL- und mTERF-Genclustern verbessert. Dies kann durch die Anzahl der in den 4 Assemblies erhaltenen RFL-Gene veranschaulicht werden (Abb. 2a). Verfeinerte Referenzgenomassemblys werden zusammen mit Zehntausenden lang gelesenen Pan-Genom- und Pan-Transkriptom-Datensätzen einen großen Beitrag zur Entschlüsselung der Diversität und Ausbreitung von RFL- und mTERF-Genen in Getreide leisten.
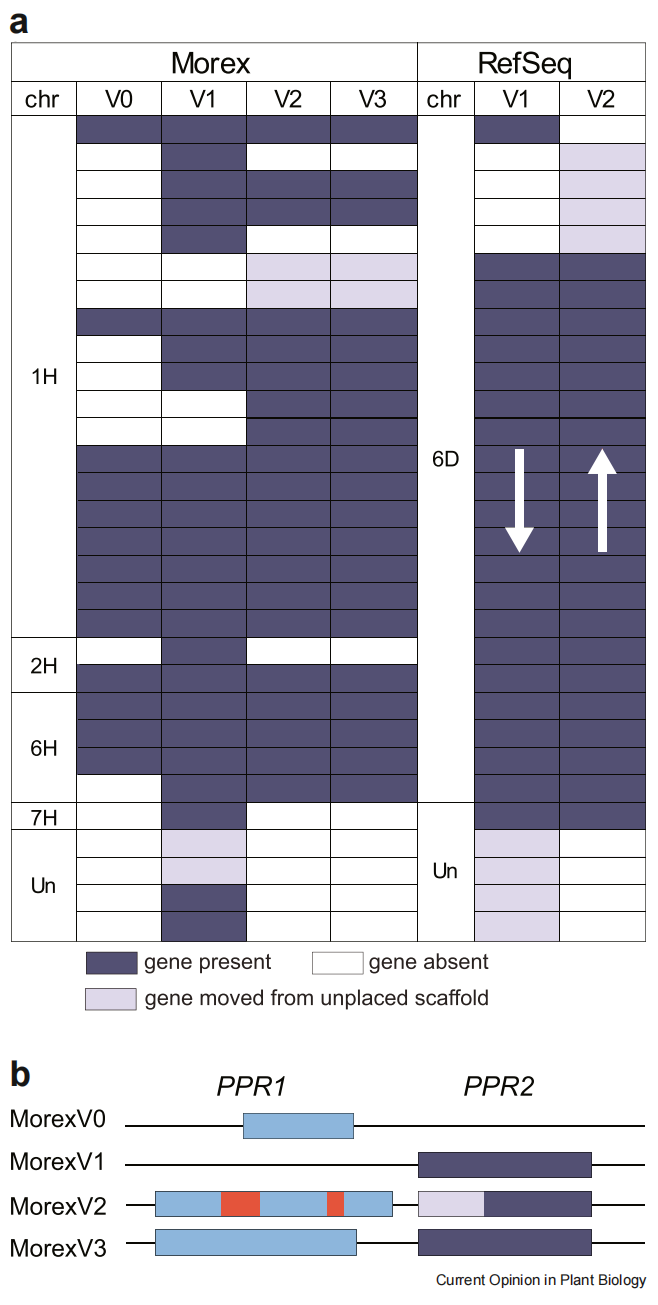
Abbildung 2 Vergleich der RFL-Genassemblierungsergebnisse verschiedener Gerste
3. Evolution der mitochondrialen Sequenz und ihr Einfluss auf die Aufrechterhaltung von CMS-Merkmalen
Umlagerungen des mitochondrialen Genoms kommen in Pflanzen häufig vor und können neue offene Leserahmen (ORFs) erzeugen, von denen einige männliche Sterilität verursachen. Alle berichteten CMS-Gene weisen eine chimäre Struktur auf, und männlich sterile Pflanzen haben bei der Samenproduktion einen Vorteil gegenüber Hermaphroditen, da sie die Energie von der Pollenentwicklung auf die Samen umverteilen. Dies erleichtert die Übertragung des mütterlich vererbten mitochondrialen CMS-Genoms. Die daraus resultierende Anhäufung männlich steriler Pflanzen erzeugt jedoch wiederum einen starken Selektionsdruck zugunsten von Rf- Genen, die CMS unterdrücken, sodass Pflanzen, die sie tragen, sterile Pflanzen bestäuben können, die CMS nicht unterdrücken. Kürzlich wurde gezeigt, dass eine einzelne Nukleotidsubstitution in der orf138- Gensequenz männliche Sterilität im Ogu-CMS-System von Rettich induziert. Es wurde festgestellt, dass diese Mutation innerhalb der Bindungsstelle des Recoverin-Rfo liegt und die Affinität von Rfo für diese Stelle nachweislich deutlich verringert (Abb. 3a). Interessanterweise identifizierte dieselbe Studie ein neues Restorer-Gen, Rfs , das in der Lage ist, die durch mutierte orf138 verursachte Sterilität zu unterdrücken.
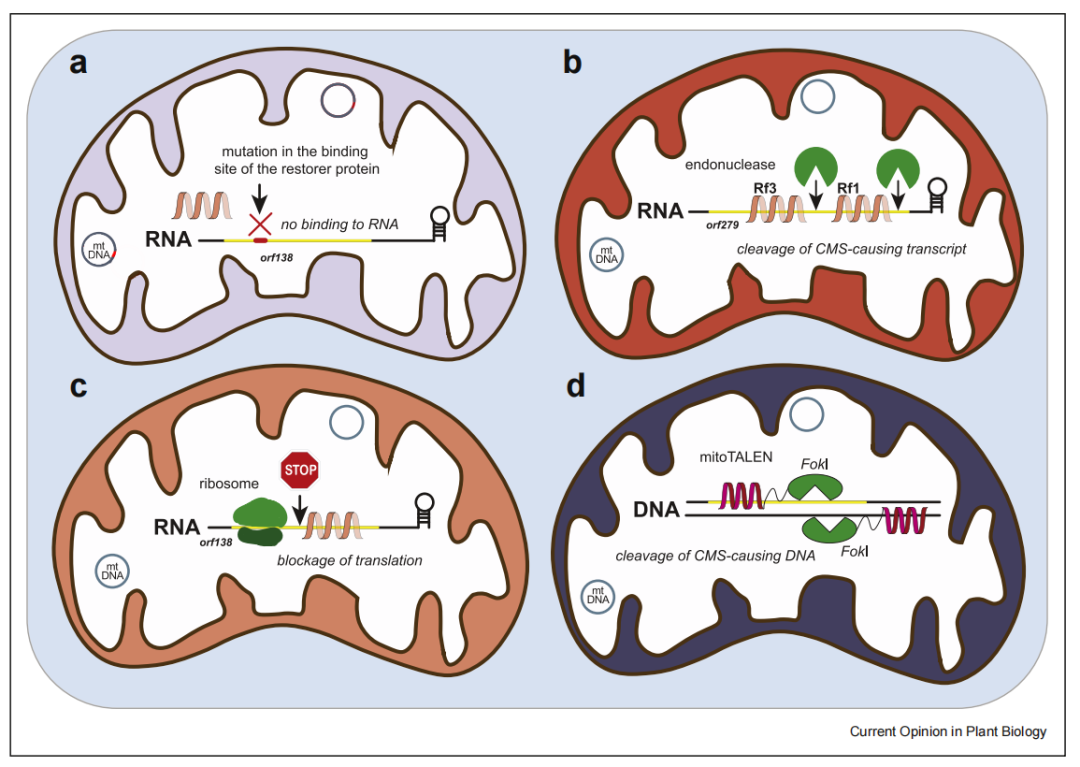
Abbildung 3 Interaktion zwischen Mitochondrien und Kerngenom und Wiederherstellung der Pflanzenfruchtbarkeit bei CMS
4. Molekulare Wirkungsweise des Rf-Proteins
Es wurden zwei Wirkungsweisen des Rf- Gens zur Blockierung der CMS-Genexpression beschrieben, nämlich der „RNA-Spaltung“- oder „Translationsblock“-Weg, und ob die beiden Wege grundlegend unterschiedlich sind, muss noch getestet werden; die molekulare Wirkungsweise des mTERF-Rf-Protein Es muss noch geklärt werden, aber es ist möglich, dass sie die CMS-induzierte Expression mitochondrialer ORfs auf ähnliche Weise wie PPR-Rf-Proteine blockieren.
In den letzten Jahren wurden Methoden zur Bearbeitung der mitochondrialen DNA entwickelt, um CMS-Merkmale in Pflanzen zu eliminieren. Es ist unwahrscheinlich, dass diese Methoden in hybriden Produktionssystemen verwendet werden, sie sind jedoch als Forschungsinstrumente zur Untersuchung und Validierung von CMS-Genen nützlich und könnten in Zukunft zur Schaffung neuer CMS-induzierender Loci verwendet werden.
In der modernen Züchtung ist die Kenntnis nicht nur der genetischen Wechselwirkungen der CMS- und Rf- Loci, sondern auch anderer Loci, die die Pflanzenfruchtbarkeit, Selbstbestäubung gegenüber Fremdbestäubung und Hybridleistung beeinflussen, für die Entwicklung unbegrenzter Kreuzungsprogramme von großer Bedeutung.
Zusammenfassen
Insgesamt zeigten vergleichende Studien von Referenzgenomsequenzen und Pangenomsequenzen eine enorme Expansion und extreme Diversität in den RFL- und mTERF-Genfamilien, zwei Hauptquellen für fruchtbarkeitswiederherstellende Gene in Getreide. In Zukunft werden genomweite Assoziationsstudien in Kombination mit Long-Read-Sequenzierungstechnologien das Klonen und Charakterisieren neuer Restorer-Gene für den Einsatz in Kreuzungsprogrammen beschleunigen. Diese Ergebnisse werden auch neue Einblicke in den Mechanismus bringen, durch den nukleare Rf-Gene das CMS-Merkmal unterdrücken.
Verweise:
Genomsequenzen von Triticeae offenbaren enorme Erweiterungen von Genfamilien, die an der Wiederherstellung der Fruchtbarkeit beteiligt sind. Aktuelle Meinung in der Pflanzenbiologie, 2022.
DOI: 10.1016/j.pbi.2021.102166