1. Introduction à l'explosion locale
Local Blast (Basic Local Alignment Search Tool) est un outil de recherche de comparaison local qui peut effectuer une recherche d'explosion dans une base de données créée par vous-même. Par rapport à l'explosion en ligne de NCBI, il offre une vitesse plus rapide, une plage de recherche plus petite et aucune connexion Internet. Par exemple, si vous connaissez déjà un gène de l'orge et que vous avez clarifié sa fonction, vous devez maintenant trouver un gène à haute similitude de séquence dans l'orge, vous pouvez créer une base de données localement, c'est-à-dire créer une base de données d'orge, puis faire exploser pour trouver la même séquence source.
2. Installation de souffle local
1. NCBI télécharger la version locale de blast
[Lien de téléchargement]: https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ pour télécharger la version adaptée à mon système informatique, ici je télécharge ncbi-blast-2.11.0

2. Installez le programme de dynamitage local
Après le téléchargement, double-cliquez pour installer, vous pouvez juger si le téléchargement est terminé en comparant la taille du programme, générer deux sous-répertoires bin et doc, où bin est le répertoire du programme et doc est le répertoire du document, vous pouvez également cliquer pour vérifier l'intégrité du programme, puis créer un nouveau dossier de répertoire, renommé endb
3. Paramètres des variables d'environnement utilisateur
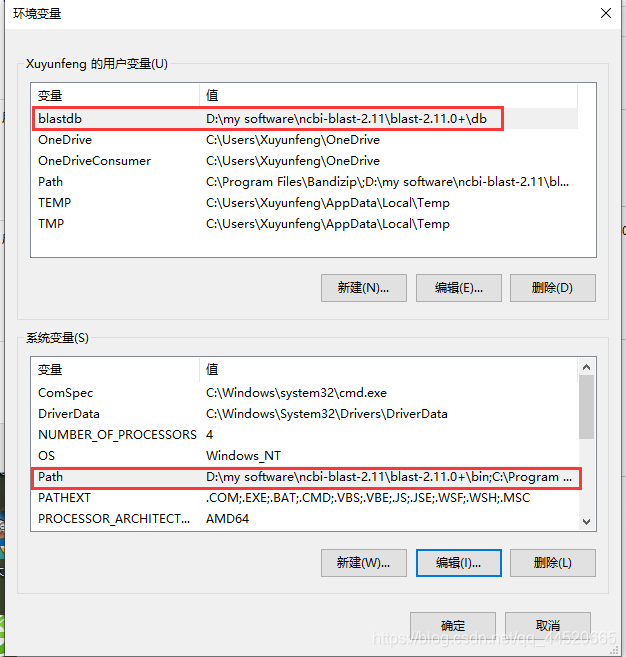
Cet ordinateur-Propriétés-Paramètres système avancés-Variables d'environnement
Sous Variables utilisateur: Nom de la nouvelle variable: balstdb, la valeur de la variable est le chemin du dossier db nouvellement créé après l'installation de l'ordinateur.
Sous Variables système: Chemin-Ajouter une valeur de variable à emplacement du dossier bin
3. Créez une base de données locale sur les explosions
1. Téléchargement de la base de données
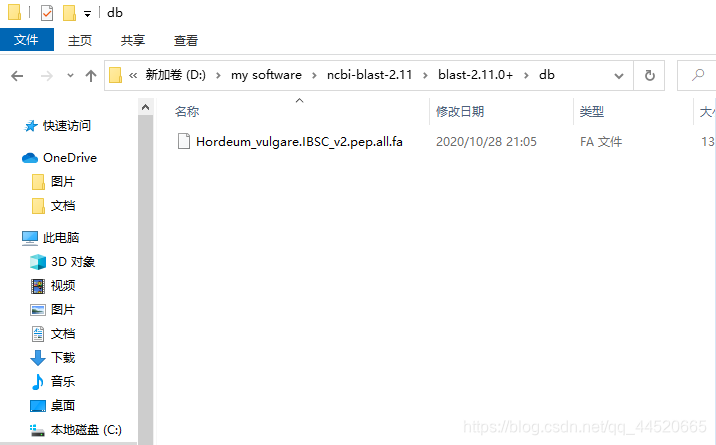
Téléchargez les données de pep de l'orge à partir de bases de données telles que l'ensemble et décompressez-les dansdbDossier
2. Formatage de la base de données
Lorsqu'aucune cible n'est sélectionnée dans le dossier db, maintenez la touche Maj enfoncée, cliquez avec le bouton droit sur l'espace vide du dossier et sélectionnez Ouvrir iciPowerShellFenêtre, ouvrez la fenêtre comme indiqué sur la figure. J'utilise un système win10 pour entrer rapidement l'emplacement du dossier db de cette manière. S'il s'agit d'une autre version du système Windows, vous devrez peut-être exécuter cmd pour entrer l'emplacement du dossier db.
attention: Le fichier se trouve dans le dossier actuellement ouvert par Windows PowerShell, il n'est donc pas nécessaire de remplacer le chemin.
Exécutez la commande:
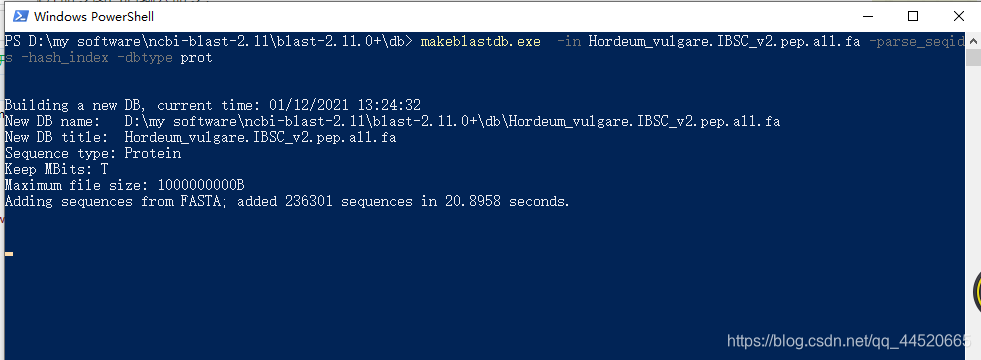
makeblastdb.exe -in Hordeum_vulgare.IBSC_v2.pep.all.fa -parse_seqids -hash_index -dbtype prot
Parmi eux, Hordeum_vulgare.IBSC_v2.pep.all.fa est le nom de la base de données que vous souhaitez formater. Apportez des modifications en fonction du nom de votre propre base de données. N'oubliez pas d'ajouter le suffixe .fa; le prot après dbtype indique le type de base de donnéesprotReprésente la base de données de séquences d'acides aminés, s'il s'agit d'une séquence nucléotidique, utiliseznucl. Une fois la base de données formatée dans la ligne de commande, la figure suivante s'affiche
4. Opération de dynamitage locale
1. Préparation de la séquence de requêtes
Créé sous le dossier D: \ mon logiciel \ ncbi-blast-2.11 \ blast-2.11.0 + \ dbtarget.seq.txtEnregistrez la séquence à interroger dans in.txt au format fasta. Nous avons pris une séquence de protéines d'orge comme exemple

2. requête de séquence de requête
Créé sous le dossier D: \ my software \ ncbi-blast-2.11 \ blast-2.11.0 + \ dbNouveau fichier texte.txtLe fichier texte, utilisez la commande blast:
blastp.exe -task blastp -query target.seq.txt -db Hordeum_vulgare.IBSC_v2.pep.all.fa -out out.txt -evalue 1e-10 -outfmt 6 -num_threads 2
Description des paramètres associés:
Le programme blastp.exe exécute la commande, le programme avant exe est modifié selon vos besoins, y compris les programmes contenus dans le dossier bin tels que blastn, blatp, tblastx;
-Après la tâche , sélectionnez le programme que vous souhaitez utiliser, blastn, blastp, tblastx, etc.
-query est suivi du nom de fichier de la séquence de requête;
-db suivi du nom de la base de données formatée;
-out est suivi du nom et du format du fichier à afficher. Le format comprend 0-10, dont 6 et 0 sont les plus couramment utilisés, vous pouvez donc l'essayer vous-même.
-num_threads paramètres peuvent être définis en fonction des performances de leurs ordinateurs, l'ordinateur portable recommandé pas plus de 2, améliorant ainsi le rapport d'efficacité pour

enregistrer après leNouveau fichier texte.txtRenommé enHordeum_vulgare.pep-blast.cmd, À ce stade, un fichier texte a été modifié pour la commande cmd dans Windows, double-cliquez pour exécuter
description du résultat de l'explosion
 Chaque colonne indique:
Chaque colonne indique:
A: Query_id
B: Subject_id
C: Identity
D: Align_length
E: Miss_match
F: Gap
G: Query_start
H: Query_end
I: Subject_start
J: Subject_end
K: E_value
L: Score
E value (Expect) : indique une correspondance aléatoire Pour exemple, E = 1, ce qui signifie que dans une base de données de la taille actuelle, le nombre moyen d'objets trouvés par hasard est de 1. Plus la valeur E est élevée, plus la probabilité de correspondance aléatoire est grande. Lorsque la valeur E est proche de zéro ou de zéro, il s'agit essentiellement d'une correspondance parfaite. De manière générale, nous pensons qu'une valeur E inférieure à 10-5 est un résultat de valeur S plus réalisable. On peut imaginer que dans la même base de données, s'il y a 1000 entrées avec une chance de valeur S supérieure à la valeur actuelle lorsque E = 0,001, alors si E n'est pas mis à 10-6, un seul résultat peut être obtenu, c'est-à-dire , la valeur S est la plus fiable. Mais la valeur E n'est pas une panacée. Il a des limites dans les situations suivantes:
1) Lorsque la séquence cible est trop petite, la valeur E sera trop grande, car une valeur S plus élevée ne peut pas être obtenue.
2) Lorsque l'homologie entre les deux séquences est élevée, mais qu'il y a un grand écart (écart), la valeur S diminuera. À l'heure actuelle, les scores d'écart sont très utiles.
3) Lorsque les régions non fonctionnelles de certaines séquences ont un faible caractère aléatoire, cela peut entraîner une homologie plus élevée entre les deux séquences.
Résumé de la valeur E: la valeur E convient aux séquences qui ont une certaine longueur et ne peuvent pas être trop peu complexes. Lorsque la valeur E est inférieure à 10-5, cela indique que les deux séquences ont une homologie plus élevée, non à cause d'erreurs de calcul. Lorsque la valeur E est inférieure à 10-6, l'homologie entre les deux séquences du tableau est très élevée et il n'y a presque pas besoin de confirmation supplémentaire.
Identité (Identités) : ou similitude. Le nombre de bases appariées est le pourcentage de la longueur totale de la séquence.
Score : plus le score est élevé, meilleure est l'homologie; plus la valeur de l'attente est petite, meilleur est le résultat de la comparaison, indiquant que l'erreur causée par certaines raisons est plus petite; les identités sont l'homologie (similitude), comme le montre l'exemple Seulement 35 sur les 1 299 bases comparées sont incomparables et les 97% restants sont identiques;
Gaps : fait référence au nombre de bases qui sont plus ou moins ou manquantes; suppressions ou insertions (Gaps): insertions ou suppressions. Utilisez "-" pour indiquer.
De plus, le Strand comparé est jugé par s. Début: et s. Fin, comme dans la troisième ligne du résultat ci-dessus. La valeur Star est supérieure à s. Fin, cela signifie une chaîne négative.
Résumé et complément: une autre méthode
Pour la comparaison des séquences d'explosion locales, nous devons créer un fichier de bibliothèque (la bibliothèque d'explosions en ligne utilise des fichiers provenant des principales bases de données biologiques), et entrez la commande:makeblastdbCommande de création de base de données.
makeblastdb -in b.fasta -dbtype nucl -out b.fasta.blastdb
makeblastdb -in b.fasta -dbtype nucl -out b.fasta.blastdb [Le fichier est dans le dossier actuellement ouvert par Windows PowerShell, il n'est donc pas nécessaire de saisir le chemin]
== Notez que l'espace ne doit pas être inférieur =
-in est le fichier de la bibliothèque de construction, nous utilisons la bibliothèque de construction de fichier plus grande
-out est suivi du nom du fichier de la bibliothèque de sortie. Généralement, après la première étape de construction de la bibliothèque, trois fichiers nhr / nin / nsq seront générés ensemble en tant que bibliothèque pour passer à l'étape suivante.
Si vous exécutez une séquence d'acide nucléique, utilisez blastn
blastn -query a.fasta -db b.fasta.blastdb -out b.blast -outfmt 6 -evalue 1e-5 -num_threads 2
BLASTN -query a.fasta -db b.fasta.blastdb -out b.blast -outfmt 6 -evalue 1E-5 -num_threads 2
-query : Chemin du fichier d'entrée et nom de fichier
-out : chemin du fichier de sortie et le nom de fichier
-db : Chemin de la base de données
formaté et nom de la base de données -outfmt : format du fichier de sortie, il y a un total de 12 formats, 6 est le format tabulaire correspondant au format BLAST
m8 -evalue : définit la valeur e-value du résultat de sortie
-num_descriptions : le tabulaire format de sortie Nombre de
-num_threads : nombre de threads [le notebook ne dépasse généralement pas 2]
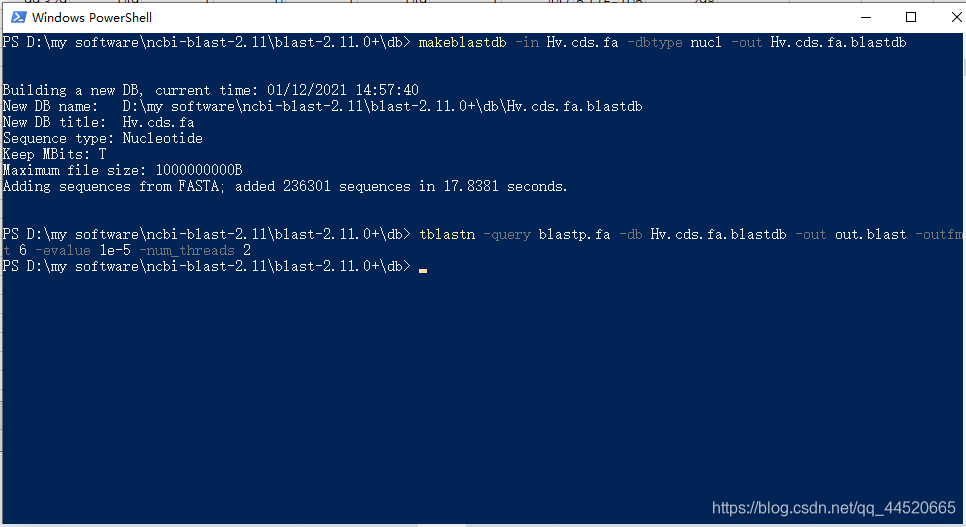
Cette image montre la commande que j'ai utilisée pour exécuter tblastn avec la séquence protéique dans la base de données d'acides nucléiques.
Enfin, attachez le
lien vers l'article de référence :
https://blog.csdn.net/zxpuls123/article/details/81407277
https: / / blog .csdn.net / qq_43337286 / article / details / 103120003