Anuncio de beneficio : para responder a las necesidades de aprendizaje de los estudiantes, después de la discusión y preparación por parte del equipo de capacitación de Yishengxin, ahora se decidió organizar clases en vivo en línea y conferencias fuera de línea sobre análisis de amplicón 16S, metagenómica , cursos de Python y transcriptómica . Los profesores que se inscriban en clases en vivo en línea pueden optar por participar en una clase fuera de línea del mismo curso dentro de un año . Espero conocerte tanto en línea como fuera de línea.
Información disponible actualmente:
Hora de inicio del curso de genómica clínica en línea/fuera de línea: 2023/6/30-7/2
Hora de inicio del curso en línea/fuera de transcriptómica: 2023/9/15-17
Hora de inicio de la clase en línea/fuera de línea de Amplicon: 2023/10/13-15
Hora de inicio del curso Metagenome en línea/fuera de línea: 2023/11/24-27
Enlace de registro: http://www.ehbio.com/Training/
Con la expectativa de los fanáticos, "Shengxin Baodian" y "Metagenome" lanzaron conjuntamente una capacitación especial sobre " Análisis de metagenoma " en Beijing ( las clases en línea y fuera de línea están disponibles al mismo tiempo ), brindando a todos un atajo para entrar por la puerta de Shengxin Los compañeros brindan la oportunidad de aprender y comunicarse sobre el análisis metagenómico, ayudar a los estudiantes a comprender realmente los principios del análisis y completar el análisis real, y crear una enseñanza original de cuatro etapas (3 días de enseñanza intensiva + 2 semanas de autopráctica + reenfocado). conferencias y preguntas y respuestas + revisión de video de clase y práctica repetida), los cuatro enlaces de "enseñanza-práctica-respuesta-uso" están unificados y coordinados para realizar verdaderamente un análisis independiente de big data .
Con respecto a la importancia de aprender el análisis bioinformático, lea "Curso intensivo de 9 días en bioinformática: convertirse en una persona indispensable en el equipo" . El análisis de bioinformática es inseparable de la escritura de programas. Esta parte no es tan difícil como se imagina. Mientras siga nuestra operación, podrá entenderla. Para obtener más información, consulte " Experiencia de aprendizaje de programas en bioinformática" .
curso introductorio
Lea la introducción del curso en detalle. Si domina todo el contenido siguiente, no es necesario que asista a esta capacitación.
El metagenoma/microbioma es uno de los campos de investigación más populares del mundo en la actualidad. Con el fin de fortalecer los intercambios técnicos y la difusión en este campo, y promover el desarrollo del programa de microbioma de China, jóvenes investigadores de la Academia de Ciencias de China crearon el "Metagenoma". cuenta pública con el objetivo de crear esta plataforma de intercambio de pensamiento y tecnología de productos secos puros en el campo. Se estableció hace tres años y ha compartido más de 3100 artículos originales de tecnología profesional, con más de 150 000 seguidores y más de 40 millones de lecturas acumuladas.
Con el fin de satisfacer las necesidades de los lectores para un mayor estudio, ahora estamos organizando un curso de capacitación especial sobre metagenómica en conjunto con "Shengxin Baodian" para aprender más y comunicar técnicas de análisis de metagenómica. Lo guiaremos para que comience rápidamente y ahorre un tiempo precioso. , y ayudar a que los resultados de la investigación científica se produzcan lo antes posible.
Este curso tiene un total de días 3, clases 6 por día y un total de clases 18. Todos los cursos se combinan con teoría y práctica (siempre que las conferencias sean análisis que pueda aprender y realizar usted mismo). Desde la base Linux y R, la construcción de la plataforma de análisis del servidor Linux de metagenómica, el software de análisis estadístico común de Windows, la interpretación de tablas de análisis de datos y el combate real, la metagenómica con referencia (referencia/basada en lectura adecuada para intestinos humanos, animales, etc.) y sin referencia ( De Novo/Assemble es adecuado para plantas, muestras ambientales, etc.) proceso de análisis estándar, Binning (extracción del genoma de una sola bacteria), análisis estadístico y varios análisis avanzados (árbol evolutivo de conexiones multigénicas, dibujo y embellecimiento de diagramas de red, atributo de red comparación, aprendizaje automático, etc.) y diseño de modificación de imagen a nivel del SNC. En 3 días, el viejo conductor lo llevará por el camino áspero que toma 3 meses o incluso 3 años de autoaprendizaje, ayudándolo a realizar verdaderamente el análisis metagenómico y optimizar el plan de análisis de acuerdo con los antecedentes de su propio sujeto.
Esquema del curso
Cada clase tiene un tema de 1 hora, combinando la teoría con la práctica, aprendiendo los principios y practicando en la práctica, todos los cuales son el intercambio desinteresado de años de experiencia y código por parte de conductores veteranos. El siguiente es el programa del curso. Por ejemplo, 11 representa la primera clase del primer día, 26 representa la sexta clase del segundo día y 41 representa el video de preguntas y respuestas centralizado en línea dos semanas después.
| número de serie | tema | Introducción |
|---|---|---|
| 11 | Conceptos básicos de Linux | Introducción , inicio de sesión remoto, transferencia de archivos, comandos comunes |
| 12 | Instalación de software Linux | Instalación y configuración de Conda, instalación de software relacionado con metagenómica y descarga de base de datos |
| 13 | Ganar instalación de software | git , R, Rstudio, paquetes R, STAMP, AI, etc. |
| 14 | interpretación de gráficos | El significado y los escenarios de uso de los gráficos de análisis de uso común en los artículos. |
| 15 | fundamentos de r | Historia del desarrollo, aplicación en biología, dibujo ggplot2. |
| dieciséis | visualización | Clasificación de datos y dibujo en línea de 16 gráficos |
| 21 | Introducción a la metagenómica | Historial de desarrollo, ámbito de aplicación de las tecnologías de uso común e ideas de análisis |
| 22 | Control de calidad del metagenoma | FastQC , Trimmomatic, MultiQC]( ), control de calidad KneadData, computación paralela paralela |
| 23 | Especies y composición funcional | Composición de especies de MetaPhlAn2, composición funcional de HUMAnN2, especies impulsoras de asociación funcional |
| 24 | Comparación y visualización de especies y diferencias funcionales | Estadísticas del lenguaje GraPhlAn , LEfSe , STAMP , R |
| 25 | Prepárate para la publicación | Composición tipográfica de imágenes , publicación de datos , acabado de código (opcional) |
| 26 | dibujo de red | Fundación , igraph , Gephi |
| 31 | Anotación y visualización de especies | Kraken, Kraken2, GraPhlAn, Krona , microbiomeViz , metacodificador |
| 32 | Ensamblaje, anotación de genes y cuantificación. | MEGAHIT, metaSPAdes, QUAST, Prokka, cd-hit, Salmón |
| 33 | Anotación de funciones genéticas | KEEG、COG/ EggNOG、CAZy/ dbcan2、ARDB/Resfams/CARD、Uniref、VFDB、TCDB |
| 34 | Agrupación | Teoría , MetaWRAP , VizBin |
| 35 | Evolución del genoma bacteriano | Los contenedores extraen genes conservados, árbol evolutivo de múltiples genes, entienden el árbol evolutivo en un artículo Evolview básico avanzado iTOL embellecimiento avanzado , |
| 36 | Charla resumida | Revisión y resumen de rutinas de análisis metagenómico |
| 37 | examen 50 preguntas | Autoevaluación de los efectos del aprendizaje, revisión de los puntos de conocimiento |
| 41 | Preguntas y respuestas: en línea | Preguntas y respuestas, conferencias sobre el contenido del examen. |
La breve introducción del contenido del curso es la siguiente:
1. Construcción de la plataforma de análisis
“ Si un trabajador quiere hacer un buen trabajo , primero debe afilar sus herramientas.” Sin una plataforma de análisis propia, ¿cómo puede ser posible analizar big data? La cantidad de datos metagenómicos es enorme, y todavía es difícil procesar los grandes datos originales fuera de la máquina en sus propios libros. Afortunadamente, en esta etapa, las universidades generales, los institutos de investigación científica y los grupos de investigación tienen sus propios servidores. Aunque no tengan servidores, también pueden alquilar servicios domésticos como Alibaba Cloud y Tencent Cloud. Ahora que las condiciones de análisis están disponibles, cómo convertir el servidor en una poderosa herramienta para el análisis metagenómico es un problema profesional muy complicado, ¡y puede aprenderlo de inmediato aquí!
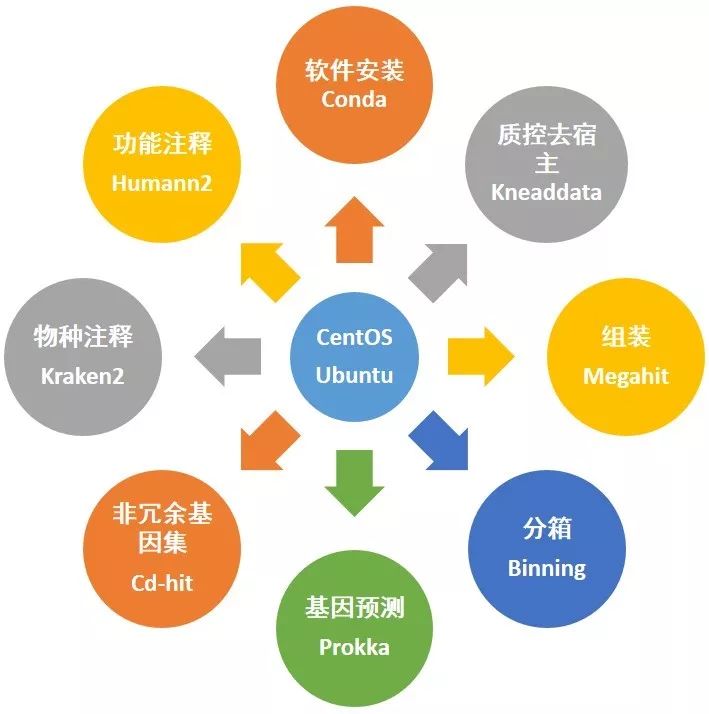
Figura 1. Construcción del proceso de análisis metagenómico - sistema, método de instalación y software principal
El servidor recomienda usar el sistema Ubuntu. La configuración mínima es de 32G de memoria y 8 núcleos; se recomiendan 256G de memoria y 24 subprocesos; cuanto mayor sea la configuración, más rápido y fluido será el análisis.
Una computadora sin software es solo un montón de chatarra, y un servidor sin un sistema de análisis metagenómico no tiene nada que ver con su análisis de datos. Si desea construir un conjunto completo de procesos de análisis metagenómico, los recursos en Internet son dispersos y escasos. El equipo de Yishengxin compartirá años de experiencia en la exploración de excelentes habilidades de diseño y software, y compartirá todos los códigos fuente , lo que le permitirá implementar rápidamente varios componentes de los que depende el proceso de análisis metagenómico en los principales sistemas de servidor Linux (Ubuntu 16/18.04, CentOS7 y otros). distribuciones principales) Diez software de uso común, cientos de paquetes dependientes de R y Python, fácil de poseer una plataforma de análisis profesional.
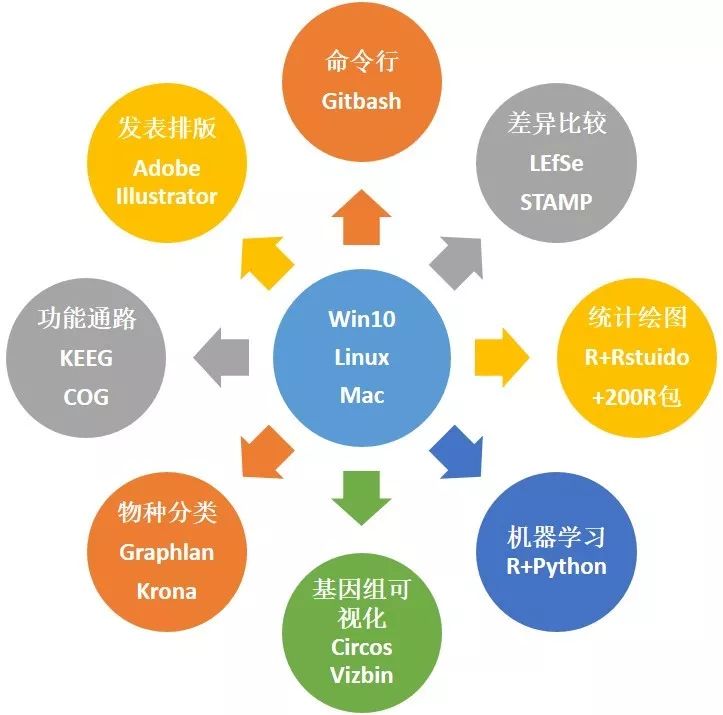
Figura 2. Yishengxin fue pionera en el análisis estadístico de datos y el proceso de visualización basado en la optimización de Win10, y la computadora portátil se convierte en una gran plataforma de análisis de datos en segundos
Se recomienda utilizar el sistema Windows 10, el análisis de memoria 8G es más rápido y fluido.
Los llamados grandes datos de secuenciación de alto rendimiento se refieren al volumen de datos sin procesar y al proceso de análisis, pero el resultado no es grande. Por lo general, el análisis metagenómico obtendrá muestras de composición de especies y tablas de composición funcional. Estas tablas son el punto de partida para el análisis posterior, el análisis avanzado y el análisis de personalidad. La mayor parte del trabajo se puede hacer en nuestros cuadernos, pero muchas personas no saben cómo hacerlo. para empezar. .
De hecho, su computadora personal es una poderosa herramienta para el análisis estadístico de la tabla de datos (matriz de abundancia). El equipo de Yishengxin ha creado un proceso de análisis multiplataforma , que puede realizar fácilmente la mayor parte del análisis de amplicones, estadísticas de macrogenoma y visualización en los portátiles de Windows de todos. plataforma para tablas de datos, optimice y pruebe en base al Win10 más convencional en la actualidad, y convierta su computadora portátil en una plataforma de análisis y visualización de datos en segundos .
También lo llevaremos a configurar toda la plataforma de análisis y visualización en Linux (Mac es similar a Linux, no se hace distinción, pero algunos software pueden instalarse de diferentes maneras, no se han realizado pruebas en profundidad y no es recomendado usarlo para entrenar).
2. La base de la carta de vida.
Con la plataforma de análisis de bioinformación, aún es necesario aprender algo único sobre cómo usarla de manera flexible. Lo más importante en el siglo XXI son los talentos, lo mejor para los talentos es dominar tres idiomas, lo que te hará invencible en la vida y es un talento indispensable en cualquier equipo. Estos tres idiomas son el chino, el inglés y el lenguaje informático . El chino se usa en la escuela todos los días, y el inglés ha estado en contacto con los médicos durante al menos 10 años y se puede usar para leer y escribir literatura.En cuanto a los lenguajes de programación, todos han aprendido el lenguaje Visual Basic, Visual Foxpro o C en la universidad. , pero pueden usarlo en el trabajo. Las aplicaciones son absolutamente raras. Además, estos lenguajes son muy ineficientes en el campo de las ciencias de la vida y no se recomienda el aprendizaje.
Los tres lenguajes más utilizados en Life Letters son Shell + R + Python/Perl, y los dos primeros son la base para asegurar que completes el análisis del proyecto. En la clase, también explicaremos los conocimientos básicos de lenguaje Shell y R que los biólogos deben dominar para garantizar que pueda usar la plataforma de análisis metagenómico de manera eficiente y estable, y asegurar las habilidades requeridas para el análisis de big data y visualización posterior a la etapa de publicación. . Proporcionamos un video de aprendizaje para una vista previa por adelantado .

Figura 3. Esquema de aprendizaje de Shell y R, el primer clic del mouse en Rstuio para completar el script de Shell y el análisis del lenguaje R, que no solo abre la puerta a la información de los estudiantes, sino que tampoco aumenta el costo de tiempo de los biólogos
Cuando use unas pocas horas para caminar hacia la puerta del análisis y la visualización de big data, descubrirá un mundo completamente nuevo. Muchas personas sentirán que es demasiado tarde para conocerse, enamorarse del análisis y tomar el carril rápido de la vida a partir de ese momento. Incluso si no está interesado en la programación, los conceptos utilizados aquí definitivamente lo beneficiarán de por vida, y obtendrá el doble de resultados con la mitad del esfuerzo en futuros análisis relacionados, y será mejor que otros. Además , incluso los estudiantes de primaria están aprendiendo Python ahora , y si no, los niños no podrán cuidarlo.
3. Temas de interpretación y dibujo de cartas
En vista del hecho de que muchos maestros carecen de una base sistemática en información de la vida, no entienden ni analizan los gráficos en los artículos, y no pueden dibujar varios gráficos, hemos lanzado las siguientes dos series, un total de 16 artículos originales, dibujo en 8 gráficos y lenguaje R Explicar.
Interpretación de gráficos de amplicones: comprensión de las ideas de artículos
Mapeo estadístico de amplicones: artículos de puntaje alto de impacto
Pero estas son solo introducciones. Durante la capacitación, combinaremos los artículos de alto nivel publicados para explicar con más detalle los principios y el alcance del uso de 16 diagramas de análisis de uso común, de modo que no solo pueda comprender los diagramas, sino también saber cómo aplíquelos a su propia investigación y podrá terminar de dibujar fácilmente.
Para resolver el problema del alto tiempo de aprendizaje y el costo del uso del dibujo en lenguaje R, el equipo de Yishengxin ha desarrollado un sitio web de dibujo gratuito para 16 dibujos de uso común , dibujo con un solo clic y el estilo individual del dibujo se puede modificar haciendo clic en los parámetros con el ratón.
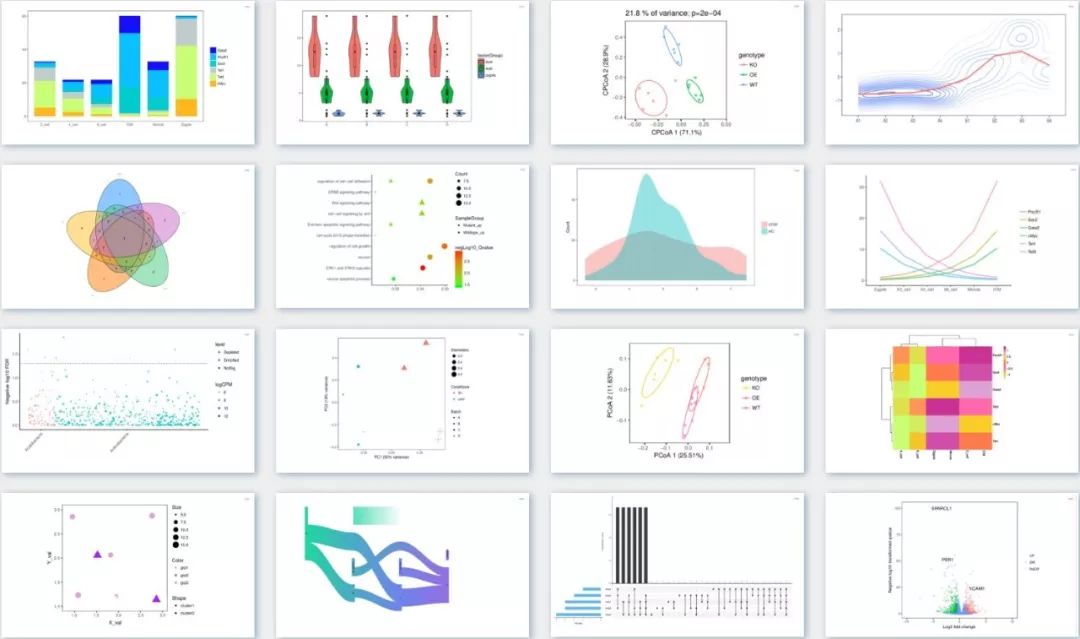
Figura 4. El significado, escenarios de uso y dibujo de 16 gráficos de uso común. Esto se puede lograr utilizando nuestra herramienta de dibujo en línea.
Con el fin de hacer que varias imágenes estadísticas logren imágenes grupales de nivel de publicación, se configura especialmente una clase de edición de imágenes y composición tipográfica de Adobe Illustrator para explicar las habilidades básicas de uso y dominar fácilmente la esencia, de modo que los gráficos de su artículo puedan estar en línea con CNS , y conviértase fácilmente en un laboratorio de edición de imágenes y maestros de rompecabezas.
Figura 5. Un ejemplo de un subgráfico de composición tipográfica de IA es un diagrama de grupo de nivel de publicación de CNS (Science, artículo de portada de 2016)
4. Visión general de la metagenómica
Después de construir una base de investigación científica integral el primer día, comenzaremos el viaje del análisis metagenómico de big data.
Como conocimientos básicos profesionales, aprenderemos lo siguiente.
Antecedentes: Microbioma Internacional, Proyecto Microbioma de China
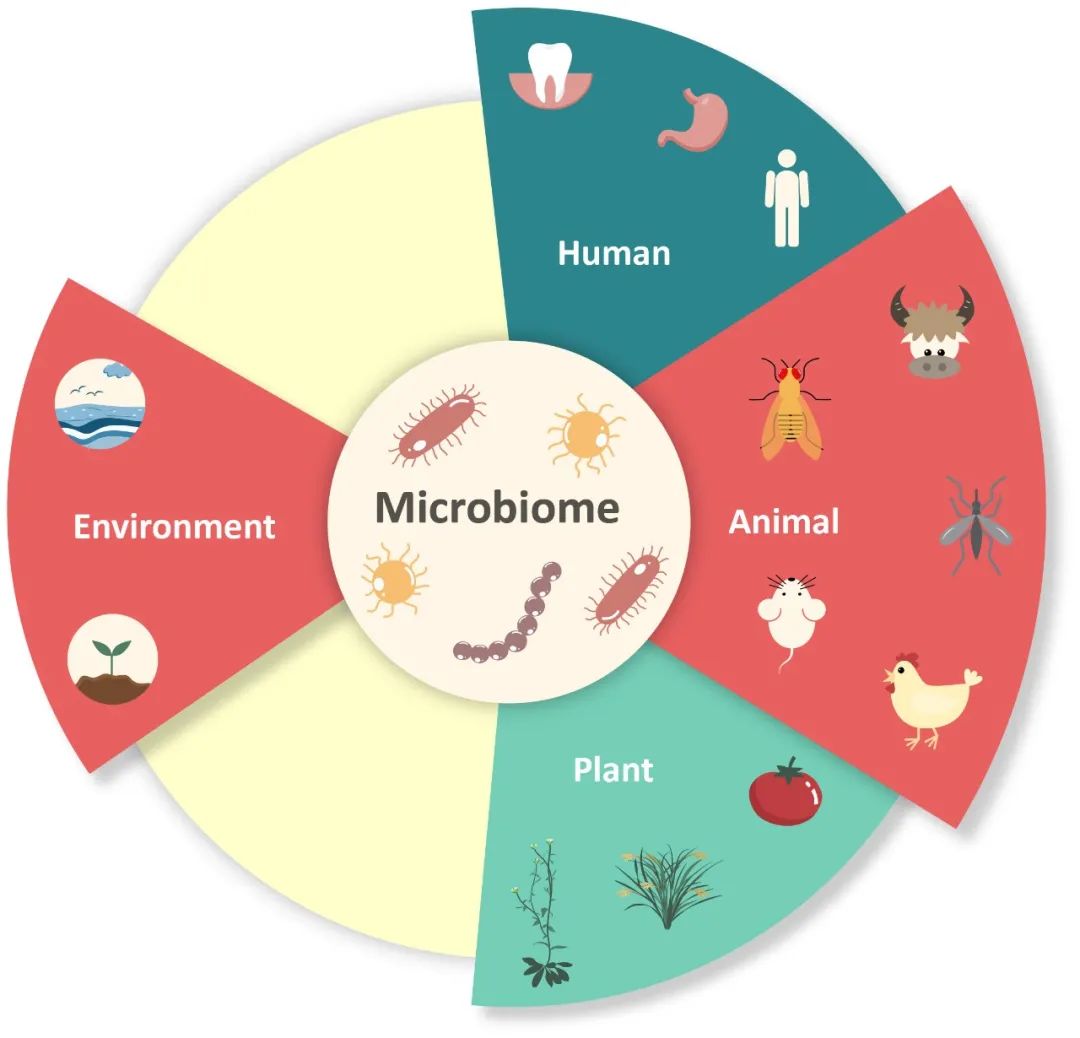
Objeto de investigación: humano, animal, vegetal, medio ambiente
Métodos de investigación: ómicas de cultivo, amplicón, metagenoma, metatranscriptoma, metaproteoma, metaboloma, análisis de asociación de metagenoma, macroepoma...
Puntos críticos de investigación de la metagenómica: grupo de cultivo, bacterias y enfermedades intestinales, análisis de asociación de metagenómica (MWAS), análisis conjunto multiómico...
Historia y principios del desarrollo de la secuenciación
Selección de preparación de muestras, réplicas experimentales y volumen de datos de secuenciación
Rutinas comunes para el análisis metagenómico de artículos SCI
Comparación de las ventajas y desventajas de la metagenómica y los amplicones
Evaluación de datos sin procesar y juicio de los resultados del ensamblaje
Figura 6. Métodos de metagenómica comúnmente utilizados: preguntas científicas que pueden responderse mediante amplicones, metagenómica y metatranscriptomas
5. Proceso de análisis paramétrico de la metagenómica
Solo comenzando con los datos de varios gigabytes a decenas de gigabytes por muestra, si no tiene forma de comenzar, se recomienda realizar un conjunto de análisis de parámetros de inmediato para obtener rápidamente la composición de especies y la composición funcional de la muestra. El método basado en referencia, como sugiere el nombre, consiste en utilizar directamente la base de datos actual de anotación de genes funcionales y especies, y los datos pueden obtener rápidamente la matriz de abundancia relativa de las especies correspondientes y los genes funcionales solo a través del control de calidad y la comparación. Este método también es muy elogiado en la última revisión de Rob Knight, el primer experto en análisis en este campo, " Nature Review | Rob Knight y otros te enseñan cómo analizar datos de flora (traducción de texto completo: 18,000 palabras) ".
Este método tiene ventajas obvias, pocos pasos, alta velocidad, ahorro de tiempo y mano de obra, y es adecuado para campos con buenas bases de datos de referencia, como el tracto intestinal humano, organismos modelo y océanos. La desventaja es que los genes funcionales de las especies no reportadas no se pueden identificar y se perderá mucha información al analizar plantas, suelo y muestras ambientales extremas.
Figura 7. La idea básica del análisis metagenómico: el proceso de análisis con parámetros . La composición de especies se obtiene principalmente a través de MetaPhlAn2 con base en todos los genomas microbianos informados, y la composición funcional se determina con base en bases de datos de proteínas como UniRef, EggNOG y KEGG. Los datos del amplicón 16S en sí solo contienen la composición de especies, y la composición funcional de KEGG/COG se puede obtener a través de PICRUSt.
Principales puntos de conocimiento:
1. Principios de escritura del diseño experimental
2. Proceso rápido de control de calidad y eliminación de host de KneadData
3. Cuantificación de la composición de especies MetaPhlAn2
4. Cuantificación de Componentes Funcionales de HUMAnN2
6. Proceso de análisis sin parámetros de la metagenómica.
El análisis sin parámetros del metagenoma tiene dos propósitos principales: uno es obtener especies no anotadas y expresión génica; el otro es extraer los genomas de nuevas especies a través de Binning. Se ve hermoso, pero la operación real requiere mucho cálculo. El proceso de análisis implica más pasos, como ensamblaje, predicción de genes, construcción de conjuntos de genes no redundantes y anotación de genes.
Figura 8. Proceso de análisis del metagenoma sin parámetros .
Pasos clave y software utilizado:
Control de calidad de datos fastqc, Trimmomatic, MultiQC, khmer
Construcción de conjuntos de genes no redundantes: CD-HIT
Estimación de la abundancia de genes: el salmón y otros métodos pueden cuantificar rápidamente la abundancia de genes , y se pueden realizar comparaciones posteriores de las diferencias generales del grupo, como PCA, PCoA y CCA; edgeR, MetaStat y LEfSe también se pueden usar para analizar más a fondo las diferencias en los genes entre grupos;
Anotación de especies: obtenga información de anotación de especies de conjuntos de genes no redundantes, o use Kraken2 para la anotación directa de especies en el nivel de lectura, combinado con el valor de abundancia en el paso 6 para analizar las diferencias de especies entre grupos;
Anotación de clasificación de función de genes: vía metabólica (KEGG), anotación de grupo de genes homólogos (eggNOG), combinada con 6 abundancias para la comparación funcional de las diferencias de grupo;
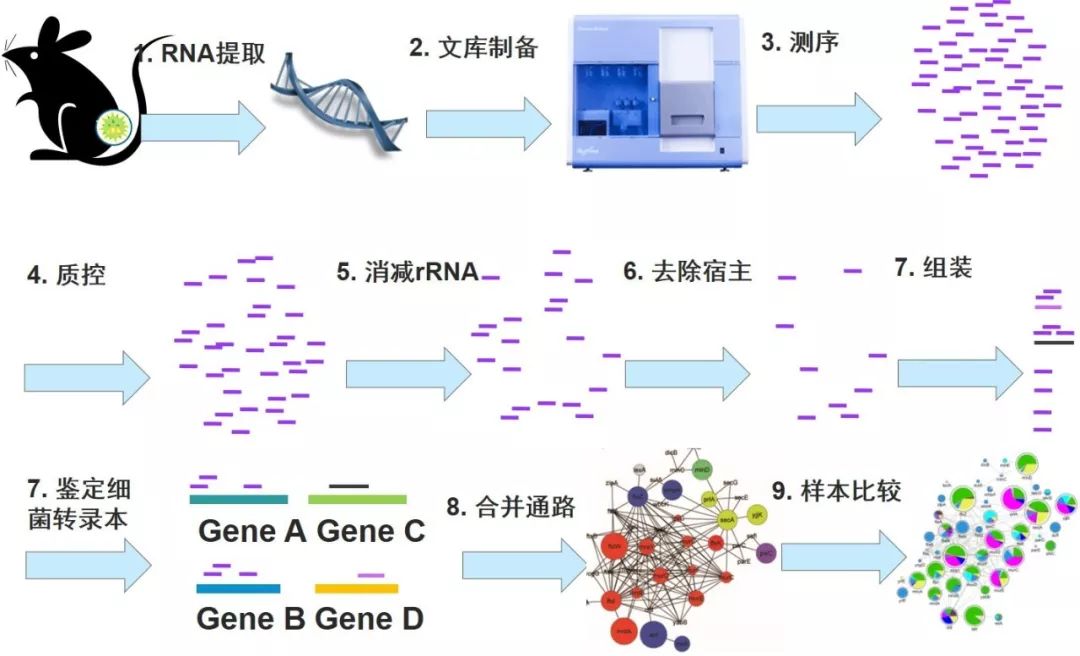
Figura 9. Flujo de trabajo de análisis de metatranscriptoma . Metatranscriptoma tiene un paso más para eliminar la secuencia del gen rRNA que la metagenómica. La desventaja de este método es que no se puede obtener la composición de especies real, pero refleja la composición de nivel de expresión de genes funcionales y especies activas en condiciones espacio-temporales específicas.
7. Análisis y visualización avanzados
Lenguaje R Dibujo estadístico y computación repetible
Identificación de bacterias individuales en la metagenómica (bin): MetaWRAP
Evaluación y visualización de resultados de contenedores: CheckM, VizBin
Visualización del Metagenoma: Circos
Procesos en línea: MEGAN, MG-RAST, EBI-metagenome
Construcción de árboles de uniones multigénicas: RaxML , fasttree, iTOL
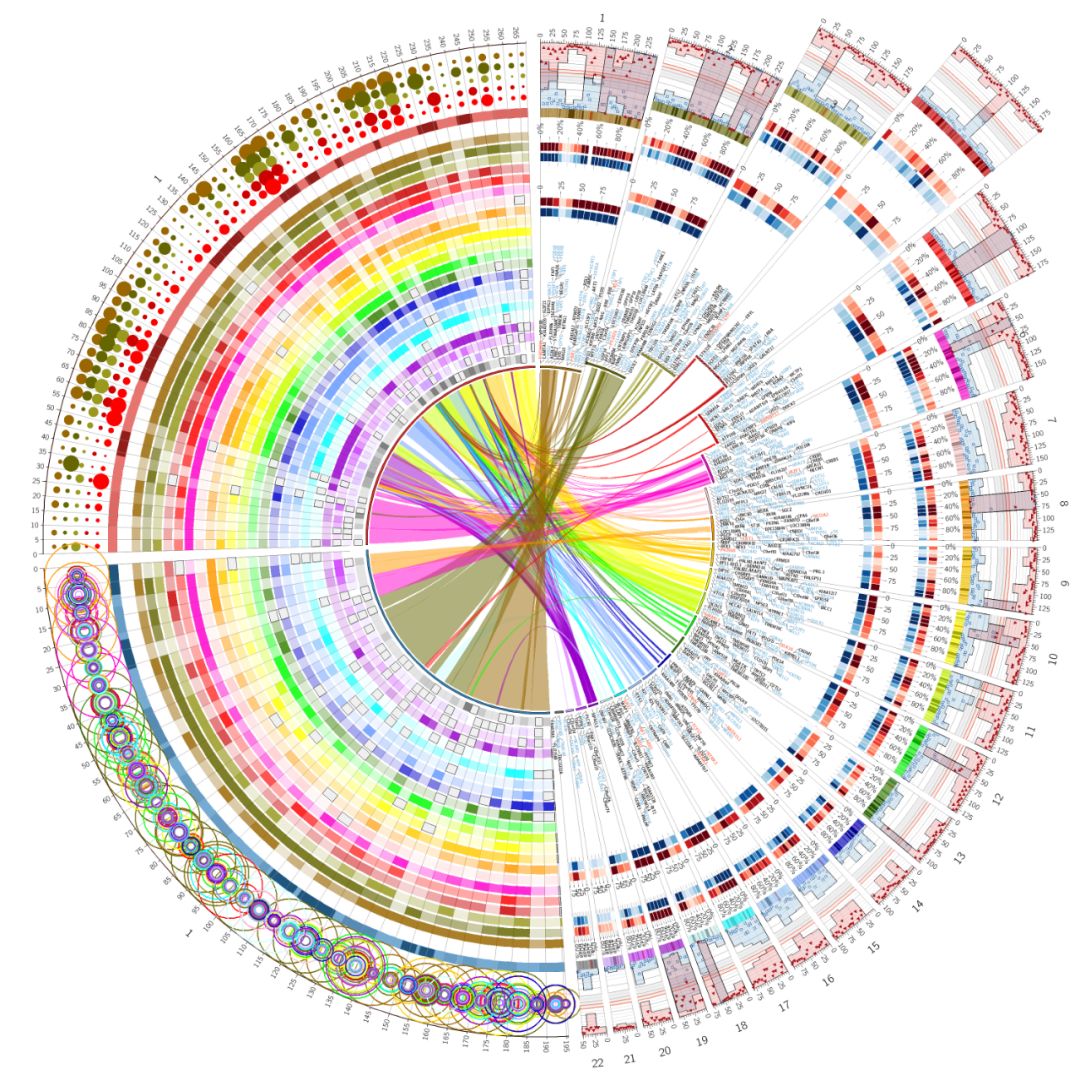
Figura 10. Visualización de composición, abundancia, cobertura y otra información del genoma del metagenoma
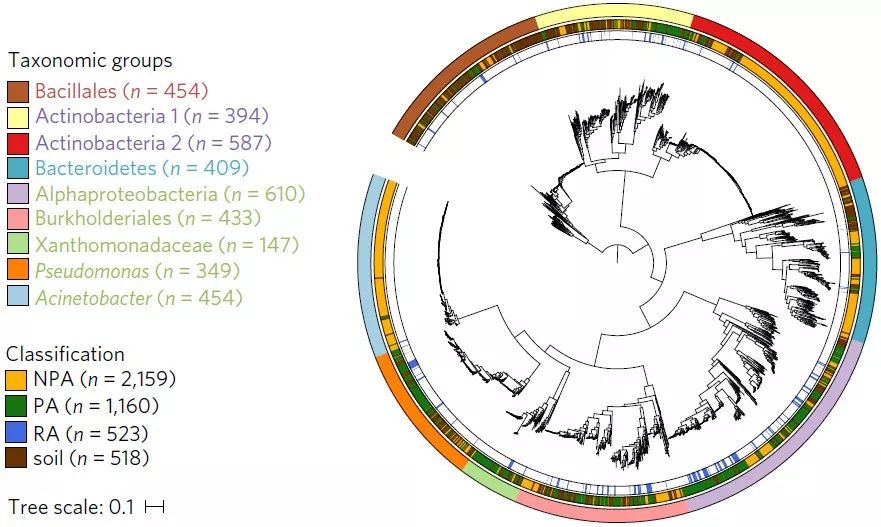
Figura 11. Construcción y embellecimiento del árbol evolutivo basado en la conexión poligénica (Levy-2018-NatureGenetics)
¿Qué puedes ganar después de estudiar este curso?
Comprender profundamente la idea básica de los datos de secuenciación biológica
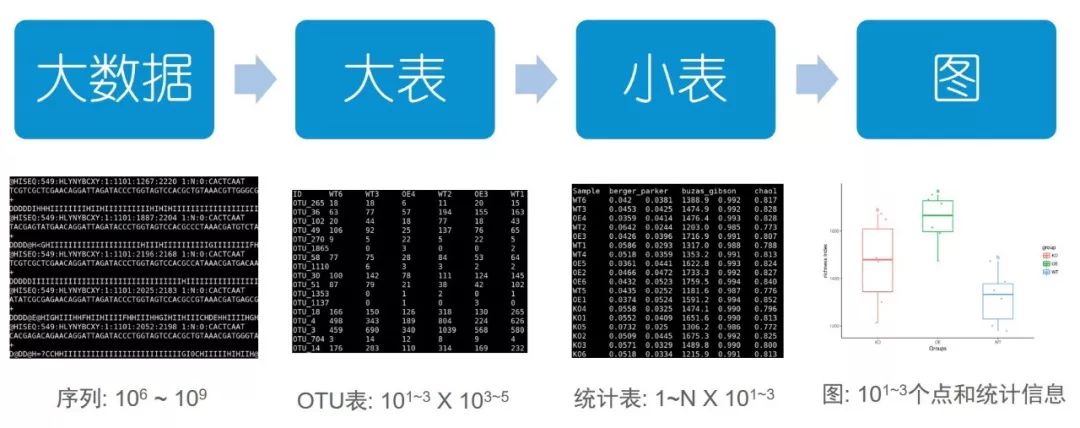
Una solución integral para tres modos de análisis metagenómico, así como análisis estadístico de los resultados.
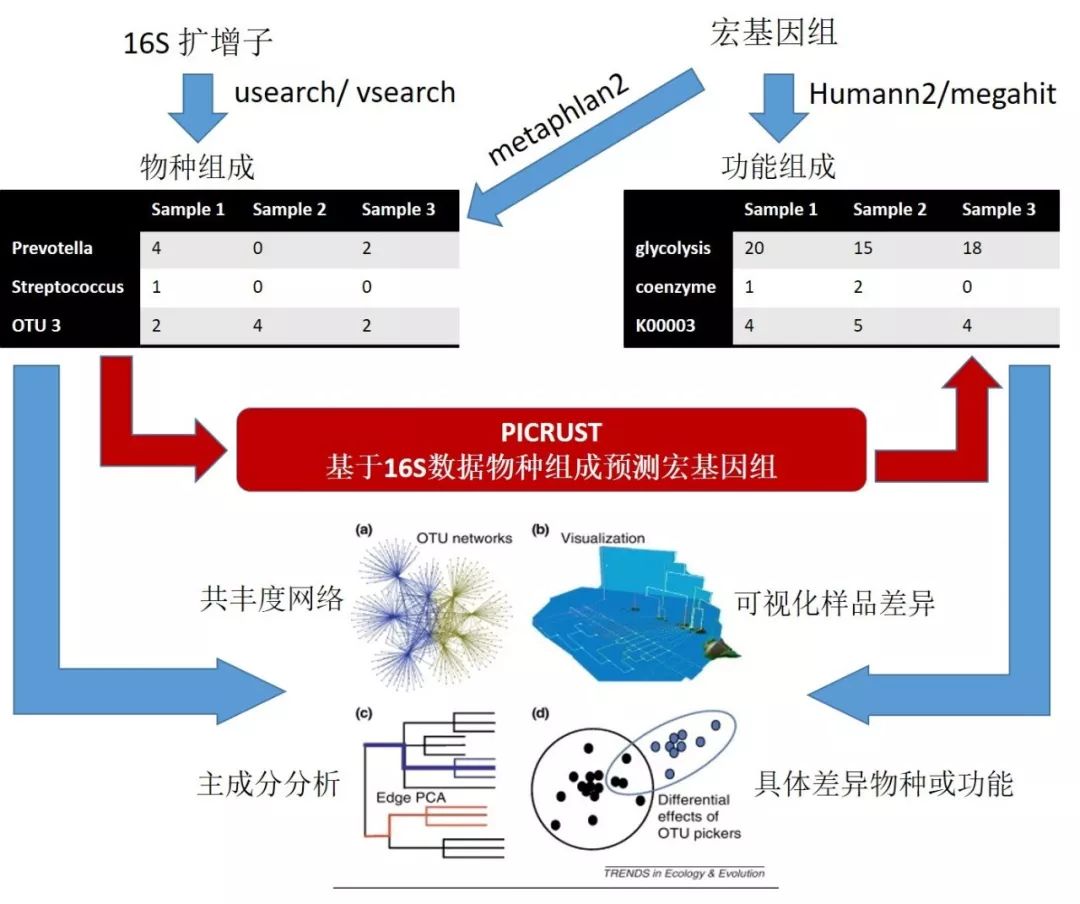
Datos de amplicón 16S Metagenómica de predicción de PICRUST
Humann2 cuantificación de especies y funciones a partir de datos metagenómicos
Binning y ensamblaje metagenómico de Denovo
Experiencia en el uso de docenas de bases de datos de software.
Docenas de tutoriales de instalación y uso de software en este campo
Comprensión y uso de bases de datos de anotaciones funcionales comunes
Visualización de resultados exigentes
Comparación de diferencias en los resultados
Varios esquemas de visualización.
Conferenciante
Los disertantes incluyen el Instituto de Microbiología, el Instituto de Genética y Desarrollo, el Instituto de Genoma, el Instituto de Biofísica, la Academia China de Ciencias, la Academia China de Ciencias Agrícolas, la Universidad de Tsinghua, la Universidad de Pekín, Zhongnong y muchos otros expertos técnicos de primera línea en este campo.
Liu Yongxin , PhD en Bioinformática, Investigador, Supervisor de Doctorado, Editor Ejecutivo en Jefe de iMeta Journal, Fundador de Metagenome Official Account. Las direcciones de investigación son el desarrollo de métodos de microbioma, la investigación de la función del microbioma alimentario y la comunicación científica. En la actualidad , ha publicado más de 50 artículos en Science , Nature Biotechnology , Cell Host & Microbe y otras revistas como primer autor (incluido el coautor) o la persona a cargo del análisis de datos del microbioma , con más de 12 000 citas citadas. Participante del proyecto plataforma de análisis de microbioma QIIME 2 . Invitado a publicar revisiones de métodos de investigación de microbiomas en Protein & Cell , Current Opinion in Microbiology , Genetics y otras revistas como primer autor y/o autor correspondiente (incluidos los coautores) . En julio de 2017, fundó la cuenta pública "Metagenomics", y actualmente comparte más de 3100 artículos originales en este campo. Los trabajos representativos incluyen "Interpretación de gráficos de microbioma, proceso de análisis y dibujo estadístico" , "Tutorial chino QIIME2" , etc. con 15 seguidores 10,000+, lectura acumulativa 40 millones+.
Chen Tong , Ph.D., se graduó del Instituto de Genética y Biología del Desarrollo, Academia de Ciencias de China en 2015, Ph.D. en bioinformática, en Cell Stem Cell (IF=23.2, primer autor y artículo de portada), Nature Communications , Nucleic Acids Research X 4, Protein & Cell y otras revistas de alto nivel publican artículos con el primer o principal autor, y operan la cuenta oficial de WeChat de "Life Letter Collection" que es seguida por decenas de miles de personas , brindándole una experiencia diferente en el aprendizaje de las letras de la vida.
Momentos de cursos anteriores
Los alumnos provienen principalmente de universidades e institutos de investigación de China continental, y también hay investigadores de importantes empresas como Moutai, Wuliangye, Angel Yeast, Huawei, etc., e incluso chinos de ultramar que han viajado miles de kilómetros desde Europa, Australia, Estados Unidos, Canadá, Nueva Zelanda, Singapur, Tailandia y otros lugares vuelan a Beijing o participan en seminarios especiales de aprendizaje en línea.





equipo asistente
Más de una docena de médicos (incluidos estudiantes) de la Academia de Ciencias de China, la Universidad de Tsinghua y la Universidad de Pekín, así como profesores rotativos y asistentes de enseñanza, ayudan a los estudiantes a aprender y corregir deficiencias en el proceso de formación.
modo de enseñanza
Este curso se centra en explicar el proceso y el funcionamiento práctico, y adopta un método de enseñanza original de cuatro etapas:
La primera etapa son 3 días de enseñanza intensiva;
La segunda etapa es de auto-práctica durante 2 semanas;
La tercera fase de preguntas y respuestas en vivo en línea;
La cuarta etapa del video de entrenamiento para seguir aprendiendo;
Realizar la coordinación unificada de los cuatro eslabones de enseñanza-práctica-respuesta-uso.
periodo de entrenamiento
De 9:00 am a 6:00 pm todos los días, enseñanza semicerrada (la última hora es para mesa redonda para aumentar la interacción. El último día será un poco más temprano para permitir más tiempo para la discusión, y también es conveniente para que el profesor regrese en coche)
Hora de inscripción: el día de la clase
lugar de enseñanza
Clases simultáneas en línea y fuera de línea: plataformas de conferencias en línea, como Tencent Conference.
La ubicación fuera de línea es C1105, Bloque C, Edificio Internacional Caizhi, No. 18 Zhongguancun East Road, distrito de Haidian, Beijing.
precio del curso
4.500 yuanes/persona dos semanas antes del inicio de clases
El número de plazas es limitado, y el canal de inscripción se cerrará automáticamente a partir de 40 personas inscritas en cada curso
Proporcionar oportunidades de pasantías u oportunidades de trabajo en Yihanbo Gene Technology
Beneficios del curso
Los asientos se clasifican de adelante hacia atrás según el orden de registro y pago exitosos (o prepago)
Dar un curso básico de programación (http://bioinfo.ke.qq.com)
Si varias personas (N, 10>N>1) se registran en un grupo y pagan la tarifa al mismo tiempo, cada persona también puede deducir N-1 cien yuanes (hasta 500)
Un disco Kingston U gratuito (32G que incluye datos de entrenamiento y scripts)
Adjunte la recomendación y comparta la información de inscripción correspondiente en Moments, y envíe la captura de pantalla a [email protected] para obtener un cupón de curso presencial Tencent de 200 yuanes para libros de cartas para estudiantes (se puede dividir para varios cursos)
Yishengxin lanzó una serie de cursos relacionados al mismo tiempo, ¡con descuentos continuos! Amplicon (proyecto de investigación preliminar) + metagenómica (alta precisión), les deseo un mayor nivel de análisis.
Precauciones *
Debe traer su propia computadora portátil, se recomienda usar el sistema Win10, 4G o más memoria (se recomienda 8G). La práctica del curso proporcionará una plataforma de computación en la nube según sea necesario
Todos los datos del curso de capacitación, los documentos son materiales internos, solo como referencia, y no se reproducirán para su publicación sin permiso.
La grabación y la grabación de video están prohibidas durante la clase.
Los estudiantes que hayan pagado con éxito pueden solicitar una extensión y cambiar a una clase de capacitación de seguimiento si no pueden venir temporalmente debido a asuntos urgentes; también pueden solicitar un reembolso
Si solicita un reembolso 2 semanas (inclusive) antes del inicio del curso, se puede reembolsar el 85% de la tarifa; si solicita un reembolso 3 días hábiles (inclusive) antes del inicio del curso, puede reembolsar el 70 % de la tarifa (si la factura ha sido emitida, debe asumir la tarifa de gestión correspondiente)
No se puede posponer primero y luego reembolsar
Para obtener introducciones más detalladas de los cursos, escanee el código QR a continuación.

Yishengxin lanzó una serie de cursos relacionados al mismo tiempo, ¡y hay descuentos por registro continuo y compras grupales!
连报优惠——Registro paranmúltiples cursos, cada curso esn-1100 yuanes más barato;El descuento para cursos de matrícula consecutiva no aparece en el primer curso, sino en forma de acumulación en los siguientes cursos, es decir, el descuento para el segundo curso está disponible solo después de completar el primer curso.
老学员优惠:El segundo curso se reducirá en 100 y el tercer curso se reducirá en 200. En la siguiente analogía, la cantidad máxima no excederá los 500 yuanes.
Descuentos para grupos de varias personas, el rango de descuento es de
参团人数-1100 yuanes (si un miembro del grupo se retira al pagar en el momento de la inscripción, el descuento se calculará de acuerdo con el número real de participantes).
4. El precio final mínimo después del descuento no es inferior a 4.000 yuanes. La información preferencial cambia dinámicamente, y el precio está sujeto al precio correcto finalmente calculado por el sistema.
¡También se puede descontar al mismo tiempo que la compra grupal! Se recomienda estudiar amplicón (principiante) + metagenómica (avanzado) en orden. Te deseo un mayor nivel de análisis y te conviertas en una persona indispensable en el experimento. ¡Date prisa e inscríbete! .
Para convertirte en una persona indispensable en el experimento, copia el enlace http://www.ehbio.com/Training/ o haz clic para leer el texto original , ¡regístrate ahora!
