部分参考自:生物信息学100个基础问题 —— 番外2: 用Anaconda快速搭建生物信息学分析平台 - 孟浩巍的文章 - 知乎 https://zhuanlan.zhihu.com/p/35711429
1.首先将别的文件夹下的fastq.gz文件复制到我的文件夹下,首先进入到要复制的文件目录下:
cp ERR522819_2.fastq.gz ../../xhs
使用这个命令复制到xhs文件夹下
2.使用下列命令查看Linux系统是32位还是64位:
getconf LONG_BIT
显示是64位系统。
3.使用命令下载anaconda
wget https://repo.anaconda.com/archive/Anaconda2-5.3.0-Linux-x86_64.sh
并且安装anaconda:
bash Anaconda2-5.3.0-Linux-x86_64.sh
并且在配置PATH时使用以下命令进行更新
source ~/.bashrc
4.设置bioconda的channel,一行一行地输入
conda config --add channels defaults conda config --add channels conda-forge conda config --add channels bioconda
5.使用conda安装fastqc
conda install fastqc
6.使用以下命令添加清华源,并且可以使用conda info查看结果,
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free/ conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main/ conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge/
7.遇到了问题,第5步之后,
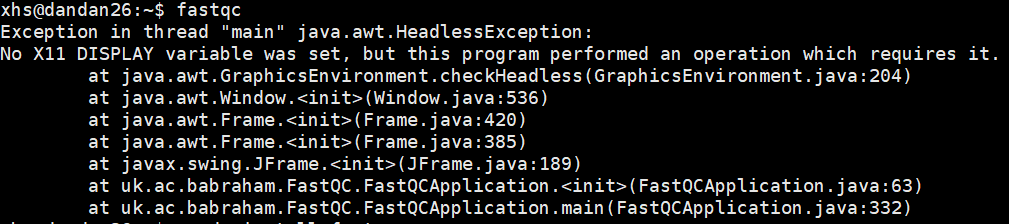
Exception in thread "main" java.awt.HeadlessException: No X11 DISPLAY variable was set, but this program performed an operation which requires it. at java.awt.GraphicsEnvironment.checkHeadless(GraphicsEnvironment.java:204) at java.awt.Window.<init>(Window.java:536) at java.awt.Frame.<init>(Frame.java:420) at java.awt.Frame.<init>(Frame.java:385) at javax.swing.JFrame.<init>(JFrame.java:189) at uk.ac.babraham.FastQC.FastQCApplication.<init>(FastQCApplication.java:63) at uk.ac.babraham.FastQC.FastQCApplication.main(FastQCApplication.java:332)
原因:因为fastqc运行需要可视化界面,但是这个linux并没有。
尝试1:在etc/profile下添加
export DISPLAY=localhost:0
失败。直接找到打开[rofile并添加失败,因为权限不够:
xhs@dandan26:/etc$ cat >>profile
-bash: profile: 权限不够
使用:
chmod 777 profile
提示:
chmod: 更改'profile' 的权限: 不允许的操作
总之就是各种权限不够无法修改,所以这个fastqc只能先放弃,待会问问其他人。[1]
8.安装fasxt-toolkit,先从官网上下载,http://hannonlab.cshl.edu/fastx_toolkit/download.html
wget http://hannonlab.cshl.edu/fastx_toolkit/fastx_toolkit_0.0.13_binaries_Linux_2.6_amd64.tar.bz2
并且使用命令查看系统版本:
lsb_release -a
结果:
No LSB modules are available. Distributor ID: Ubuntu Description: Ubuntu 16.04.5 LTS Release: 16.04 Codename: xenial
9.根据文档指导安装fastx-toolkit——http://hannonlab.cshl.edu/fastx_toolkit/install_ubuntu.txt
使用以下命令安装,提示已经有gcc环境了,然后使用gcc -v查看它的版本:
sudo apt-get install gcc g++ pkg-config wget
Install libgtextutils
wget http://cancan.cshl.edu/labmembers/gordon/files/libgtextutils-0.6.tar.bz2
又有错误了,顺着命令安装,到make命令时,就错了。
pipe_fitter.c: In function ‘pipe_close’: pipe_fitter.c:30:6: error: variable ‘i’ set but not used [-Werror=unused-but-set-variable] int i, status ; ^ cc1: all warnings being treated as errors Makefile:336: recipe for target 'pipe_fitter.lo' failed make[3]: *** [pipe_fitter.lo] Error 1 make[3]: Leaving directory '/home/xhs/libgtextutils-0.6/src/gtextutils' Makefile:235: recipe for target 'all-recursive' failed make[2]: *** [all-recursive] Error 1
给这些东西也不明白。尝试了这个不能行。
$ CXXFLAGS="-Wall" $ ./waf configure
按照CSDN论坛上的方法,将makefile文件中的-Werror(共2个)都去掉了:还是不行,并且重新断开连接之后,makefile中的-Werror还是会出现。[2]
想接着往下安装根本不行啊,因为之前的这个No package 'gtextutils' found,就进行不了后续的步骤了。哭唧唧。[3]
其中用到了./configure
——./configure是源代码安装的第一步,主要的作用是对即将安装的软件进行配置,检查当前的环境是否满足要安装软件的依赖关系,
又仔细看发现,有很多下面都有makefile
config.status: creating Makefile config.status: creating README config.status: creating doc/Makefile config.status: creating m4/Makefile config.status: creating src/Makefile config.status: creating src/gtextutils/Makefile config.status: creating gtextutils.pc config.status: creating tests/Makefile config.status: creating config.h config.status: config.h is unchanged config.status: executing depfiles commands config.status: executing libtool commands
共有6个makefile将其中的-werror都去掉,这个可以顺利安上,因为这个gtextutils安装不是在默认路径下,所以用下面的命令:
export PKG_CONFIG_PATH=/home/xhs/libgtextutils-0.6:$PKG_CONFIG_PATH
在这个路径下有gtextutils.pc文件,这样就可以找到了。
*那么.pc文件是什么呢?proc语法写成.pc文件。输入make安装fastx-toolkit时依旧是出现了和上述一样的error,但是本次由于makeprofile文件实在太多,所以选择使用命令。
使用unset PKG_CONFIG_PATH 命令可以清空设置的路径。
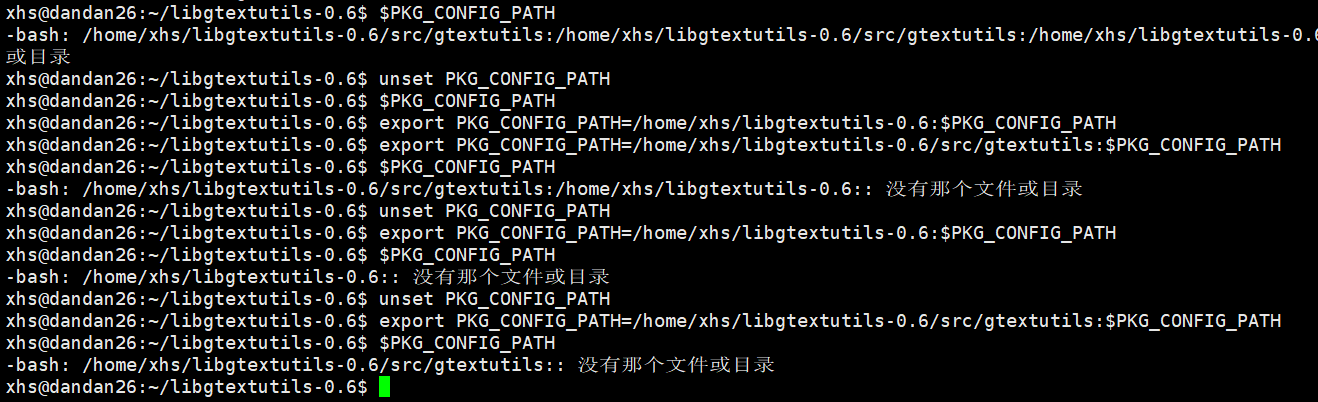
怎么就找不到了?
实在是搞不定了。
在fastx-toolkit下安装有:
Making all in fastx_collapser make[3]: Entering directory '/home/xhs/fastx_toolkit-0.0.12/src/fastx_collapser' make[3]: Nothing to be done for 'all'. make[3]: Leaving directory '/home/xhs/fastx_toolkit-0.0.12/src/fastx_collapser' Making all in fastx_uncollapser make[3]: Entering directory '/home/xhs/fastx_toolkit-0.0.12/src/fastx_uncollapser' g++ -DHAVE_CONFIG_H -I. -I../.. -I/usr/local/include/gtextutils -I../libfastx -I../libfastx -g -O2 -Wall -Wextra -Wformat-nonliteral -Wformat-security -Wswitch-default -Wswitch-enum -Wunused-parameter -Wfloat-equal -DDEBUG -g -O1 -DDEBUG -g -O1 -MT fastx_uncollapser.o -MD -MP -MF .deps/fastx_uncollapser.Tpo -c -o fastx_uncollapser.o fastx_uncollapser.cpp fastx_uncollapser.cpp:31:39: fatal error: gtextutils/stream_wrapper.h: 没有那个文件或目录 compilation terminated. Makefile:283: recipe for target 'fastx_uncollapser.o' failed make[3]: *** [fastx_uncollapser.o] Error 1 make[3]: Leaving directory '/home/xhs/fastx_toolkit-0.0.12/src/fastx_uncollapser' Makefile:252: recipe for target 'all-recursive' failed make[2]: *** [all-recursive] Error 1 make[2]: Leaving directory '/home/xhs/fastx_toolkit-0.0.12/src' Makefile:279: recipe for target 'all-recursive' failed make[1]: *** [all-recursive] Error 1 make[1]: Leaving directory '/home/xhs/fastx_toolkit-0.0.12' Makefile:209: recipe for target 'all' failed make: *** [all] Error 2
没有哪个文件或目录,真是绝望。不知道怎么处理了。