prefácio
um pouco.
1 método
O efeito do uso de uma única rede neural para segmentação de imagens geralmente é de baixa precisão e não pode atender às expectativas; portanto, o autor otimiza a integração de redes neurais profundas para se adaptar à tarefa de segmentação de tumores cerebrais. Especificamente, é proposto um método de ensemble leve composto por 2 redes, cada uma das quais é treinada seletivamente no conjunto de treinamento. As saídas dessas redes são mapas de segmentação que diferem na segmentação de sub-regiões tumorais e, finalmente, os mapas de segmentação são combinados para obter a previsão final. Os detalhes de treinamento dessas duas redes são os seguintes.
1.1 Sub-rede 1: 3D - ConvNet
O primeiro submodelo usado no conjunto é o 3D-ConvNet. Ele usa unidades multifibras com convoluções atrosas ponderadas para representações multiescala para segmentação de volume 3D, conforme mostrado na Figura 1. Além disso, a rede é ajustada para melhorar a segmentação.

Antes que os dados sejam inseridos na rede para treinamento, os dados são aprimorados usando várias técnicas de aumento de dados, como corte, rotação e espelhamento. O modelo é treinado em 150 épocas usando um tamanho de bloco de 128×128 e combinando as funções de perda de dados e perda focal. Os hiperparâmetros ajustados são mostrados na Tabela 1.

O preenchimento zero é aplicado aos dados de ressonância magnética de modo que os voxels originais de tamanho 240 × 240 × 155 sejam convertidos em 240 × 240 × 160, uma profundidade exatamente divisível pela rede. Uma vez que os dados estejam prontos para validação, a rede treinada é usada para gerar mapas de probabilidade. Esses mapas de probabilidade são então usados para integrar e prever o resultado final.
1.2 Sub-rede 2: 3D - ConvNet em forma de U
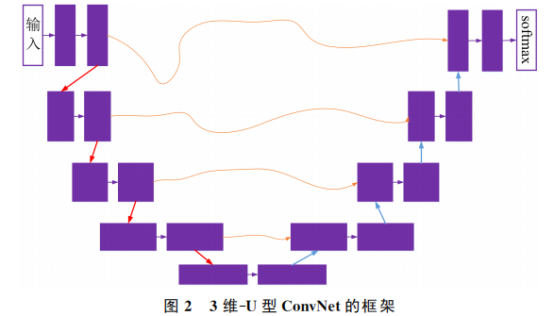
O segundo submodelo da integração é uma variante U-Net tridimensional que é diferente da arquitetura U-ConvNet clássica. A diferença é que a função de ativação ReLU é substituída por Leaky-ReLU e normalização de instância (instance normalization) é usado. , referido como IN) para substituir a normalização do lote (normalização do lote, referido como BN). Use-o para treinar do zero no conjunto de dados MBTSC-2019, conforme mostrado na Figura 2, onde a caixa roxa representa a unidade de convolução 3D, a linha vermelha representa a operação de agrupamento máximo, a linha azul representa a operação de upsampling trilinear (trilinearupsampling) e as linhas laranja representam operações de mesclagem. A rede também é ajustada para melhorar o efeito de segmentação.
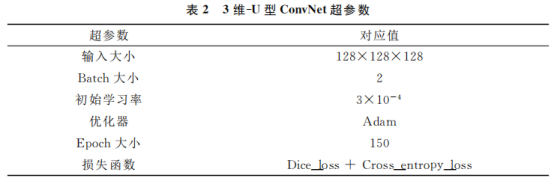
Reduza o tamanho da ressonância magnética recortando os dados. Em seguida, reamostrar através do espaço voxel mediano de outros dados heterogêneos, seguido pela normalização do escore z. Para treinar a rede, o tamanho do bloco de entrada é 128 × 128 × 128 voxels e o tamanho do lote é 2. Diferentes técnicas de aprimoramento de imagem, como rotação, inversão de espelho e correção de gama, são aplicadas aos dados durante o aprendizado para evitar o overfitting, melhorando assim a precisão da segmentação do modelo. A função de perda usada combina a função de perda de entropia cruzada e a função de perda de dados. A Tabela 2 detalha os hiperparâmetros no processo de treinamento.
A validação é realizada em patches de imagem onde todos os patches de imagem se sobrepõem pela metade do tamanho e os voxels próximos ao centro têm um peso maior atribuído a eles. Durante o teste, dados de aumento adicionais foram obtidos pela inversão do espelho ao longo do eixo da imagem. A saída do 3D-U ConvNet também é um mapa de probabilidade para integração.
1.3 Detalhes do método de integração
O método ensemble proposto pelo autor não é construído por uma média simples das previsões (ou seja, mapas de probabilidade) gerados pelos dois modelos, mas uma estratégia de "integração otimizada" é proposta após testes rigorosos, após os quais os dois modelos são combinados A saída é mostrada na Figura 3.
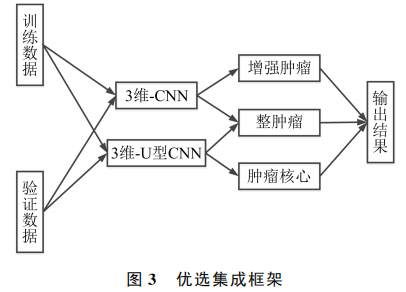
Teste a rede treinada no conjunto de verificação para obter os resultados de segmentação de imagem correspondentes. As previsões de modelos individuais são avaliadas independentemente no servidor MBTSC online para determinar sua eficácia na segmentação de regiões tumorais. Em seguida, as pontuações dos dados dos dois modelos são comparadas para determinar qual sub-rede é mais precisa e melhor que a outra para uma região específica do tumor. Após experimentos, descobriu-se que o 3D-ConvNet teve melhor desempenho na segmentação de tumores aprimorados. Ao mesmo tempo, o 3D-U ConvNet pode segmentar com mais precisão o núcleo do tumor. Finalmente, a combinação de previsões de ambas as redes supera os resultados de segmentação independentes no caso de tumores inteiros. Portanto, o esquema final de integração de três regiões pode ser obtido: (1) Para o núcleo do tumor, apenas a saída do 3D-U ConvNet é usada; (2) Para o tumor aprimorado, apenas a saída do 3D-ConvNet é usada usado; (3) para todo o tumor, atribua o mesmo peso à saída das duas sub-redes. Observe que a etapa de predição é avaliada no servidor online e, finalmente, a pontuação dos dados do método integrado do autor é obtida. Esses resultados serão discutidos com mais detalhes posteriormente.
1.4 Introdução a algumas técnicas
Aqui estão algumas técnicas que não são comumente usadas em redes profundas em geral, mas têm efeitos significativos quando aplicadas no método do autor:
(1) normalização do escore z
A normalização do escore Z, também conhecida como normalização do escore padrão, está na forma da diferença entre um número e a média e depois dividida pelo desvio padrão, conforme mostrado na fórmula (1)

Onde: μ é a média e δ é a variância. Por meio dele, os dados de diferentes dimensões podem ser convertidos em um z-score de dimensão uniforme e depois comparados, melhorando assim a comparabilidade dos dados e enfraquecendo ao máximo a interpretabilidade dos dados. Além disso, também pode evitar a covariância matriz de ser mal condicionada, equivale a transformar a matriz de covariâncias em uma matriz de coeficientes de correlação, menos suscetível a perturbações.
(2) Correção gama
A correção de gama é para calibrar a curva de gama da imagem (no campo da imagem de computador, a curva de relação de conversão entre a tensão de saída da tela e o brilho correspondente é chamada de curva de gama) para executar a edição de tom não linear na imagem. Ele pode detectar a parte escura e a parte clara no sinal da imagem e aumentar a proporção dos dois, de modo a melhorar o efeito de contraste da imagem, conforme mostrado na fórmula (2), o núcleo da correção gama está entre colchetes.
![]()
Entre eles: Iout é o valor do pixel de saída, Iin é o valor do pixel de entrada e gamma é o fator de correção.
(3) pontuação de dados e função de perda de dados
A pontuação do dado é uma função usada para medir a similaridade de um conjunto, geralmente usada para calcular a similaridade de duas amostras, e o intervalo de valores é [0, 1], conforme mostrado na fórmula (3)

Para a tarefa de segmentação de imagens neste artigo, X representa a imagem segmentada com o rótulo de valor verdadeiro e Y representa a imagem segmentada da saída prevista, o denominador representa a soma do número de pixels em X e Y e o numerador representa duas vezes a interseção de X e Y (multiplicada por 2 para eliminar o efeito da contagem dupla de pixels de interseção X e Y no numerador). A perda de dados é expressa como

(4) Perda de entropia cruzada e função de perda focal
A forma da função de perda de entropia cruzada é mostrada na fórmula (5)
![]()
Onde: d é um vetor de rótulo booleano e y é o vetor de saída de uma função de ativação de ponto flutuante. Pode-se ver que para entropia cruzada ordinária, para amostras positivas, quanto maior a probabilidade de saída, menor a perda. Para amostras negativas, quanto menor a probabilidade de saída, menor a perda. Neste momento, a função de perda converge lentamente no processo iterativo de um grande número de amostras simples e pode não ser otimizada para o ótimo.
Melhora da função de perda focal 1

Em primeiro lugar, um fator de peso é adicionado à função de perda de entropia cruzada original, de modo que mais atenção seja dada às amostras que são difíceis de distinguir, onde γ é geralmente definido como 2.
Melhora da função de perda focal 2

A função de perda focal também introduz um fator de equilíbrio α para equilibrar a proporção desigual entre amostras positivas e negativas. O valor de α varia de 0 a 1. Quando é 0,5, a proporção de d=1 pode ser aumentada relativamente para garantir o equilíbrio de amostras positivas e negativas.
2 Resultados experimentais e análises
2.1 Conjunto de dados
O desafio de segmentação de tumor cerebral multimodal [16] (desafio de segmentação de tumor cerebral multimodal, conhecido como MBTSC) é uma plataforma para avaliar o desenvolvimento de métodos de segmentação de tumor baseados em aprendizado de máquina. O conjunto de dados de referência inclui imagens de ressonância magnética 3D de glioma ( LGG e HGG ) e rótulos de verdade anotados por médicos profissionais, onde as imagens de varredura multimodal fornecidas podem ser usadas para treinamento e validação de redes neurais para tarefas de segmentação específicas [6, 11].
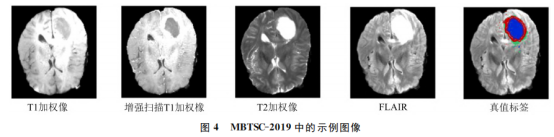
O conjunto de dados MBTSC-2019 [16] é usado para experimentos. O conjunto de dados MBTSC-2019 é um conjunto de dados abrangente obtido de 19 organizações e instituições diferentes, incluindo exames de ressonância magnética multimodais de cada paciente, ou seja, imagens ponderadas em T1, imagens aprimoradas ponderadas em T1, imagens ponderadas em T2 e FLAIR, suas sub-regiões tumorais foram segmentado. Esses dados foram pré-processados para normalização: foram removidos cranialmente e alinhados para corresponder ao modelo anatômico e reamostrados com resolução de 1 mm3. A dimensão de cada sequência é 240×240×155. Imagens de exemplo do conjunto de treinamento e seus rótulos reais correspondentes são mostrados na Fig. 4, onde diferentes modalidades de regiões tumorais são codificadas por cores: tumores aprimorados são representados em vermelho, tumores inteiros são representados em verde e núcleos de tumor são representados em azul e vermelho disseram. Pode ser visto na Figura 4 que o valor do rótulo manual destaca três regiões tumorais: edema peritumoral, tumor realçado, necrose e núcleo sem realce.
O conjunto de treinamento no MBTSC-2019 é usado para treinar o modelo, e o conjunto de validação é usado para avaliar o método de conjunto proposto. O conjunto de treinamento consiste em 259 pacientes HGG e 76 pacientes LGG, todos contendo rótulos de base anotados profissionalmente. O conjunto de validação inclui 125 exemplos de níveis de doença desconhecidos e os rótulos reais não são abertos ao público.
Vale ressaltar que o método proposto pelo autor pode efetivamente melhorar a taxa de precisão, mas ao contrário de alguns outros métodos, no experimento da fase de treinamento, o autor não utilizou nenhum conjunto de dados externo para o pré-treinamento. Além disso, como o acesso ao conjunto de testes MBTSC-2019 é limitado àqueles que participaram da competição, os resultados do teste são relatados com base no conjunto de validação MBTSC-2019. Os resultados da segmentação do método de conjunto proposto são primeiro relatados no conjunto de validação e, em seguida, comparados com os métodos de estado da arte existentes.
2.2 Resultados experimentais e análises
As 2 sub-redes são treinadas seletivamente no conjunto de treinamento MBTSC-2019 (n = 335) e testadas no conjunto de validação MBTSC-2019 fornecido (n = 125) e, em seguida, de forma inteligente Os mapas de segmentação desses modelos são combinados e, finalmente, é dada a previsão final do tipo de tecido tumoral. Os resultados finais mostram que a pontuação dos dados obtida pelo nosso método para segmentação aumentada do tumor é 0,749, a pontuação dos dados para a segmentação total do tumor é 0,905 e a pontuação dos dados para a segmentação do núcleo do tumor é 0,847. As Figuras 5-7 mostram um exemplo de resultados de segmentação para MRIFLAIR, que são a seção transversal, a seção coronal e a seção sagital em sequência. Para este exemplo, primeiro gere mapas de segmentação dos dois submodelos, respectivamente, e depois exiba a saída mesclada final. As pontuações dos dados para o tumor realçado dos pacientes, tumor total e núcleo do tumor foram 0,927, 0,947 e 0,924, respectivamente.

Diferentes técnicas de integração são analisadas posteriormente para determinar se existem diferenças entre esses métodos e para determinar o método mais preciso. Os resultados da análise estão listados na Tabela 3.
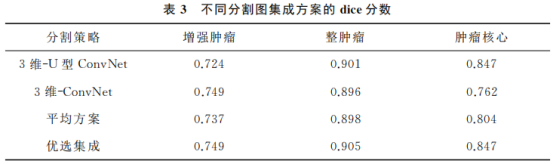
O ConvNet em forma de U pode segmentar com mais precisão o núcleo do tumor; no caso de tumores inteiros, os efeitos das duas sub-redes são relativamente próximos e os resultados combinados são ligeiramente melhorados. Comparado com a média simples, o esquema proposto de "integração ótima" tem melhor precisão.
2.3 Comparação com os 3 melhores métodos na classificação final da competição MBTSC-2019
O método ConvNet integrado otimizado proposto pelo autor foi avaliado no conjunto de validação MBTSC-2019 e, em seguida, comparado com as pontuações dos dados dos três melhores métodos da competição MBTSC-2019, os resultados estão listados na Tabela 4.
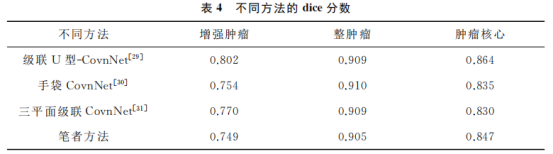
Pode-se observar na Tabela 4 que o U-CovnNet em cascata [29] obteve os melhores resultados no desafio, e os resultados obtidos pelo método do autor mostraram uma lacuna significativa no realce do tumor, enquanto para todo o tumor e o núcleo do tumor os resultados não são tão ruins; melhores resultados para núcleos tumorais do que CovnNet de bolso [30], e apenas uma pequena lacuna entre tumores aprimorados e núcleos tumorais. Da mesma forma, em comparação com o método CovnNet em cascata de três planos [31], nosso método pode segmentar com mais precisão o núcleo do tumor.
2.4 Comparação com os resultados de outros métodos
A Tabela 5 mostra a comparação com o método na literatura [23-27] (a validação é realizada no conjunto de dados MBTSC-2019 de acordo com as configurações originais). Como comparação, nenhum outro dado externo é usado durante o treinamento, e o melhor desempenho do método é sublinhado.

Pode-se ver na Tabela 5 que, além de melhorar o tumor, em comparação com outras redes em todo o tumor e no núcleo do tumor, o ConvNet integrado ideal proposto pode obter melhores resultados de segmentação. Razões para não escolher a rede principal com melhor desempenho como sub-rede: Em primeiro lugar, do ponto de vista da medicina clínica, a frequência de uso do núcleo do tumor e de todo o tumor e a importância no diagnóstico são maiores do que a do tumor aprimorado; em segundo lugar, da regra MBTSC Do ponto de vista, a importância de marcar pontos é núcleo do tumor> tumor inteiro> tumor aprimorado; finalmente, o foco do método do autor é verificar o método de integração anterior, então aqui " A navalha de Occam" também é totalmente aplicável sem ter que substituí-la por outras redes mais avançadas. Deste ponto de vista, através da integração ideal de 3D-ConvNet e 3D-U em forma de ConvNet, apresenta um futuro promissor na segmentação de imagens de tumores cerebrais.
Sua alta eficiência finalmente alcança melhor precisão de segmentação do que os excelentes métodos do mesmo período.
3 discussão
O autor propõe um método ensemble de 3D-ConvNet e 3D-U-ConvNet para segmentação de tumor cerebral em dados multimodais de ressonância magnética.A precisão da classificação competitiva é obtida no conjunto. O método atinge pontuações médias de dados de 0,750, 0,906 e 0,846 em termos de aumento do tumor, tumor total e núcleo do tumor, respectivamente, superando
muitos métodos atuais.
Embora o método tenha um bom desempenho em todas as categorias tumorais e centrais do tumor, a precisão da segmentação do tumor aumentado ainda precisa ser melhorada. Um esquema de limiar interessante foi implementado em [29], no qual se o tumor de realce for menor que o limiar definido, a área será substituída por tecido gangrenado, esta operação melhora significativamente a precisão da classificação de tumores de realce. No momento, o trabalho do autor ainda precisa ser aprimorado: em primeiro lugar, a integração de segmentação proposta é atualmente avaliada apenas no conjunto de verificação oficial do MBTSC-2019 e pode ser verificada posteriormente testando dados clínicos individuais de ressonância magnética. Em segundo lugar, o autor não realizou um pré-processamento extensivo no conjunto de dados e pós-processamento nos resultados. Como comparação, muitas literaturas propõem que o modelo seja normalizado pela intensidade
[32] e correção de viés [33] para minimizar a variabilidade de seus dados. Os mesmos métodos de pós-processamento, como o uso de campos aleatórios condicionais [34], também demonstraram melhorar a precisão da segmentação. Embora ainda existam os aspectos acima que precisam ser complementados e aprimorados, o método de conjunto ótimo proposto pelo autor ainda mostra precisão de segmentação tumoral eficiente e robusta em regiões multimodais. No trabalho futuro, além dos aspectos mencionados acima, o autor planeja adicionar alguns dados clínicos à integração e ajustar ainda mais os hiperparâmetros para melhorar o efeito do modelo.
4. Conclusão
A segmentação automática e precisa de tumores cerebrais a partir de imagens de ressonância magnética multimodal é muito importante para o diagnóstico e tratamento de cânceres cerebrais relacionados. O autor propõe um método ConvNet de conjunto ideal para integrar o ConvNet 3D que tem bom desempenho no MBTSC-2018 com o mapa de segmentação da rede ConvNet em forma de U 3D que também tem bom desempenho na segmentação médica. Especificamente, os dois modelos são treinados no conjunto de dados multimodal usado no MBTSC-2019 e diferentes caminhos de segmentação são executados analisando seu desempenho diferencial. Por fim, o método do conjunto ótimo é verificado no conjunto de validação e os resultados mostram que o método proposto pelo autor obteve melhor acurácia do que outros métodos no mesmo período em imagens tumorais multimodais.
5 Referências
um pouco.
Os alunos interessados podem ir a Zhiwang para fazer o download deste artigo.
Fonte do artigo: Han Bing, Wang Peng, Zhou Yi. Método de segmentação de imagens de câncer cerebral baseado em conjunto otimizado ConvNet [J]. Journal of Anhui University (Natural Science Edition), 2022,46(06)