Pratique d'apprentissage en profondeur (10) : Utilisation de PyTorch pour la segmentation d'images médicales en 3D
Article MedicalZoo : Apprentissage profond dans l'analyse d'images médicales : une analyse comparative de la segmentation multimodale de l'IRM cérébrale avec
le code des réseaux de neurones profonds 3D a été open source :MédicalZooPytorche
Plus d'informations sur Ai: Princesse AiCharm

1. Présentation du projet
L'essor des réseaux profonds dans la vision par ordinateur a fourni des solutions de pointe aux problèmes où les techniques de traitement d'image classiques sont moins performantes. On peut facilement prétendre que les DNN (Deep Neural Networks) ont atteint des performances supérieures dans les tâches de reconnaissance d'images généralisées, y compris des problèmes tels que la détection d'objets, la classification et la segmentation des images, la reconnaissance d'activité, le flux optique et l'estimation de la pose.
Parallèlement à l'essor de la vision par ordinateur, il y a eu un vif intérêt pour son application dans le domaine de l'imagerie médicale. Bien que les données d'imagerie médicale ne soient pas si facilement disponibles, les DNN semblent être des candidats idéaux pour modéliser ces données complexes de grande dimension.
Récemment, l'Imperial College de Londres a lancé un cours sur le COVID-19. De nombreuses études ont tenté de détecter automatiquement le COVID-19 via des réseaux profonds de tomodensitogrammes 3D. Malgré cela, les données d'application spécifiques ne sont toujours pas disponibles. Il est clair que l'intelligence artificielle aura un impact énorme sur le développement de la médecine par l'imagerie médicale.
Comme je le verrai, les images médicales sont généralement en trois ou quatre dimensions. Une autre raison pour laquelle ce domaine attire beaucoup d'attention est son impact direct sur la vie humaine. Aux États-Unis, la faute professionnelle médicale est la troisième cause de décès après les maladies cardiaques et le cancer. Par conséquent, il est clair que les trois principales causes de décès chez l'homme sont toutes liées à l'imagerie médicale. C'est pourquoi on estime que d'ici 2023, l'intelligence artificielle et l'apprentissage en profondeur créeront un tout nouveau marché de plus d'un milliard de dollars dans l'imagerie médicale.
Ce travail se situe à l'intersection de ces deux mondes : les réseaux de neurones profonds et l'imagerie médicale. Dans cet article, j'aborderai le problème de la segmentation des images médicales, en mettant l'accent sur les images par résonance magnétique, qui est l'une des tâches les plus populaires car c'est celle pour laquelle l'ensemble de données le mieux structuré auquel tout le monde a accès. Étant donné que la collecte de données médicales en ligne n'est pas aussi simple qu'il y paraît, une collection de liens est fournie à la fin de l'article pour commencer votre voyage.
Cet article présente quelques résultats préliminaires d'une bibliothèque open-source en cours de développement appelée MedicalZoo , qui peut être trouvée ici.
2. Exigences pour la segmentation d'images médicales 3D
La segmentation d'images volumétriques 3D dans les images médicales est obligatoire pour le diagnostic, la surveillance et la planification du traitement. Je n'utiliserai que des images par résonance magnétique (IRM). Les procédures manuelles nécessitent une connaissance de l'anatomie, et elles sont coûteuses et prennent du temps. En outre, ils peuvent être inexacts en raison de facteurs humains. Cependant, la segmentation automatique du volume peut faire gagner du temps aux médecins et fournir des solutions précises et reproductibles pour une analyse plus approfondie.
Je commencerai par décrire les principes fondamentaux de l'imagerie par résonance magnétique, car la compréhension de vos données d'entrée est essentielle à la formation d'une architecture approfondie. Ensuite, le lecteur reçoit un aperçu de 3D-UNET qui peut être utilisé efficacement pour cette tâche.
3. Images médicales et IRM
L'imagerie médicale tente de révéler les structures internes cachées par la peau et les os, ainsi que de diagnostiquer et de traiter les maladies. L'imagerie médicale par résonance magnétique (RM) utilise les signaux des noyaux d'hydrogène pour créer des images. Dans le cas du noyau d'hydrogène : lorsqu'il est exposé à un champ magnétique externe, noté B0, le moment magnétique, ou spin, s'aligne avec la direction du champ magnétique comme l'aiguille d'une boussole.
Toute magnétisation constante est tournée vers l'autre plan par une impulsion radiofréquence supplémentaire suffisamment forte et appliquée suffisamment longtemps pour incliner la magnétisation. Immédiatement après l'excitation, l'aimantation tourne dans l'autre plan. L'aimantation tournante génère le signal MR dans la bobine réceptrice. Cependant, le signal MR s'estompe rapidement en raison de deux processus distincts qui réduisent l'aimantation, ce qui conduit à un retour à l'état stable de pré-excitation, entraînant l'image dite T1 et l'image T2 MR. La relaxation T1 est liée à l'excès d'énergie du nucléon par rapport à son environnement, tandis que la relaxation T2 fait référence au phénomène dans lequel les vecteurs d'aimantation individuels commencent à s'annuler. Les phénomènes ci-dessus sont complètement indépendants. Par conséquent, différentes intensités représentent différents tissus, comme le montre la figure ci-dessous.

4. Représentation d'images médicales 3D
Étant donné que les images médicales représentent des structures 3D, elles peuvent être traitées en utilisant des tranches de volumes 3D et en effectuant des convolutions glissantes 2D conventionnelles, comme le montre la figure ci-dessous. Nous supposons que le rectangle rouge est un patch d'image 5x5, qui peut être représenté par une matrice contenant des valeurs d'intensité. Les intensités de voxel et les noyaux sont convolués avec un noyau de convolution 3x3, comme indiqué dans la figure ci-dessous. Dans le même schéma, le noyau glisse sur toute la grille 2D (tranches d'images médicales), et à chaque fois nous faisons une corrélation croisée. Le résultat d'un patch convolutif 5x5 est stocké dans une matrice 3x3 (sans remplissage pour l'illustration) et propagé à travers la couche suivante du réseau. 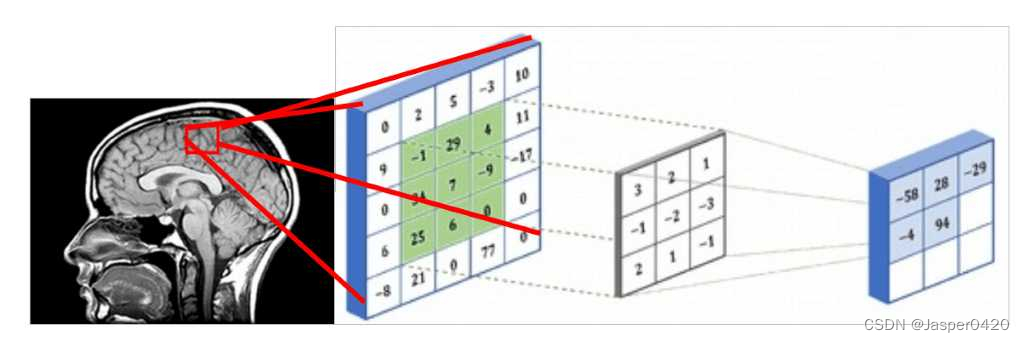 Alternativement, vous pouvez les représenter comme des sorties similaires aux couches intermédiaires. Dans les architectures profondes, nous avons généralement plusieurs cartes de caractéristiques, qui sont en fait un tenseur 3D. S'il y a des raisons de croire qu'il existe un motif dans la dimension supplémentaire, alors effectuer une convolution glissante 3D est le meilleur choix. C'est le cas des images médicales. Semblable à la convolution bidimensionnelle, la convolution bidimensionnelle code la relation spatiale des objets dans le champ bidimensionnel, et la convolution tridimensionnelle peut décrire la relation spatiale des objets dans l'espace tridimensionnel. Les représentations 2D étant sous-optimales pour les images médicales, nous choisirons d'utiliser des réseaux convolutifs 3D dans cet article.
Alternativement, vous pouvez les représenter comme des sorties similaires aux couches intermédiaires. Dans les architectures profondes, nous avons généralement plusieurs cartes de caractéristiques, qui sont en fait un tenseur 3D. S'il y a des raisons de croire qu'il existe un motif dans la dimension supplémentaire, alors effectuer une convolution glissante 3D est le meilleur choix. C'est le cas des images médicales. Semblable à la convolution bidimensionnelle, la convolution bidimensionnelle code la relation spatiale des objets dans le champ bidimensionnel, et la convolution tridimensionnelle peut décrire la relation spatiale des objets dans l'espace tridimensionnel. Les représentations 2D étant sous-optimales pour les images médicales, nous choisirons d'utiliser des réseaux convolutifs 3D dans cet article.
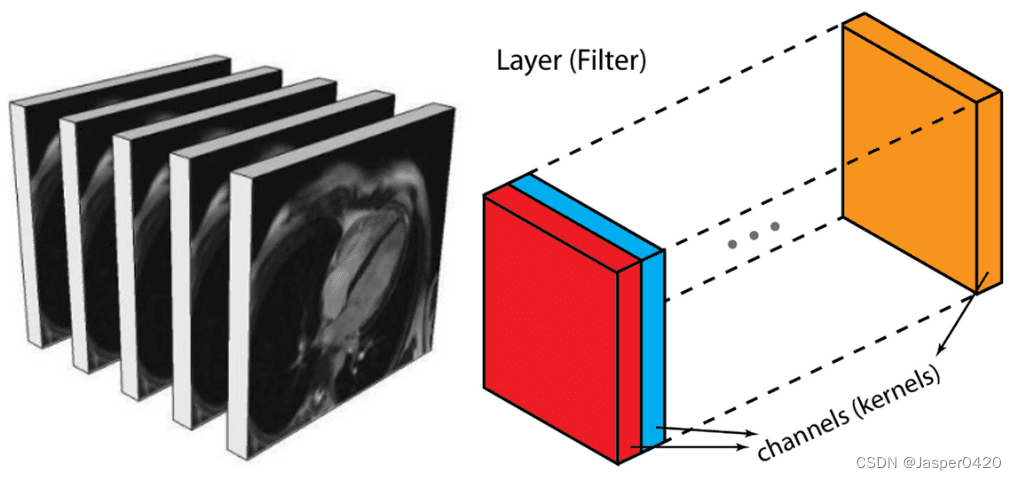
Les tranches d'images médicales peuvent être considérées comme des cartes de caractéristiques multiples dans une couche intermédiaire, à la différence qu'elles ont de fortes relations spatiales.
5. Modèle 3D-Unet
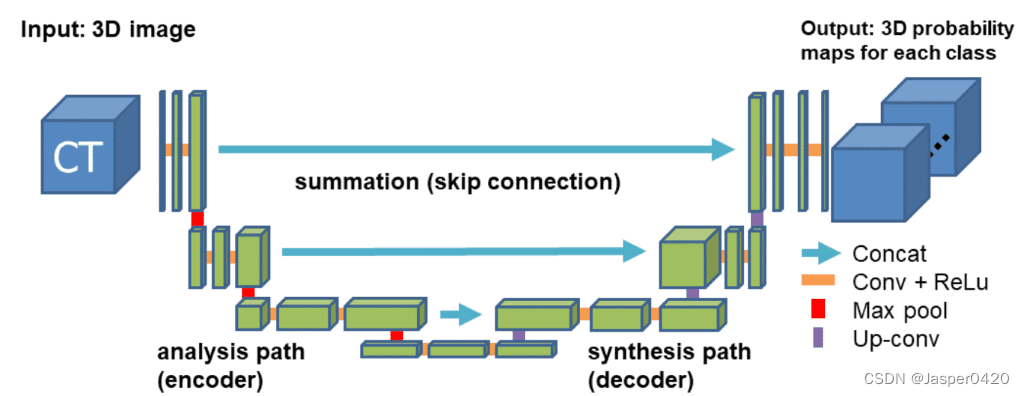
Dans notre exemple, nous utiliserons le réseau en forme de U 3D accepté. Ce dernier (code) étend l'idée continue de réseaux Unet 2D symétriques en forme de U, donnant des résultats impressionnants dans les tâches liées au RVB telles que la segmentation sémantique. Le modèle a un encodeur (chemin de rétrécissement) et un décodeur (chemin de synthèse), chacun avec quatre étapes d'analyse. Dans le chemin du codeur, chaque couche est constituée de deux convolutions 3 × 3 × 3, chacune suivie d'une unité linéaire redressée (ReLu), suivie d'un ensemble maximal 2 × 2 × 2, chaque dimension La taille du pas est de 2. Dans le chemin de décodage, chaque couche est constituée d'une convolution transposée 2×2×2 avec une foulée de 2 dans chaque dimension, suivie de deux convolutions 3×3×3, chacune suivie d'un ReLu . Les connexions de saut de raccourci des couches à résolution égale dans le chemin d'analyse fournissent des fonctionnalités haute résolution essentielles pour le chemin de synthèse. Dans la dernière couche, une convolution 1×1×1 réduit le nombre de canaux de sortie au nombre d'étiquettes. En doublant le nombre de canaux avant la mise en commun maximale, le problème de goulot d'étranglement est évité. Avant chaque ReLU, une normalisation de lot 3D est introduite. Pendant la formation, la moyenne et l'écart type de chaque lot sont normalisés et les statistiques globales sont mises à jour avec ces valeurs. Vient ensuite une couche qui apprend explicitement l'échelle et le biais. La figure ci-dessous illustre la structure du réseau.
5.1 Fonction de perte : perte de dés
En raison du déséquilibre inhérent à la tâche, l'entropie croisée ne peut pas toujours fournir une bonne solution pour cette tâche. Plus précisément, la perte d'entropie croisée examine chaque pixel individuellement, en comparant la prédiction de classe (vecteur de pixel dans la direction de la profondeur) à notre vecteur cible codé à chaud. Étant donné que la perte d'entropie croisée évalue la prédiction de classe pour chaque vecteur de pixel individuellement, puis fait la moyenne sur tous les pixels, nous affirmons essentiellement que chaque pixel de l'image est appris de manière égale. Cela peut être un problème si vous avez une représentation déséquilibrée de vos différentes classes dans vos images, car les classes les plus répandues peuvent dominer la formation.
Les 4 classes que nous essaierons de distinguer dans l'IRM cérébrale ont des fréquences différentes dans les images (c'est-à-dire que l'air a beaucoup plus d'instances que les autres tissus). C'est pourquoi l'indicateur de perte de dés est utilisé. Il est basé sur le coefficient de dé, qui est essentiellement une mesure du chevauchement entre deux échantillons. Cette mesure va de 0 à 1, où un coefficient Dice de 1 indique un chevauchement parfait et complet. La perte de dés a été développée à l'origine pour la classification binaire, mais elle peut être généralisée au travail multiclasse. N'hésitez pas à utiliser notre implémentation multi-classes de la perte de dés.
5.2 Données d'imagerie médicale
Les architectures profondes nécessitent un grand nombre d'échantillons d'apprentissage avant de pouvoir produire des représentations de généralisation utiles, et les données d'apprentissage étiquetées sont souvent coûteuses et difficiles à produire. C'est pourquoi nous voyons chaque jour de plus en plus de données d'imagerie médicale être produites à l'aide de nouvelles techniques d'apprentissage génératif. De plus, les données de formation doivent être représentatives de ce que le réseau rencontrera à l'avenir. Si les échantillons d'apprentissage proviennent d'une distribution de données différente de celle rencontrée dans le monde réel, les performances de généralisation du réseau seront inférieures à celles attendues.
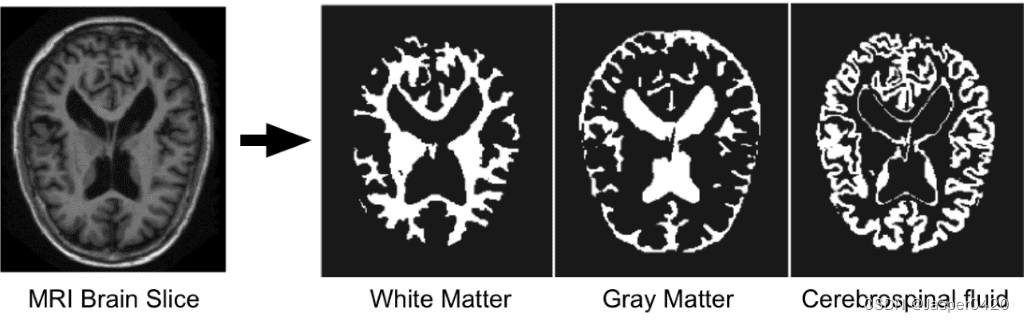
Puisque nous nous concentrons sur la segmentation automatique de l'IRM cérébrale, il est nécessaire d'introduire brièvement les structures de base du cerveau que les DNN tentent de distinguer : a) la matière blanche (WM), b) la matière grise (GM), c) le liquide céphalo-rachidien (CSF ). La figure ci-dessous illustre un tissu segmenté dans une tranche d'IRM cérébrale.
5.2.1 Défi des données d'imagerie médicale I-Seg 2017
La segmentation précise des images IRM du cerveau du nourrisson en matière blanche (WM), matière grise (GM) et liquide céphalo-rachidien (LCR) au cours de cette période critique est fondamentale pour étudier le développement normal et anormal du cerveau précoce. La première année de vie est la période la plus active du développement postnatal du cerveau humain, avec une croissance rapide des tissus et le développement de diverses fonctions cognitives et motrices. Ce stade précoce est essentiel dans de nombreux troubles neurodéveloppementaux et neuropsychiatriques, tels que la schizophrénie et l'autisme. De plus en plus d'attention est portée à cette période critique.
Le but de cet ensemble de données est de faciliter les algorithmes de segmentation automatique pour les IRM cérébrales IRM des nourrissons de 6 mois. Ce défi a eu lieu en même temps que MICCAI 2017, et un total de 21 équipes internationales y ont participé. L'ensemble de données contient 10 images densément annotées par des experts et 13 images d'imagerie pour les tests. Aucune balise de test n'est fournie et vous ne pouvez voir votre score qu'après avoir téléchargé les résultats sur le site officiel. Chaque sujet a une image pondérée T1 et pondérée T2.
Le premier sujet sera utilisé pour les tests. La taille du volume MR d'origine est de 256x192x144. Dans 3D-Unet, la taille du sous-volume d'échantillon utilisé est de 128x128x64. L'ensemble de données de formation résultant se compose de 500 sous-volumes. Pour l'ensemble de validation, 10 échantillons aléatoires d'un sujet ont été utilisés.
Téléchargement du jeu de données
6. Zoo médical
Notre objectif est d'implémenter dans PyTorch une bibliothèque de segmentation d'images médicales open source composée de réseaux de neurones profonds 3D de pointe et de chargeurs de données pour les ensembles de données médicales les plus courants. La première version stable de notre référentiel devrait être publiée prochainement.
Nous croyons fermement à la recherche en apprentissage profond ouverte et reproductible. Pour reproduire nos résultats, le code et les matériaux de ce travail peuvent être trouvés dans ce référentiel. Ce projet a commencé comme une thèse de maîtrise et est actuellement en cours de développement.
6.1 Détails de mise en œuvre
Nous utilisons le framework PyTorch, qui est considéré comme l'outil de recherche d'apprentissage en profondeur le plus largement accepté. Toutes les expériences utilisent une descente de gradient stochastique avec une seule taille de lot, un taux d'apprentissage de 1e-3 et une décroissance de poids de 1e-8. Nous fournissons des tests dans le référentiel où vous pouvez facilement reproduire nos résultats afin que vous puissiez utiliser le code, les modèles et les chargeurs de données.
Récemment, nous avons augmenté les capacités de visualisation de Tensorboard avec Pytorch. Cette fonctionnalité étonnante maintient votre santé mentale en place et vous permet de suivre le processus d'entraînement de votre modèle. Ci-dessous, vous pouvez voir un exemple qui conserve les statistiques d'entraînement, le coefficient et la perte des dés et les scores par classe pour avoir une idée du comportement du modèle.
6.2 Code
Assemblons tous les modules décrits et mettons en place une expérience avec un court script (pour illustration) de MedicalZoo.
# Python libraries
import argparse
import os
# Lib files
import lib.medloaders as medical_loaders
import lib.medzoo as medzoo
import lib.train as train
import lib.utils as utils
from lib.losses3D import DiceLoss
def main():
args = get_arguments()
utils.make_dirs(args.save)
training_generator, val_generator, full_volume, affine = medical_loaders.generate_datasets(args,
path='.././datasets')
model, optimizer = medzoo.create_model(args)
criterion = DiceLoss(classes=args.classes)
if args.cuda:
model = model.cuda()
print("Model transferred in GPU.....")
trainer = train.Trainer(args, model, criterion, optimizer, train_data_loader=training_generator,
valid_data_loader=val_generator, lr_scheduler=None)
print("START TRAINING...")
trainer.training()
def get_arguments():
parser = argparse.ArgumentParser()
parser.add_argument('--batchSz', type=int, default=4)
parser.add_argument('--dataset_name', type=str, default="iseg2017")
parser.add_argument('--dim', nargs="+", type=int, default=(64, 64, 64))
parser.add_argument('--nEpochs', type=int, default=200)
parser.add_argument('--classes', type=int, default=4)
parser.add_argument('--samples_train', type=int, default=1024)
parser.add_argument('--samples_val', type=int, default=128)
parser.add_argument('--inChannels', type=int, default=2)
parser.add_argument('--inModalities', type=int, default=2)
parser.add_argument('--threshold', default=0.1, type=float)
parser.add_argument('--terminal_show_freq', default=50)
parser.add_argument('--augmentation', action='store_true', default=False)
parser.add_argument('--normalization', default='full_volume_mean', type=str,
help='Tensor normalization: options ,max_min,',
choices=('max_min', 'full_volume_mean', 'brats', 'max', 'mean'))
parser.add_argument('--split', default=0.8, type=float, help='Select percentage of training data(default: 0.8)')
parser.add_argument('--lr', default=1e-2, type=float,
help='learning rate (default: 1e-3)')
parser.add_argument('--cuda', action='store_true', default=True)
parser.add_argument('--loadData', default=True)
parser.add_argument('--resume', default='', type=str, metavar='PATH',
help='path to latest checkpoint (default: none)')
parser.add_argument('--model', type=str, default='VNET',
choices=('VNET', 'VNET2', 'UNET3D', 'DENSENET1', 'DENSENET2', 'DENSENET3', 'HYPERDENSENET'))
parser.add_argument('--opt', type=str, default='sgd',
choices=('sgd', 'adam', 'rmsprop'))
parser.add_argument('--log_dir', type=str,
default='../runs/')
args = parser.parse_args()
args.save = '../saved_models/' + args.model + '_checkpoints/' + args.model + '_{}_{}_'.format(
utils.datestr(), args.dataset_name)
return args
if __name__ == '__main__':
main()
6.3 Résultats expérimentaux
Ci-dessous, vous pouvez voir les courbes Dice LOSS d'entraînement et de validation pour le modèle. Il est important de surveiller les performances de votre modèle et d'ajuster les paramètres pour obtenir une courbe d'entraînement aussi fluide. Il est facile de comprendre l'efficacité de ce modèle.
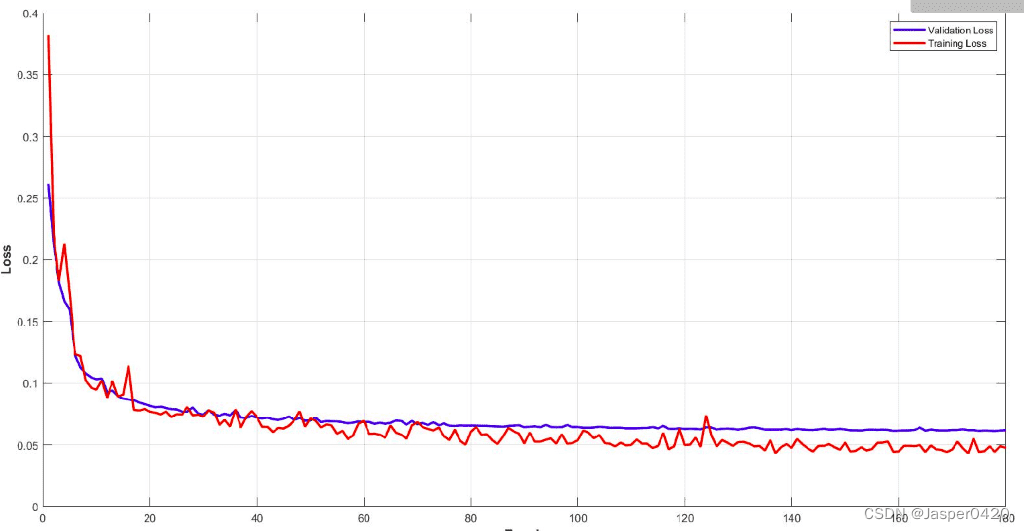
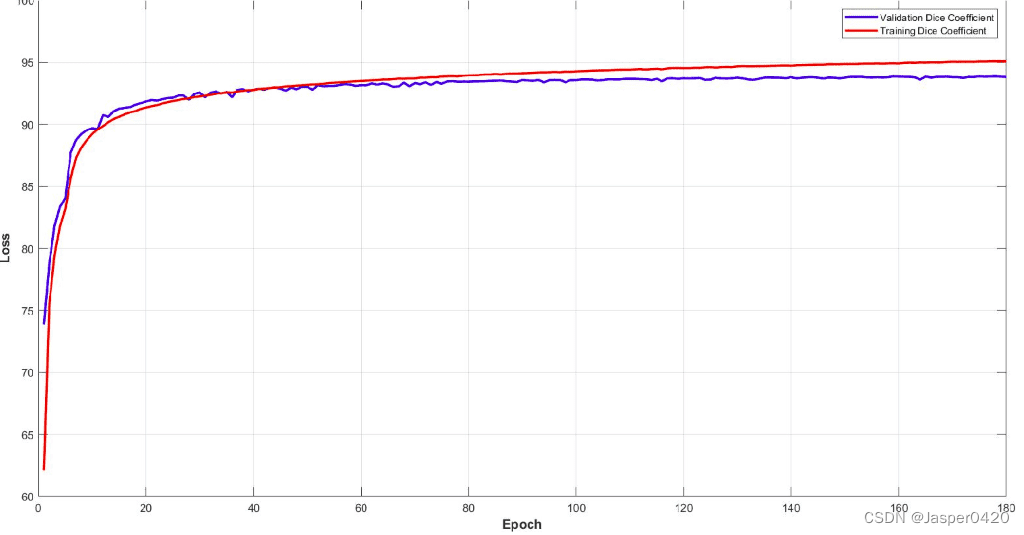
Étonnamment, le modèle atteint environ 93 % des scores de coefficient de dés sur l'ensemble de validation du sous-volume. Enfin, voyons quelques prédictions visuelles de 3D-Unet sur l'ensemble de validation. Nous ne montrons ici qu'une tranche représentative, malgré la prévision d'un volume 3D. En extrayant plusieurs sous-volumes de l'IRM, nous pouvons les combiner pour former une segmentation IRM 3D complète. Notez que le fait que nous utilisons un échantillonnage en sous-volume est une augmentation des données.

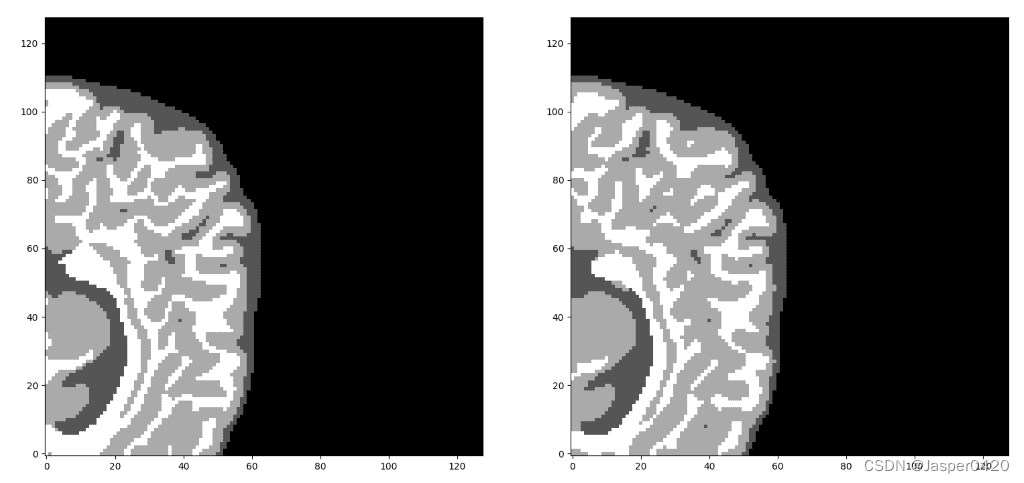
Préactivation de la dernière couche non normalisée à partir de 3D-Unet formé. Le réseau apprend un contenu hautement sémantique lié aux tâches, correspondant à des structures cérébrales similaires à l'entrée.
Nos prédictions contre la vérité terrain. Selon vous, quelle prédiction est la vérité fondamentale ? Avant de vous décider, regardez de plus près. Il convient de noter que nous n'avons montré ici que des tranches médianes, mais la projection est un volume 3D. On peut observer que le réseau prédit parfaitement les voxels d'air, alors qu'il a du mal à distinguer les frontières tissulaires. Mais vérifions une deuxième fois et découvrons ce qui est réel !
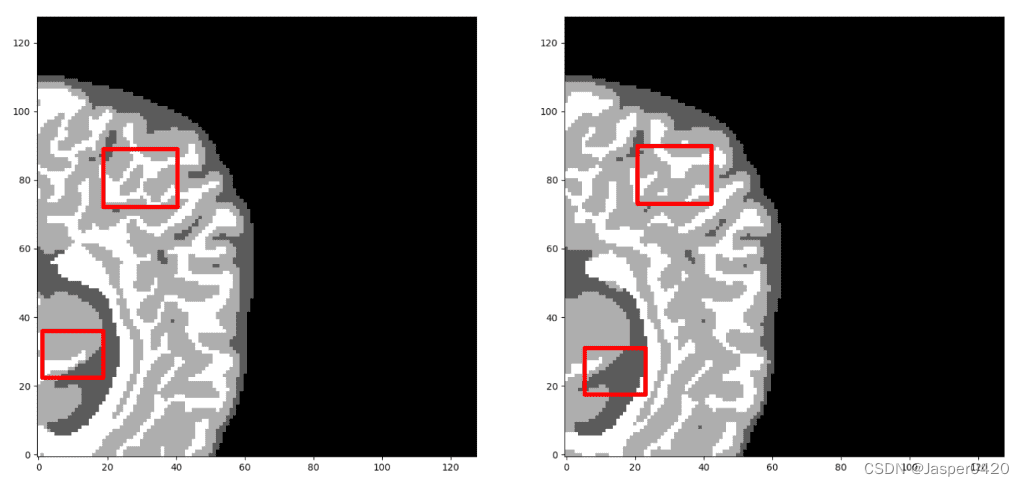
Maintenant, je suis sûr que vous pouvez discerner la vérité à partir du sol. Si vous n'êtes pas sûr, veuillez consulter la fin de l'article
Récemment, nous avons également ajouté les capacités de visualisation Tensorboard de Pytorch. Cette fonctionnalité étonnante maintient votre santé mentale en place et vous permet de suivre le processus d'entraînement de votre modèle. Ci-dessous, vous pouvez voir un exemple de conservation des statistiques d'entraînement, des coefficients et des pertes de dés et des scores par classe pour avoir une idée du comportement du modèle.
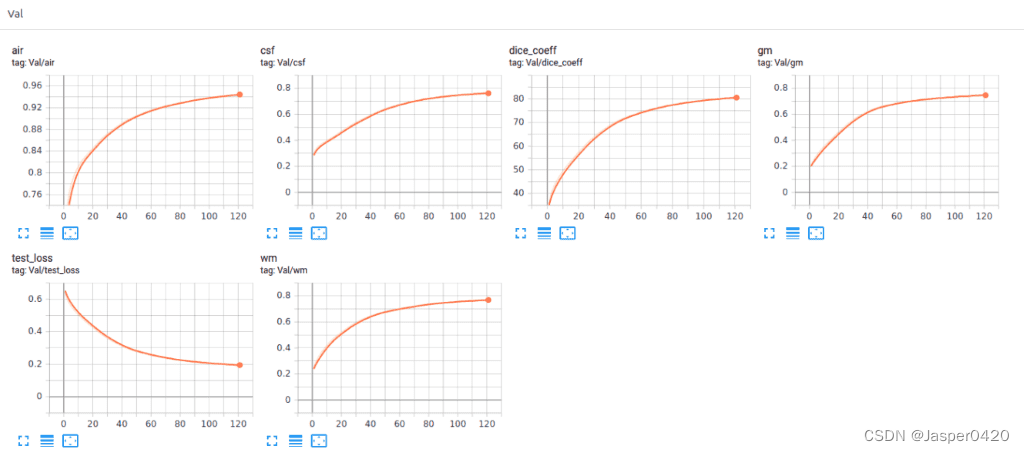
Il est clair que différents tissus ont des précisions différentes, même dès le début de l'entraînement. Par exemple, en regardant les voxels d'air dans l'ensemble de validation, ils commencent par des valeurs élevées car c'est la classe la plus dominante dans l'ensemble de données déséquilibré. La matière grise, en revanche, part de la valeur la plus basse car elle est la plus difficile à distinguer et a moins d'exemples d'entraînement.
7. Résumé
Cet article illustre partiellement certaines fonctionnalités de la bibliothèque MedicalZoo Pytorch. Les modèles d'apprentissage en profondeur fourniront à la société des solutions d'imagerie médicale immersives.
Dans cet article, les concepts de base de l'imagerie médicale et de l'IRM sont passés en revue, ainsi que la manière dont ils sont représentés et utilisés dans les architectures d'apprentissage en profondeur. Ensuite, une architecture 3D efficace et largement acceptée (Unet) et une fonction de perte de dés pour gérer le déséquilibre des classes sont décrites. Enfin, les résultats préliminaires de notre analyse expérimentale en IRM cérébrale sont présentés en combinant toutes les fonctionnalités décrites ci-dessus et en utilisant des scripts de bibliothèque. Ces résultats démontrent l'efficacité des architectures 3D et le potentiel du deep learning dans l'analyse d'images médicales.
Plus d'informations sur Ai: Princesse AiCharm
