
Depois de baixar os dados do banco de dados do SEER, algumas pessoas se sentirão perdidas, pois os dados não foram limpos e classificados, não podem ser transformados na forma reconhecida por nosso software estatístico e não podem ser analisados. Hoje vamos ensinar como limpar o banco de dados seed usando a linguagem R, para que os dados possam ser usados por nós.
Primeiro, importe o pacote R de que precisamos, o que requer estrangeiro, carro e stringr, que deve ser baixado primeiro.

Em seguida, importamos os dados baixados para o R. Existem mais de 200.000 dados, que é impossível modificar manualmente.
be<-read.csv("E:/r/test/seer4.csv",sep=',',header=TRUE)

Observe o nome e a forma dos dados e a variável
head (be)
names (be)


parece muito confusa, alguns nomes são muito longos, mude os nomes todos
colnames(be)<-c("sex","time","rezult","rezult1","status","race","Subtype","nodes","Lymph.Invasion",
"tumor.size","extension","lymph.nodes","age","ajcc")#数据太长,重新命名
Vamos verificar o conjunto de dados novamente. Desta vez, é muito mais refrescante.

Descobrimos que há 14 variáveis no total, entre as quais Lymph.Invasion tem dados ausentes, que não podem ser analisados de todo e só podem ser excluídos. Essa é a frustração de bases de dados públicas.
be<-be[,-9]#删掉第9列Lymph.Invasion,因为都是缺失的数据

Muitas variáveis nos dados são strings, que não atendem aos requisitos, temos que transformá-las em números
be$sex<-ifelse(be$sex=="Female",1,ifelse(be$sex=="Male",2,NA))#性别转换成1和2,缺失的使用NA表示,其他的相同
be$rezult1 <-ifelse(be$rezult1 =="Alive or dead due to cancer",1,
ifelse(be$rezult1 =="Dead (attributable to causes other than this cancer dx)",
2,NA))
be$status<-ifelse(be$status=="Alive",0,ifelse(be$status=="Dead",1,NA))
be$race<-ifelse(be$race=="White",1,ifelse(be$race=="Black",2,3))
be$Subtype<-recode(be$Subtype,"'HR-/HER2- (Triple Negative)'=1;
'HR-/HER2+ (HER2 enriched)'=2;'HR+/HER2- (Luminal A)'=3;
'HR+/HER2+ (Luminal B)'=4;else=NA")#这里是4个分类变量,使用ifelse函数套叠胎麻烦,改用car函数
be$nodes[be$nodes=="Blank(s)"]=NA#让数据中的Blank(s)变为缺失值,下面同理
be$tumor.size[be$tumor.size=="Blank(s)"]=NA
be$extension[be$extension=="Blank(s)"]=NA
be$lymph.nodes[be$lymph.nodes=="Blank(s)"]=NA
be$age<-str_extract(be$age, "\\d+")#把年龄里面的数字提取出来
be$ajcc[be$ajcc=="Blank(s)"]=NA

OK, a conversão está quase concluída. Vamos dar uma olhada. Rezult é inútil. Nós o ignoramos e o excluímos mais tarde. O que precisamos é rezult1.
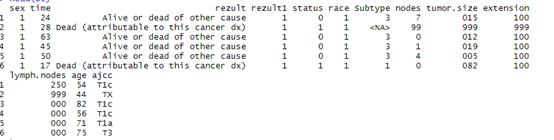
Ajcc. Não convertemos porque não precisamos usá-lo ainda. Quando falamos sobre a exploração dos efeitos de interação Vamos falar sobre isso durante a análise. Agora ignore-o primeiro. Se você tem transtorno obsessivo-compulsivo, também pode convertê-lo de acordo com nosso código acima.
OK, já está feito? Não, há outra variável importante que não foi gerada, que é o resultado do risco competitivo.
Vamos gerá-la agora.
be$status1<-ifelse(be$status==0,0,ifelse(be$rezult1==1,1,2))
Finalmente os dados saem,
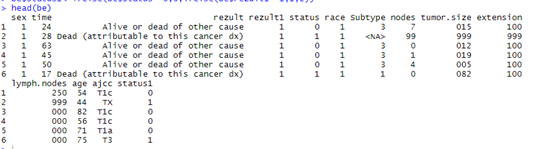
imprima-os como 1.csv
write.csv(be,file = "1.csv")
Por fim, abra 1.csv e resolva-os. Estes são os dados que queremos publicar.

Mais de 200.000 dados. Enviar um SCI básico ou de pontuação baixa em chinês não é fácil, como jogar.
Se você quiser saber mais sobre o processo de mineração de dados, preste atenção aos meus tutoriais de pesquisa científica. Para
artigos mais interessantes, preste atenção à conta pública: pesquisa baseada em zero
