医用画像セグメンテーション システムのレビュー 医用画像における人工知能のためのデータ準備: 包括的なガイド ...
開発
2023-05-08 07:57:30
訪問数: null
- 医用画像における人工知能のためのデータ準備: オープンアクセスのプラットフォームとツールの包括的なガイド
- データ準備プロセス
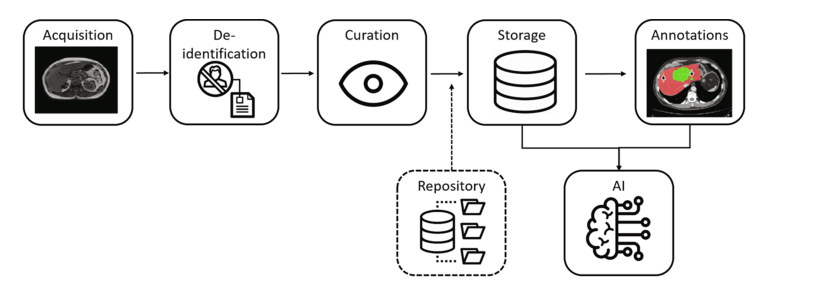
(i) 臨床現場での画像取得、(ii) 個人情報を削除して患者のプライバシーを保護するための画像の匿名化、(iii) 画像および非画像情報の品質を制御するためのデータ管理、(iv) 画像の保存と管理、最後に (v) 画像注釈。
- FAIR データの基本原則: 検索可能、アクセス可能、相互運用可能、再利用可能
- データ分類: 保護された健康情報 (PHI) および患者の健康情報に関連付けられた個人を特定できる情報 (PII)
PII: 電子健康記録 (EHR)、医療画像、臨床および生物学的データ、および医療サービスからのデータを含むことができる 収集されたその他のデータプロバイダによる
- データ形式:DICOM。メタデータと呼ばれることが多い画像には、画像シーケンス、病院、プロバイダー、臨床医、または患者の情報などに関する情報を含むヘッダーが含まれています。
- 匿名化ツール: FreeSurfer の mri_deface、pydeface、mridefacer、Quickshear の最初の 3 つは
Python ライブラリです。
- データ匿名化ツールの要件リスト: 未使用の識別情報を削除し、状況に応じて保持する; データの二次送信を回避するための臨床現場でのトレーニング
- ISO 25237: 既存のプライバシー保護の採用基準
- 詳細リスト
-


- コンテンツ管理ツール: フォーマット変換とデータ テスト トレーニング セット管理を実行できます
-

- GUI: Posda、DVTk、dcm4che、BrainVoyager、LONI Debabeler、dcm2niix の GUI は MRIcroGL
Posda、DVTk、および dcm4che は web\GUI インターフェイスを提供します
- 画像保管
-
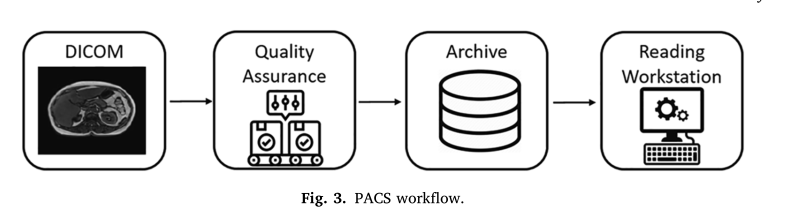
- PACS: マルチモーダル医療画像 (MRI、コンピューター断層撮影 CT、陽電子放出断層撮影 PET など) を保存します。複数のデバイスや場所から簡単にアクセスできます。(複数のバックアップ
品質保証 (QA) ワークステーション: 患者の人口統計および研究のその他の重要な属性を検証します。 アーカイブ (中央ストレージ デバイス): 検証済みの画像を、レポート、測定値、およびその他の関連情報と共に保存します。 読み取りワークステーション: 放射線科医がデータを確認する場所そして診断書を作成します。
- 医療自動化システム インターフェイス: 病院情報システム (HIS)、電子カルテ (EMR)、および放射線情報システム (RIS)
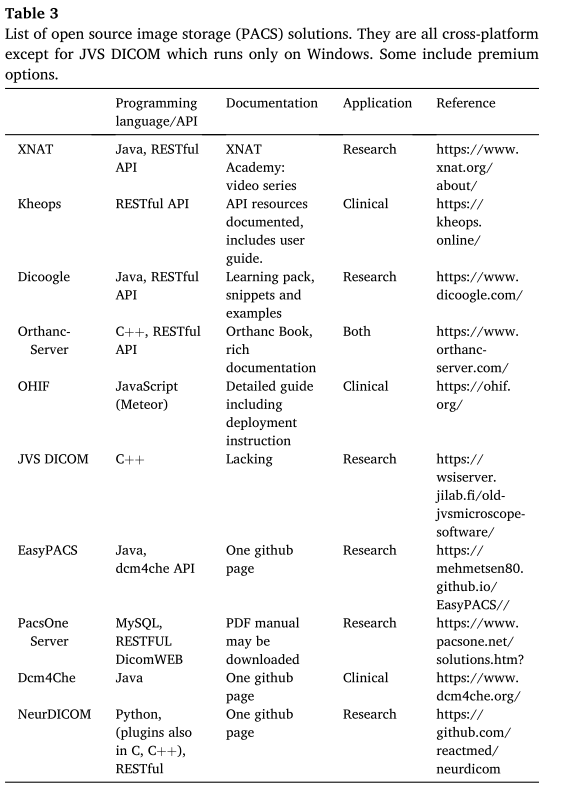
- オープンソース ソリューションの例
Dcm4che ( https://www.dcm4che.org )
Kheops (ホーム - KHEOPS )
Extensible Neuroimaging Archive Toolkit (XNAT) XNAT - ホーム
-
- XNAT: Java Web、Postgres データベースでの検索、XML ベースのデータ モデル、あらゆる種類の表形式データをサポートできます
- Dicoogle: オープンソース PACS アーカイブ、データベースなし
- 画像注釈ツール: 3D スライサー
-

- ITK-SNAP: 直交する 3 つのスライス (軸方向、冠状方向、矢状方向) のすべてに対してラベリングを実行し、3D レンダリングとして表示できます。
- MITK: アノテーション サーバーを支援し、自動セグメンテーションを実行する機能
- Horos Viewer: OsiriXTM およびその他のオープンソースの医用画像ライブラリに基づく
- プラグイン:
- Bio-Formats: 独自の顕微鏡画像データとメタデータを標準化されたオープン フォーマットに変換
- SciView: 画像とメッシュの 3D ビジュアライゼーションとバーチャル リアリティ機能
- MaMuT: 大量の画像データを閲覧、注釈付け、管理するための注釈
- トレーニング可能な Weka セグメンテーション: ピクセルベースのセグメンテーションを作成します
- クラウドキュアと CMRAD プラットフォーム: クラウド
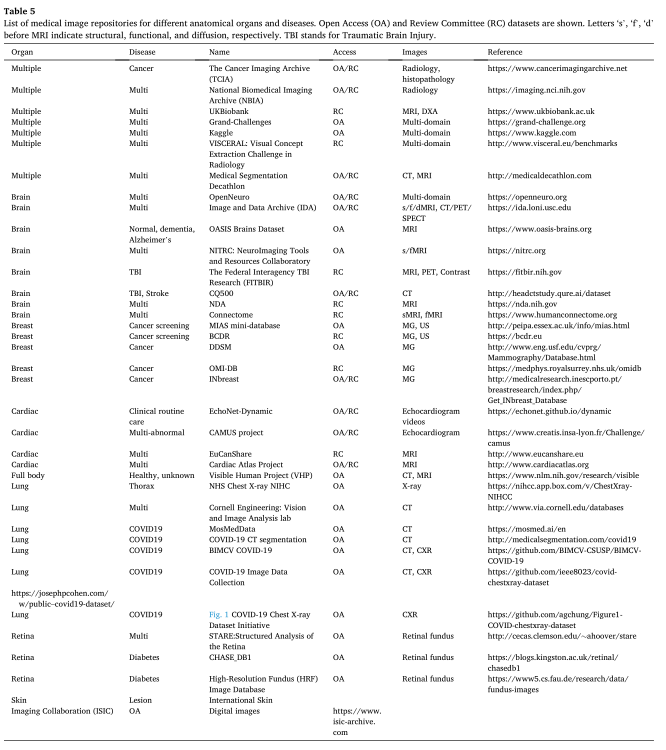
- 医用画像リポジトリ
- 対象臓器・疾患別オープンアクセス医用画像データベースの各種ソース
-
- UK Biobank:
電子医療記録などの臨床データ。脳、心臓、腹部、骨、頸動脈のスキャンを含む、100,000 人を超える参加者の画像コレクションがあります。
- ニューロイメージング データセット: IDA、OASIS、NITRC、および CQ500
-
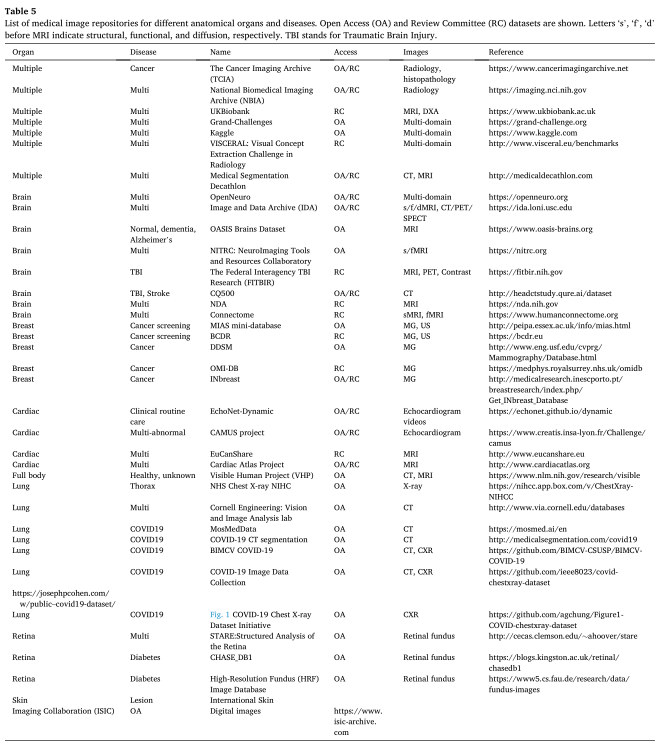
- 網膜眼底画像セグメンテーション: STARE、DRIVE (Grand-Challenges の一部)、および HRF
- 皮膚病変のデジタル画像の収集: International Skin Imaging Collaboration (ISIC)
- 乳がん検診マンモグラム:OPTIMAM(OMI-DB)
- 心エコー検査のビデオ: EchoNet- Dynamic
- 一般的な胸部疾患の注釈付き X 線: NHS 胸部 X 線
- 診断用肺 CT 画像: コーネル エンジニアリング: 視覚および画像解析ラボ リポジトリ
- covid - 19:BIMCV COVID19 , COVID-19 画像データ集
- 課題:あります
- AI 開発の方向性: (i) データの強化と合成、(ii) 連合学習、(iii) AI の合理的な使用、(iv) 不確実性の推定
- データの拡張と合成: 幾何学的変換、フリッピング、色の変更、クロッピング、回転、ノイズ注入、ランダム消去などの実行可能な基本戦略、および敵対的生成ネットワークなどの新しい合成画像の作成を含むその他のより高度な手法を使用します。
- フェデレーテッド ラーニング: アルゴリズムは、データ サンプルを交換することなく、ローカル データ サンプルを保持する複数の分散型臨床サイトでトレーニングされます。ローカルでトレーニングされた AI の結果は、中央の場所で結合されます。
- 公正な使用: 臨床現場で収集される定期的な臨床データには、欠陥、偏り (性別の不均衡など)、またはノイズが発生しやすい (画像アーチファクトの存在など) 場合があります。これらの問題を効率的に追跡するアルゴリズムを定義することで、ある程度の進歩を遂げることができます
- 不確実性: モデルの精度。臨床医がソフトウェアにどの程度依存しているかを示します
転載: blog.csdn.net/qq_56061892/article/details/126224572








