
1ケースの説明(グラフ畳み込みニューラルネットワーク)
CORAデータセットには、各論文のキーワードと分類情報、および論文間の相互引用の情報が含まれています。AIモデルを構築し、データセット内の論文情報を分析し、既存の論文の分類特性に従って、分類が不明な論文のカテゴリを予測します。
1.1グラフ畳み込みニューラルネットワークを使用する機能
分類は、グラフニューラルネットワークを使用して実装されます。深層学習モデルとの違いは、グラフニューラルネットワークがテキスト自体の特性と論文間の関係を処理に使用し、良好な結果を達成するために必要なサンプル数が少ないことです。
1.2CORAデータセット
CORAデータセットは、機械学習の論文から編集され、各論文で使用されているキーワードと論文間の関係が記録されています。
1.2.1CORAの内容
CORAデータセットの論文は、ケースベース、遺伝的アルゴリズム、ニューラルネットワーク、確率的手法、強化学習、ルール学習、理論の7つのカテゴリに分類されます。
1.2.2CORAの構成
データセットには合計2708の論文があり、それぞれが少なくとも1つの他の論文を引用または引用しています。コーパス全体に2708の論文があります。同時に、すべての論文の語幹、ストップワード、低頻度の単語が削除され、論文の個々の特徴として1433個のキーワードが残されました。

1.2.3CORAデータセットのファイルと構造の説明
(1)コンテンツファイル形式での論文の説明:
<paper-id> <word-attributes> <class-label>
各行の最初のエントリには、論文の一意の文字列IDが含まれ、その後に、語彙の各単語が論文に存在する(1で表される)か存在しない(0で表される)かを示すバイナリ値が続きます。行の最後の項目には、紙のクラスラベルが含まれています。
(2)citesファイルには、コーパスの引用グラフが含まれており、各行には次の形式のリンクが記述されています。
<参考論文のID><参考論文のID>
各行には2つの紙のIDが含まれています。最初のエントリは引用された論文のIDであり、2番目のIDは引用を含む論文を表します。リンクの方向は右から左です。線が「paper2paper1」で表されている場合、接続は「paper2->paper1」です。
2コードの記述
2.1コードコンバット:基本モジュールを紹介し、実行環境をセットアップします----Cora_GNN.py(パート1)
from pathlib import Path # 引入提升路径的兼容性
# 引入矩阵运算的相关库
import numpy as np
import pandas as pd
from scipy.sparse import coo_matrix,csr_matrix,diags,eye
# 引入深度学习框架库
import torch
from torch import nn
import torch.nn.functional as F
# 引入绘图库
import matplotlib.pyplot as plt
import os
os.environ["KMP_DUPLICATE_LIB_OK"]="TRUE"
# 1.1 导入基础模块,并设置运行环境
# 输出计算资源情况
device = torch.device('cuda')if torch.cuda.is_available() else torch.device('cpu')
print(device) # 输出 cuda
# 输出样本路径
path = Path('./data/cora')
print(path) # 输出 cuda出力結果:
2.2コードの実装:紙のデータの読み取りと解析----Cora_GNN.py(パート2)
# 1.2 读取并解析论文数据
# 读取论文内容数据,将其转化为数据
paper_features_label = np.genfromtxt(path/'cora.content',dtype=np.str_) # 使用Path对象的路径构造,实例化的内容为cora.content。path/'cora.content'表示路径为'data/cora/cora.content'的字符串
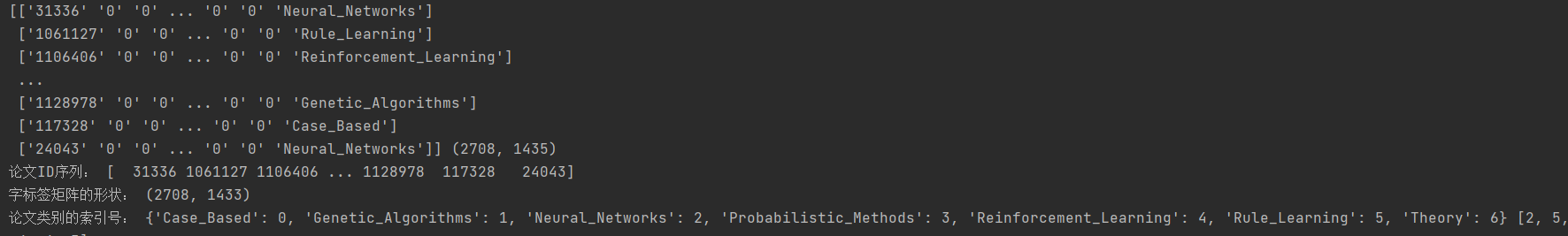
print(paper_features_label,np.shape(paper_features_label)) # 打印数据集内容与数据的形状
# 取出数据集中的第一列:论文ID
papers = paper_features_label[:,0].astype(np.int32)
print("论文ID序列:",papers) # 输出所有论文ID
# 论文重新编号,并将其映射到论文ID中,实现论文的统一管理
paper2idx = {k:v for v,k in enumerate(papers)}
# 将数据中间部分的字标签取出,转化成矩阵
features = csr_matrix(paper_features_label[:,1:-1],dtype=np.float32)
print("字标签矩阵的形状:",np.shape(features)) # 字标签矩阵的形状
# 将数据的最后一项的文章分类属性取出,转化为分类的索引
labels = paper_features_label[:,-1]
lbl2idx = { k:v for v,k in enumerate(sorted(np.unique(labels)))}
labels = [lbl2idx[e] for e in labels]
print("论文类别的索引号:",lbl2idx,labels[:5])出力:
2.3紙のリレーショナルデータを読み取って解析する
論文の関係データを読み込み、データ内の論文IDで表される関係を番号を付け直した関係に変換し、各論文を頂点として扱い、論文間の引用関係をエッジとして扱い、論文の関係データでグラフ構造で表されます。

このグラフ構造の隣接行列を計算し、それを無向グラフ隣接行列に変換します。
2.3.1コードの実装:変換行列----Cora_GNN.py(パート3)
# 1.3 读取并解析论文关系数据
# 读取论文关系数据,并将其转化为数据
edges = np.genfromtxt(path/'cora.cites',dtype=np.int32) # 将数据集中论文的引用关系以数据的形式读入
print(edges,np.shape(edges))
# 转化为新编号节点间的关系:将数据集中论文ID表示的关系转化为重新编号后的关系
edges = np.asarray([paper2idx[e] for e in edges.flatten()],np.int32).reshape(edges.shape)
print("新编号节点间的对应关系:",edges,edges.shape)
# 计算邻接矩阵,行与列都是论文个数:由论文引用关系所表示的图结构生成邻接矩阵。
adj = coo_matrix((np.ones(edges.shape[0]), (edges[:, 0], edges[:, 1])),shape=(len(labels), len(labels)), dtype=np.float32)
# 生成无向图对称矩阵:将有向图的邻接矩阵转化为无向图的邻接矩阵。Tip:转化为无向图的原因:主要用于对论文的分类,论文的引用关系主要提供单个特征之间的关联,故更看重是不是有关系,所以无向图即可。
adj_long = adj.multiply(adj.T < adj)
adj = adj_long + adj_long.T出力:
2.4処理グラフ構造のマトリックスデータ
グラフ構造の行列データは、グラフ構造の特徴をよりよく示すために処理され、ニューラルネットワークのモデル計算に関与します。
2.4.1グラフ構造のマトリックスデータを処理する手順
1.各ノードの特徴データを正規化します。
2.隣接行列の対角線に1を追加します:分類タスクでは、隣接行列の主な機能は、論文間の関連付けを通じてノードの分類を支援することであるためです。対角線上のノードの場合、表現の意味はそれ自体とそれ自体の間の関係です。対角ノードを1(自己ループグラフ)に設定すると、ノードが分類タスクにも役立つことを示します。
3. 1で補完した後、隣接行列を正規化します。

2.4.2コードの実装:処理グラフ構造のマトリックスデータ----Cora_GNN.py(パート4)
# 1.4 加工图结构的矩阵数据
def normalize(mx): # 定义函数,对矩阵的数据进行归一化处理
rowsum = np.array(mx.sum(1)) # 计算每一篇论文的字数==>02 对A中的边数求和,计算出矩阵A的度矩阵D^的特征向量
r_inv = (rowsum ** -1).flatten() # 取总字数的倒数==>03 对矩阵A的度矩阵D^的特征向量求逆,并得到D^逆的特征向量
r_inv[np.isinf(r_inv)] = 0.0 # 将NaN值取为0
r_mat_inv = diags(r_inv) # 将总字数的倒数变为对角矩阵===》对图结构的度矩阵求逆==>04 D^逆的特征向量转化为对角矩阵,得到D^逆
mx = r_mat_inv.dot(mx) # 左乘一个矩阵,相当于每个元素除以总数===》对每个论文顶点的边进行归一化处理==>05 计算D^逆与A加入自环(对角线为1)的邻接矩阵所得A^的点积,得到拉普拉斯矩阵。
return mx
# 对features矩阵进行归一化处理(每行总和为1)
features = normalize(features) #在函数normalize()中,分为两步对邻接矩阵进行处理。1、将每篇论文总字数的倒数变成对角矩阵。该操作相当于对图结构的度矩阵求逆。2、用度矩阵的逆左乘邻接矩阵,相当于对图中每个论文顶点的边进行归一化处理。
# 对邻接矩阵的对角线添1,将其变为自循环图,同时对其进行归一化处理
adj = normalize(adj + eye(adj.shape[0])) # 对角线补1==>01实现加入自环的邻接矩阵A2.5データをテンソルに変換し、コンピューティングリソースを割り当てます
処理されたグラフ構造行列データをPyTorchでサポートされているテンソル型に変換し、トレーニング、テスト、検証のために3つの部分に分割します。
2.5.1コードの実装:データをテンソルに変換し、コンピューティングリソースを割り当てる----Cora_GNN.py(パート5)
# 1.5 将数据转化为张量,并分配运算资源
adj = torch.FloatTensor(adj.todense()) # 节点间关系 todense()方法将其转换回稠密矩阵。
features = torch.FloatTensor(features.todense()) # 节点自身的特征
labels = torch.LongTensor(labels) # 对每个节点的分类标签
# 划分数据集
n_train = 200 # 训练数据集大小
n_val = 300 # 验证数据集大小
n_test = len(features) - n_train - n_val # 测试数据集大小
np.random.seed(34)
idxs = np.random.permutation(len(features)) # 将原有的索引打乱顺序
# 计算每个数据集的索引
idx_train = torch.LongTensor(idxs[:n_train]) # 根据指定训练数据集的大小并划分出其对应的训练数据集索引
idx_val = torch.LongTensor(idxs[n_train:n_train+n_val])# 根据指定验证数据集的大小并划分出其对应的验证数据集索引
idx_test = torch.LongTensor(idxs[n_train+n_val:])# 根据指定测试数据集的大小并划分出其对应的测试数据集索引
# 分配运算资源
adj = adj.to(device)
features = features.to(device)
labels = labels.to(device)
idx_train = idx_train.to(device)
idx_val = idx_val.to(device)
idx_test = idx_test.to(device)2.6グラフの畳み込み
グラフの畳み込みの本質は、次元変換です。つまり、次元内の各ノード特徴データを次元なしのノード特徴データに変換します。
グラフ畳み込みの操作は、入力ノードの特徴、重みパラメーター、および処理された隣接行列を組み合わせて、内積操作を実行します。
重みパラメータは、サイズin×outの行列です。ここで、inは入力ノードの特徴次元を表し、outは出力される最終的な特徴次元を表します。次元変換における重みパラメーターの関数は、完全に接続されたネットワークの重みとして理解されますが、グラフの畳み込みでは、完全に接続されたネットワークよりもノード関係情報の内積演算を実行します。
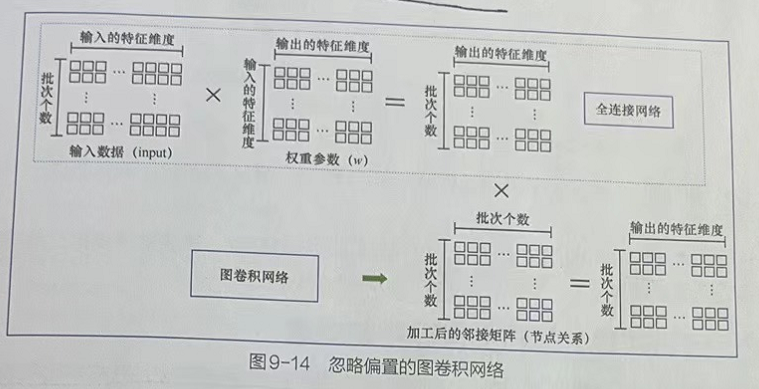
上図に示すように、バイアスを無視した後の完全接続ネットワークとグラフ畳み込みネットワークの関係を示します。このことから、グラフ畳み込みネットワークは、完全に接続されたネットワークに基づいてノード関係情報を実際に追加していることがはっきりとわかります。
2.6.1コードの実装:Mishの活性化関数とグラフの畳み込み演算クラスを定義する----Cora_GNN.py(パート6)
上の図に示されているアルゴリズムベースにバイアスを追加し、GraphConvolutionクラスを定義します
# 1.6 定义Mish激活函数与图卷积操作类
def mish(x): # 性能优于RElu函数
return x * (torch.tanh(F.softplus(x)))
# 图卷积类
class GraphConvolution(nn.Module):
def __init__(self,f_in,f_out,use_bias = True,activation=mish):
# super(GraphConvolution, self).__init__()
super().__init__()
self.f_in = f_in
self.f_out = f_out
self.use_bias = use_bias
self.activation = activation
self.weight = nn.Parameter(torch.FloatTensor(f_in, f_out))
self.bias = nn.Parameter(torch.FloatTensor(f_out)) if use_bias else None
self.initialize_weights()
def initialize_weights(self):# 对参数进行初始化
if self.activation is None: # 初始化权重
nn.init.xavier_uniform_(self.weight)
else:
nn.init.kaiming_uniform_(self.weight, nonlinearity='leaky_relu')
if self.use_bias:
nn.init.zeros_(self.bias)
def forward(self,input,adj): # 实现模型的正向处理流程
support = torch.mm(input,self.weight) # 节点特征与权重点积:torch.mm()实现矩阵的相乘,仅支持二位矩阵。若是多维矩则使用torch.matmul()
output = torch.mm(adj,support) # 将加工后的邻接矩阵放入点积运算
if self.use_bias:
output.add_(self.bias) # 加入偏置
if self.activation is not None:
output = self.activation(output) # 激活函数处理
return output2.7多層グラフ畳み込みの構築
GCNクラスを定義して、GraphConvolutionクラスによって完成されたグラフ畳み込み層をスタックし、多層グラフ畳み込みネットワークを形成します。同時に、トレーニングおよび評価機能がネットワークモデルに実装されます。
2.7.1コードの実装:多層グラフの畳み込み----Cora_GNN.py(パート7)
# 1.7 搭建多层图卷积网络模型
class GCN(nn.Module):
def __init__(self, f_in, n_classes, hidden=[16], dropout_p=0.5): # 实现多层图卷积网络,该网的搭建方法与全连接网络的搭建一致,只是将全连接层转化成GraphConvolution所实现的图卷积层
# super(GCN, self).__init__()
super().__init__()
layers = []
# 根据参数构建多层网络
for f_in, f_out in zip([f_in] + hidden[:-1], hidden):
# python 在list上的“+=”的重载函数是extend()函数,而不是+
# layers = [GraphConvolution(f_in, f_out)] + layers
layers += [GraphConvolution(f_in, f_out)]
self.layers = nn.Sequential(*layers)
self.dropout_p = dropout_p
# 构建输出层
self.out_layer = GraphConvolution(f_out, n_classes, activation=None)
def forward(self, x, adj): # 实现前向处理过程
for layer in self.layers:
x = layer(x,adj)
# 函数方式调用dropout():必须指定模型的运行状态,即Training标志,这样可减少很多麻烦
F.dropout(x,self.dropout_p,training=self.training,inplace=True)
return self.out_layer(x,adj)
n_labels = labels.max().item() + 1 # 获取分类个数7
n_features = features.shape[1] # 获取节点特征维度 1433
print(n_labels,n_features) # 输出7与1433
def accuracy(output,y): # 定义函数计算准确率
return (output.argmax(1) == y).type(torch.float32).mean().item()
### 定义函数来实现模型的训练过程。与深度学习任务不同,图卷积在训练时需要传入样本间的关系数据。
# 因为该关系数据是与节点数相等的方阵,所以传入的样本数也要与节点数相同,在计算loss值时,可以通过索引从总的运算结果中取出训练集的结果。
def step(): # 定义函数来训练模型 Tip:在图卷积任务中,无论是用模型进行预测还是训练,都需要将全部的图结构方阵输入
model.train()
optimizer.zero_grad()
output = model(features,adj) # 将全部数据载入模型,只用训练数据计算损失
loss = F.cross_entropy(output[idx_train],labels[idx_train])
acc = accuracy(output[idx_train],labels[idx_train]) # 计算准确率
loss.backward()
optimizer.step()
return loss.item(),acc
def evaluate(idx): # 定义函数来评估模型 Tip:在图卷积任务中,无论是用模型进行预测还是训练,都需要将全部的图结构方阵输入
model.eval()
output = model(features, adj) # 将全部数据载入模型,用指定索引评估模型结果
loss = F.cross_entropy(output[idx], labels[idx]).item()
return loss, accuracy(output[idx], labels[idx])2.8レンジャーオプティマイザー
グラフ畳み込みニューラルネットワークの層の数は多すぎてはならず、通常は約3層です。この例では、3層グラフの畳み込みニューラルネットワークを実装します。各層の寸法変化を図9-15に示します。
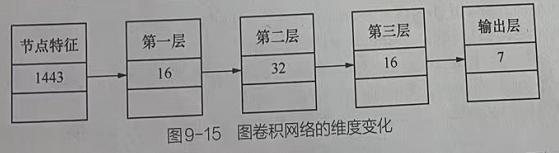
ループステートメントを使用してモデルをトレーニングし、モデルの結果を視覚化します。
2.8.1コードの実装:レンジャーオプティマイザーを使用してモデルをトレーニングし、結果を視覚化する---- Cora_GNN.py(パート8)
# 1.8 使用Ranger优化器训练模型并可视化
model = GCN(n_features, n_labels, hidden=[16, 32, 16]).to(device)
from tqdm import tqdm
from Cora_ranger import * # 引入Ranger优化器
optimizer = Ranger(model.parameters()) # 使用Ranger优化器
# 训练模型
epochs = 1000
print_steps = 50
train_loss, train_acc = [], []
val_loss, val_acc = [], []
for i in tqdm(range(epochs)):
tl,ta = step()
train_loss = train_loss + [tl]
train_acc = train_acc + [ta]
if (i+1) % print_steps == 0 or i == 0:
tl,ta = evaluate(idx_train)
vl,va = evaluate(idx_val)
val_loss = val_loss + [vl]
val_acc = val_acc + [va]
print(f'{i + 1:6d}/{epochs}: train_loss={tl:.4f}, train_acc={ta:.4f}' + f', val_loss={vl:.4f}, val_acc={va:.4f}')
# 输出最终结果
final_train, final_val, final_test = evaluate(idx_train), evaluate(idx_val), evaluate(idx_test)
print(f'Train : loss={final_train[0]:.4f}, accuracy={final_train[1]:.4f}')
print(f'Validation: loss={final_val[0]:.4f}, accuracy={final_val[1]:.4f}')
print(f'Test : loss={final_test[0]:.4f}, accuracy={final_test[1]:.4f}')
# 可视化训练过程
fig, axes = plt.subplots(1, 2, figsize=(15,5))
ax = axes[0]
axes[0].plot(train_loss[::print_steps] + [train_loss[-1]], label='Train')
axes[0].plot(val_loss, label='Validation')
axes[1].plot(train_acc[::print_steps] + [train_acc[-1]], label='Train')
axes[1].plot(val_acc, label='Validation')
for ax,t in zip(axes, ['Loss', 'Accuracy']): ax.legend(), ax.set_title(t, size=15)
# 输出模型的预测结果
output = model(features, adj)
samples = 10
idx_sample = idx_test[torch.randperm(len(idx_test))[:samples]]
# 将样本标签与预测结果进行比较
idx2lbl = {v:k for k,v in lbl2idx.items()}
df = pd.DataFrame({'Real': [idx2lbl[e] for e in labels[idx_sample].tolist()],'Pred': [idx2lbl[e] for e in output[idx_sample].argmax(1).tolist()]})
print(df)2.7プログラム出力の要約
2.7.1トレーニングプロセス
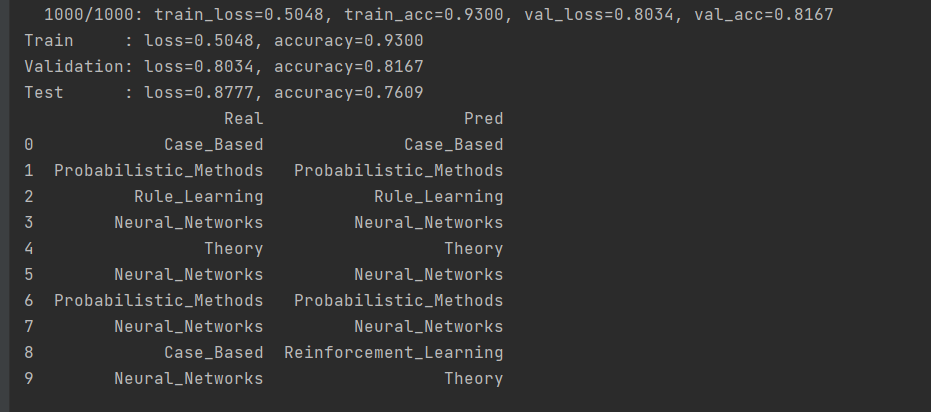
2.7.3検証結果
2.8結論
トレーニング結果から、モデルのフィッティング能力が優れていることがわかります。グラフ畳み込みモデルが使用するトレーニングサンプルはごくわずかであり、2708サンプルのうち200のみがトレーニングに使用されます。サンプル間の関係情報が追加されるため、サンプルサイズへのモデルの依存性が大幅に減少します。これは、グラフニューラルネットワークモデルの利点でもあります。
3コードの概要
3.1 Cora_GNN.py
from pathlib import Path # 引入提升路径的兼容性
# 引入矩阵运算的相关库
import numpy as np
import pandas as pd
from scipy.sparse import coo_matrix,csr_matrix,diags,eye
# 引入深度学习框架库
import torch
from torch import nn
import torch.nn.functional as F
# 引入绘图库
import matplotlib.pyplot as plt
import os
os.environ["KMP_DUPLICATE_LIB_OK"]="TRUE"
# 1.1 导入基础模块,并设置运行环境
# 输出计算资源情况
device = torch.device('cuda')if torch.cuda.is_available() else torch.device('cpu')
print(device) # 输出 cuda
# 输出样本路径
path = Path('./data/cora')
print(path) # 输出 cuda
# 1.2 读取并解析论文数据
# 读取论文内容数据,将其转化为数据
paper_features_label = np.genfromtxt(path/'cora.content',dtype=np.str_) # 使用Path对象的路径构造,实例化的内容为cora.content。path/'cora.content'表示路径为'data/cora/cora.content'的字符串
print(paper_features_label,np.shape(paper_features_label)) # 打印数据集内容与数据的形状
# 取出数据集中的第一列:论文ID
papers = paper_features_label[:,0].astype(np.int32)
print("论文ID序列:",papers) # 输出所有论文ID
# 论文重新编号,并将其映射到论文ID中,实现论文的统一管理
paper2idx = {k:v for v,k in enumerate(papers)}
# 将数据中间部分的字标签取出,转化成矩阵
features = csr_matrix(paper_features_label[:,1:-1],dtype=np.float32)
print("字标签矩阵的形状:",np.shape(features)) # 字标签矩阵的形状
# 将数据的最后一项的文章分类属性取出,转化为分类的索引
labels = paper_features_label[:,-1]
lbl2idx = { k:v for v,k in enumerate(sorted(np.unique(labels)))}
labels = [lbl2idx[e] for e in labels]
print("论文类别的索引号:",lbl2idx,labels[:5])
# 1.3 读取并解析论文关系数据
# 读取论文关系数据,并将其转化为数据
edges = np.genfromtxt(path/'cora.cites',dtype=np.int32) # 将数据集中论文的引用关系以数据的形式读入
print(edges,np.shape(edges))
# 转化为新编号节点间的关系:将数据集中论文ID表示的关系转化为重新编号后的关系
edges = np.asarray([paper2idx[e] for e in edges.flatten()],np.int32).reshape(edges.shape)
print("新编号节点间的对应关系:",edges,edges.shape)
# 计算邻接矩阵,行与列都是论文个数:由论文引用关系所表示的图结构生成邻接矩阵。
adj = coo_matrix((np.ones(edges.shape[0]), (edges[:, 0], edges[:, 1])),shape=(len(labels), len(labels)), dtype=np.float32)
# 生成无向图对称矩阵:将有向图的邻接矩阵转化为无向图的邻接矩阵。Tip:转化为无向图的原因:主要用于对论文的分类,论文的引用关系主要提供单个特征之间的关联,故更看重是不是有关系,所以无向图即可。
adj_long = adj.multiply(adj.T < adj)
adj = adj_long + adj_long.T
# 1.4 加工图结构的矩阵数据
def normalize(mx): # 定义函数,对矩阵的数据进行归一化处理
rowsum = np.array(mx.sum(1)) # 计算每一篇论文的字数==>02 对A中的边数求和,计算出矩阵A的度矩阵D^的特征向量
r_inv = (rowsum ** -1).flatten() # 取总字数的倒数==>03 对矩阵A的度矩阵D^的特征向量求逆,并得到D^逆的特征向量
r_inv[np.isinf(r_inv)] = 0.0 # 将NaN值取为0
r_mat_inv = diags(r_inv) # 将总字数的倒数变为对角矩阵===》对图结构的度矩阵求逆==>04 D^逆的特征向量转化为对角矩阵,得到D^逆
mx = r_mat_inv.dot(mx) # 左乘一个矩阵,相当于每个元素除以总数===》对每个论文顶点的边进行归一化处理==>05 计算D^逆与A加入自环(对角线为1)的邻接矩阵所得A^的点积,得到拉普拉斯矩阵。
return mx
# 对features矩阵进行归一化处理(每行总和为1)
features = normalize(features) #在函数normalize()中,分为两步对邻接矩阵进行处理。1、将每篇论文总字数的倒数变成对角矩阵。该操作相当于对图结构的度矩阵求逆。2、用度矩阵的逆左乘邻接矩阵,相当于对图中每个论文顶点的边进行归一化处理。
# 对邻接矩阵的对角线添1,将其变为自循环图,同时对其进行归一化处理
adj = normalize(adj + eye(adj.shape[0])) # 对角线补1==>01实现加入自环的邻接矩阵A
# 1.5 将数据转化为张量,并分配运算资源
adj = torch.FloatTensor(adj.todense()) # 节点间关系 todense()方法将其转换回稠密矩阵。
features = torch.FloatTensor(features.todense()) # 节点自身的特征
labels = torch.LongTensor(labels) # 对每个节点的分类标签
# 划分数据集
n_train = 200 # 训练数据集大小
n_val = 300 # 验证数据集大小
n_test = len(features) - n_train - n_val # 测试数据集大小
np.random.seed(34)
idxs = np.random.permutation(len(features)) # 将原有的索引打乱顺序
# 计算每个数据集的索引
idx_train = torch.LongTensor(idxs[:n_train]) # 根据指定训练数据集的大小并划分出其对应的训练数据集索引
idx_val = torch.LongTensor(idxs[n_train:n_train+n_val])# 根据指定验证数据集的大小并划分出其对应的验证数据集索引
idx_test = torch.LongTensor(idxs[n_train+n_val:])# 根据指定测试数据集的大小并划分出其对应的测试数据集索引
# 分配运算资源
adj = adj.to(device)
features = features.to(device)
labels = labels.to(device)
idx_train = idx_train.to(device)
idx_val = idx_val.to(device)
idx_test = idx_test.to(device)
# 1.6 定义Mish激活函数与图卷积操作类
def mish(x): # 性能优于RElu函数
return x * (torch.tanh(F.softplus(x)))
# 图卷积类
class GraphConvolution(nn.Module):
def __init__(self,f_in,f_out,use_bias = True,activation=mish):
# super(GraphConvolution, self).__init__()
super().__init__()
self.f_in = f_in
self.f_out = f_out
self.use_bias = use_bias
self.activation = activation
self.weight = nn.Parameter(torch.FloatTensor(f_in, f_out))
self.bias = nn.Parameter(torch.FloatTensor(f_out)) if use_bias else None
self.initialize_weights()
def initialize_weights(self):# 对参数进行初始化
if self.activation is None: # 初始化权重
nn.init.xavier_uniform_(self.weight)
else:
nn.init.kaiming_uniform_(self.weight, nonlinearity='leaky_relu')
if self.use_bias:
nn.init.zeros_(self.bias)
def forward(self,input,adj): # 实现模型的正向处理流程
support = torch.mm(input,self.weight) # 节点特征与权重点积:torch.mm()实现矩阵的相乘,仅支持二位矩阵。若是多维矩则使用torch.matmul()
output = torch.mm(adj,support) # 将加工后的邻接矩阵放入点积运算
if self.use_bias:
output.add_(self.bias) # 加入偏置
if self.activation is not None:
output = self.activation(output) # 激活函数处理
return output
# 1.7 搭建多层图卷积网络模型
class GCN(nn.Module):
def __init__(self, f_in, n_classes, hidden=[16], dropout_p=0.5): # 实现多层图卷积网络,该网的搭建方法与全连接网络的搭建一致,只是将全连接层转化成GraphConvolution所实现的图卷积层
# super(GCN, self).__init__()
super().__init__()
layers = []
# 根据参数构建多层网络
for f_in, f_out in zip([f_in] + hidden[:-1], hidden):
# python 在list上的“+=”的重载函数是extend()函数,而不是+
# layers = [GraphConvolution(f_in, f_out)] + layers
layers += [GraphConvolution(f_in, f_out)]
self.layers = nn.Sequential(*layers)
self.dropout_p = dropout_p
# 构建输出层
self.out_layer = GraphConvolution(f_out, n_classes, activation=None)
def forward(self, x, adj): # 实现前向处理过程
for layer in self.layers:
x = layer(x,adj)
# 函数方式调用dropout():必须指定模型的运行状态,即Training标志,这样可减少很多麻烦
F.dropout(x,self.dropout_p,training=self.training,inplace=True)
return self.out_layer(x,adj)
n_labels = labels.max().item() + 1 # 获取分类个数7
n_features = features.shape[1] # 获取节点特征维度 1433
print(n_labels,n_features) # 输出7与1433
def accuracy(output,y): # 定义函数计算准确率
return (output.argmax(1) == y).type(torch.float32).mean().item()
### 定义函数来实现模型的训练过程。与深度学习任务不同,图卷积在训练时需要传入样本间的关系数据。
# 因为该关系数据是与节点数相等的方阵,所以传入的样本数也要与节点数相同,在计算loss值时,可以通过索引从总的运算结果中取出训练集的结果。
def step(): # 定义函数来训练模型 Tip:在图卷积任务中,无论是用模型进行预测还是训练,都需要将全部的图结构方阵输入
model.train()
optimizer.zero_grad()
output = model(features,adj) # 将全部数据载入模型,只用训练数据计算损失
loss = F.cross_entropy(output[idx_train],labels[idx_train])
acc = accuracy(output[idx_train],labels[idx_train]) # 计算准确率
loss.backward()
optimizer.step()
return loss.item(),acc
def evaluate(idx): # 定义函数来评估模型 Tip:在图卷积任务中,无论是用模型进行预测还是训练,都需要将全部的图结构方阵输入
model.eval()
output = model(features, adj) # 将全部数据载入模型,用指定索引评估模型结果
loss = F.cross_entropy(output[idx], labels[idx]).item()
return loss, accuracy(output[idx], labels[idx])
# 1.8 使用Ranger优化器训练模型并可视化
model = GCN(n_features, n_labels, hidden=[16, 32, 16]).to(device)
from tqdm import tqdm
from Cora_ranger import * # 引入Ranger优化器
optimizer = Ranger(model.parameters()) # 使用Ranger优化器
# 训练模型
epochs = 1000
print_steps = 50
train_loss, train_acc = [], []
val_loss, val_acc = [], []
for i in tqdm(range(epochs)):
tl,ta = step()
train_loss = train_loss + [tl]
train_acc = train_acc + [ta]
if (i+1) % print_steps == 0 or i == 0:
tl,ta = evaluate(idx_train)
vl,va = evaluate(idx_val)
val_loss = val_loss + [vl]
val_acc = val_acc + [va]
print(f'{i + 1:6d}/{epochs}: train_loss={tl:.4f}, train_acc={ta:.4f}' + f', val_loss={vl:.4f}, val_acc={va:.4f}')
# 输出最终结果
final_train, final_val, final_test = evaluate(idx_train), evaluate(idx_val), evaluate(idx_test)
print(f'Train : loss={final_train[0]:.4f}, accuracy={final_train[1]:.4f}')
print(f'Validation: loss={final_val[0]:.4f}, accuracy={final_val[1]:.4f}')
print(f'Test : loss={final_test[0]:.4f}, accuracy={final_test[1]:.4f}')
# 可视化训练过程
fig, axes = plt.subplots(1, 2, figsize=(15,5))
ax = axes[0]
axes[0].plot(train_loss[::print_steps] + [train_loss[-1]], label='Train')
axes[0].plot(val_loss, label='Validation')
axes[1].plot(train_acc[::print_steps] + [train_acc[-1]], label='Train')
axes[1].plot(val_acc, label='Validation')
for ax,t in zip(axes, ['Loss', 'Accuracy']): ax.legend(), ax.set_title(t, size=15)
# 输出模型的预测结果
output = model(features, adj)
samples = 10
idx_sample = idx_test[torch.randperm(len(idx_test))[:samples]]
# 将样本标签与预测结果进行比较
idx2lbl = {v:k for k,v in lbl2idx.items()}
df = pd.DataFrame({'Real': [idx2lbl[e] for e in labels[idx_sample].tolist()],'Pred': [idx2lbl[e] for e in output[idx_sample].argmax(1).tolist()]})
print(df)3.2 Cora_ranger.py
#Ranger deep learning optimizer - RAdam + Lookahead combined.
#https://github.com/lessw2020/Ranger-Deep-Learning-Optimizer
#Ranger has now been used to capture 12 records on the FastAI leaderboard.
#This version = 9.3.19
#Credits:
#RAdam --> https://github.com/LiyuanLucasLiu/RAdam
#Lookahead --> rewritten by lessw2020, but big thanks to Github @LonePatient and @RWightman for ideas from their code.
#Lookahead paper --> MZhang,G Hinton https://arxiv.org/abs/1907.08610
#summary of changes:
#full code integration with all updates at param level instead of group, moves slow weights into state dict (from generic weights),
#supports group learning rates (thanks @SHolderbach), fixes sporadic load from saved model issues.
#changes 8/31/19 - fix references to *self*.N_sma_threshold;
#changed eps to 1e-5 as better default than 1e-8.
import math
import torch
from torch.optim.optimizer import Optimizer, required
import itertools as it
class Ranger(Optimizer):
def __init__(self, params, lr=1e-3, alpha=0.5, k=6, N_sma_threshhold=5, betas=(.95,0.999), eps=1e-5, weight_decay=0):
#parameter checks
if not 0.0 <= alpha <= 1.0:
raise ValueError(f'Invalid slow update rate: {alpha}')
if not 1 <= k:
raise ValueError(f'Invalid lookahead steps: {k}')
if not lr > 0:
raise ValueError(f'Invalid Learning Rate: {lr}')
if not eps > 0:
raise ValueError(f'Invalid eps: {eps}')
#parameter comments:
# beta1 (momentum) of .95 seems to work better than .90...
#N_sma_threshold of 5 seems better in testing than 4.
#In both cases, worth testing on your dataset (.90 vs .95, 4 vs 5) to make sure which works best for you.
#prep defaults and init torch.optim base
defaults = dict(lr=lr, alpha=alpha, k=k, step_counter=0, betas=betas, N_sma_threshhold=N_sma_threshhold, eps=eps, weight_decay=weight_decay)
super().__init__(params,defaults)
#adjustable threshold
self.N_sma_threshhold = N_sma_threshhold
#now we can get to work...
#removed as we now use step from RAdam...no need for duplicate step counting
#for group in self.param_groups:
# group["step_counter"] = 0
#print("group step counter init")
#look ahead params
self.alpha = alpha
self.k = k
#radam buffer for state
self.radam_buffer = [[None,None,None] for ind in range(10)]
#self.first_run_check=0
#lookahead weights
#9/2/19 - lookahead param tensors have been moved to state storage.
#This should resolve issues with load/save where weights were left in GPU memory from first load, slowing down future runs.
#self.slow_weights = [[p.clone().detach() for p in group['params']]
# for group in self.param_groups]
#don't use grad for lookahead weights
#for w in it.chain(*self.slow_weights):
# w.requires_grad = False
def __setstate__(self, state):
print("set state called")
super(Ranger, self).__setstate__(state)
def step(self, closure=None):
loss = None
#note - below is commented out b/c I have other work that passes back the loss as a float, and thus not a callable closure.
#Uncomment if you need to use the actual closure...
#if closure is not None:
#loss = closure()
#Evaluate averages and grad, update param tensors
for group in self.param_groups:
for p in group['params']:
if p.grad is None:
continue
grad = p.grad.data.float()
if grad.is_sparse:
raise RuntimeError('Ranger optimizer does not support sparse gradients')
p_data_fp32 = p.data.float()
state = self.state[p] #get state dict for this param
if len(state) == 0: #if first time to run...init dictionary with our desired entries
#if self.first_run_check==0:
#self.first_run_check=1
#print("Initializing slow buffer...should not see this at load from saved model!")
state['step'] = 0
state['exp_avg'] = torch.zeros_like(p_data_fp32)
state['exp_avg_sq'] = torch.zeros_like(p_data_fp32)
#look ahead weight storage now in state dict
state['slow_buffer'] = torch.empty_like(p.data)
state['slow_buffer'].copy_(p.data)
else:
state['exp_avg'] = state['exp_avg'].type_as(p_data_fp32)
state['exp_avg_sq'] = state['exp_avg_sq'].type_as(p_data_fp32)
#begin computations
exp_avg, exp_avg_sq = state['exp_avg'], state['exp_avg_sq']
beta1, beta2 = group['betas']
#compute variance mov avg
exp_avg_sq.mul_(beta2).addcmul_(1 - beta2, grad, grad)
#compute mean moving avg
exp_avg.mul_(beta1).add_(1 - beta1, grad)
state['step'] += 1
buffered = self.radam_buffer[int(state['step'] % 10)]
if state['step'] == buffered[0]:
N_sma, step_size = buffered[1], buffered[2]
else:
buffered[0] = state['step']
beta2_t = beta2 ** state['step']
N_sma_max = 2 / (1 - beta2) - 1
N_sma = N_sma_max - 2 * state['step'] * beta2_t / (1 - beta2_t)
buffered[1] = N_sma
if N_sma > self.N_sma_threshhold:
step_size = math.sqrt((1 - beta2_t) * (N_sma - 4) / (N_sma_max - 4) * (N_sma - 2) / N_sma * N_sma_max / (N_sma_max - 2)) / (1 - beta1 ** state['step'])
else:
step_size = 1.0 / (1 - beta1 ** state['step'])
buffered[2] = step_size
if group['weight_decay'] != 0:
p_data_fp32.add_(-group['weight_decay'] * group['lr'], p_data_fp32)
if N_sma > self.N_sma_threshhold:
denom = exp_avg_sq.sqrt().add_(group['eps'])
p_data_fp32.addcdiv_(-step_size * group['lr'], exp_avg, denom)
else:
p_data_fp32.add_(-step_size * group['lr'], exp_avg)
p.data.copy_(p_data_fp32)
#integrated look ahead...
#we do it at the param level instead of group level
if state['step'] % group['k'] == 0:
slow_p = state['slow_buffer'] #get access to slow param tensor
slow_p.add_(self.alpha, p.data - slow_p) #(fast weights - slow weights) * alpha
p.data.copy_(slow_p) #copy interpolated weights to RAdam param tensor
return loss