1.Principe
La spline cubique restreinte (RCS) est l'une des méthodes les plus courantes pour analyser les relations non linéaires. RCS utilise une fonction cubique pour ajuster les courbes entre différents nœuds et les connecter en douceur, réalisant ainsi le processus d'ajustement de la courbe entière et de test de sa linéarité. Comme vous pouvez l'imaginer, le nombre de nœuds dans RCS est très important pour les résultats d'ajustement. Habituellement, 3 nœuds sont utilisés pour les petits échantillons contenant moins de 30 échantillons et 5 nœuds sont utilisés pour les grands échantillons.
Implémentation 2.R
1.cox revient
#Used for RCS(Restricted Cubic Spline)
#我们使用rms包
library(ggplot2)
library(rms)
library(survminer)
library(survival)Ici, nous utilisons les données pulmonaires dans le package de survie
#####基于cox回归
#这里用survival包里的lung数据集来做范例分析
head(lung)
# inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
# 3 306 2 74 1 1 90 100 1175 NA
# 3 455 2 68 1 0 90 90 1225 15
# 3 1010 1 56 1 0 90 90 NA 15
# 5 210 2 57 1 1 90 60 1150 11
# 1 883 2 60 1 0 100 90 NA 0
# 12 1022 1 74 1 1 50 80 513 0
#status: censoring status 1=censored, 2=dead
#sex: Male=1 Female=2
#ph.ecog:医生对患者的体能状态评级
#ph.karno:医生对患者的另一种体能状态评级karnofsky
#pat.karno:患者karnofsky自评
#meal.cal:摄入卡路里
#wt.loss:过去半年体重减轻
# 对数据进行打包,整理
dd <- datadist(lung) #为后续程序设定数据环境
options(datadist='dd') #为后续程序设定数据环境
#用AIC法计算不同节点数选择下的模型拟合度来决定最佳节点数
for (knot in 3:10) {
fit <- cph(Surv(time,status) ~ rcs(meal.cal,knot) + sex+age , x=TRUE, y=TRUE,data=lung)
tmp <- extractAIC(fit)
if(knot==3){AIC=tmp[2];nk=3}
if(tmp[2]<AIC){AIC=tmp[2];nk=knot}
}
nk #3
#cox回归中自变量对HR的rcs
fit <- cph(Surv(time,status) ~ rcs(meal.cal,3) + sex+age , x=TRUE, y=TRUE,data=lung)#大样本5节点,小样本(<30)3节点
#比例风险PH假设检验,p>0.05满足假设检验
cox.zph(fit,"rank")
#非线性检验,p<0.05为有非线性关系
anova(fit)
#这里的结果是
# Wald Statistics Response: Surv(time, status)
#
# Factor Chi-Square d.f. P
# meal.cal 0.42 2 0.8113
# Nonlinear 0.09 1 0.7643,呈线性
# sex 6.61 1 0.0101
# age 1.99 1 0.1582
# TOTAL 10.29 4 0.0358
#查看各meal.cal对应的HR值
HR<-Predict(fit, meal.cal,fun=exp)
head(HR)
#画图
ggplot()+
geom_line(data=HR, aes(meal.cal,yhat),
linetype="solid",size=1,alpha = 0.7,colour="#0070b9")+
geom_ribbon(data=HR,
aes(meal.cal,ymin = lower, ymax = upper),
alpha = 0.1,fill="#0070b9")+
theme_classic()+
geom_hline(yintercept=1, linetype=2,size=1)+
labs(title = "Lung Cancer Risk", x="Age", y="HR (95%CI)") Le résultat est tracé comme indiqué sur la figure :
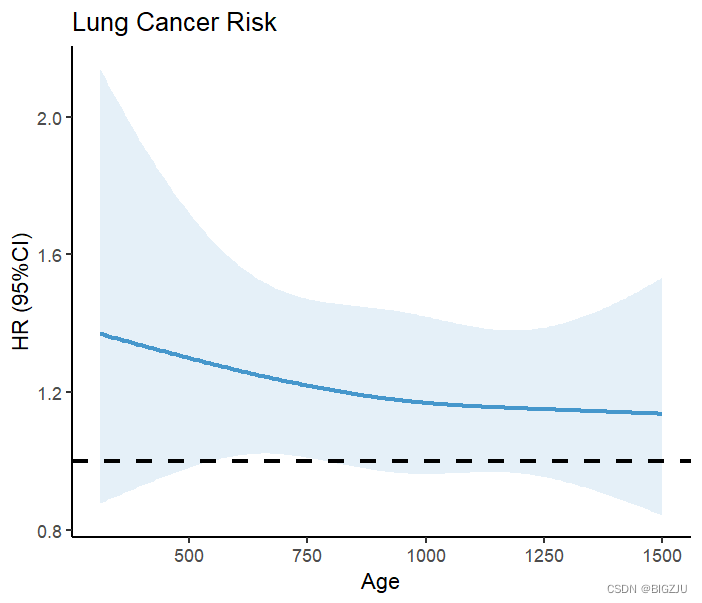
Les résultats de l'anova (fit) et de la présentation visuelle montrent une relation linéaire entre l'apport énergétique alimentaire et la mortalité sous RCS.
Trouvons un autre exemple non linéaire. Cet exemple utilise les données deux-points du package de survie :
#结肠癌病人数据
Colon <- colon
Colon$sex <- as.factor(Colon$sex)#1 for male,0 for female
Colon$etype <- as.factor(Colon$etype-1)
dd <- datadist(Colon)
options(datadist='dd')
for (knot in 3:10) {
fit <- cph(Surv(time,etype==1) ~ rcs(age,knot) +sex , x=TRUE, y=TRUE,data=Colon)
tmp <- extractAIC(fit)
if(knot==3){AIC=tmp[2];nk1=3}
if(tmp[2]<AIC){AIC=tmp[2];nk1=knot}
}
nk1 #3
fit <- cph(Surv(time,etype==1) ~ rcs(age,3) +sex , x=TRUE, y=TRUE,data=Colon)
cox.zph(fit,"rank")
anova(fit)
# Wald Statistics Response: Surv(time, etype == 1)
#
# Factor Chi-Square d.f. P
# age 6.90 2 0.0317
# Nonlinear 6.32 1 0.0120,非线性
# sex 1.20 1 0.2732
# TOTAL 7.54 3 0.0565
HR<-Predict(fit, age,fun=exp)
head(HR)
ggplot()+
geom_line(data=HR, aes(age,yhat),
linetype="solid",size=1,alpha = 0.7,colour="#0070b9")+
geom_ribbon(data=HR,
aes(age,ymin = lower, ymax = upper),
alpha = 0.1,fill="#0070b9")+
theme_classic()+
geom_hline(yintercept=1, linetype=2,size=1)+
geom_vline(xintercept=47.35176,size=1,color = '#d40e8c')+#查表HR=1对应的age
geom_vline(xintercept=65.26131,size=1,color = '#d40e8c')+
labs(title = "Colon Cancer Risk", x="Age", y="HR (95%CI)") Le résultat est le suivant :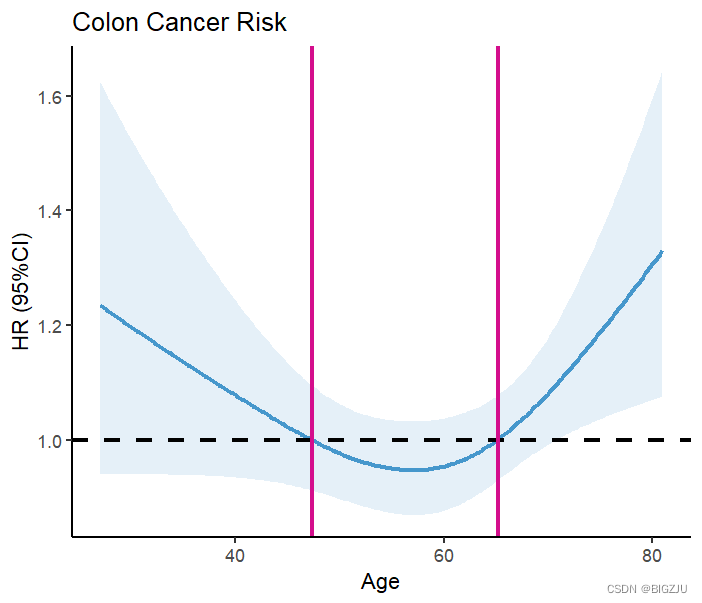
Les résultats de anova(fit) et la présentation visuelle montrent que l'exemple présente une relation non linéaire
Nous pouvons également effectuer des recherches groupées et une présentation visuelle, il suffit de modifier les paramètres dans la fonction Predict()
HR1 <- Predict(fit, age, sex=c('0','1'),
fun=exp,type="predictions",
conf.int = 0.95,digits =2)
HR1
ggplot()+
geom_line(data=HR1, aes(age,yhat, color = sex),
linetype="solid",size=1,alpha = 0.7)+
geom_ribbon(data=HR1,
aes(age,ymin = lower, ymax = upper,fill = sex),
alpha = 0.1)+
scale_color_manual(values = c('#0070b9','#d40e8c'))+
scale_fill_manual(values = c('#0070b9','#d40e8c'))+
theme_classic()+
geom_hline(yintercept=1, linetype=2,size=1)+
labs(title = "Colon Cancer Risk", x="Age", y="HR (95%CI)")
résultat:
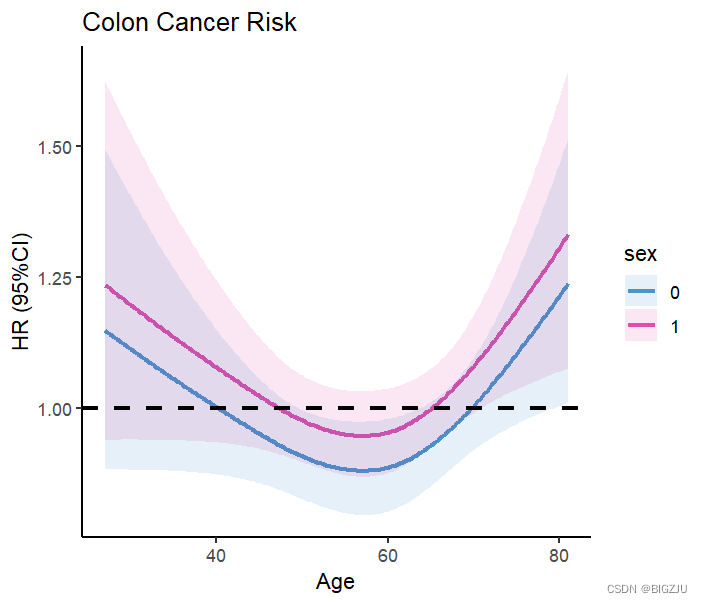
2.régression logistique
Si au lieu de la régression de Cox, on utilise la régression logistique, le résultat global est presque le même, il suffit de remplacer le modèle.
#####基于logistic回归的rcs
#建模型
fit <-lrm(status ~ rcs(age, 3)+sex,data=lung)
OR <- Predict(fit, age,fun=exp)
#画图
ggplot()+
geom_line(data=OR, aes(age,yhat),
linetype="solid",size=1,alpha = 0.7,colour="#0070b9")+
geom_ribbon(data=OR,
aes(age,ymin = lower, ymax = upper),
alpha = 0.1,fill="#0070b9")+
theme_classic()+
geom_hline(yintercept=1, linetype=2,size=1)+
geom_vline(xintercept=38.93970,size=1,color = '#d40e8c')+ #查表OR=1对应的age
labs(title = "Lung Cancer Risk", x="Age", y="OR (95%CI)")3. Régression linéaire
Il en va de même pour la régression linéaire
#####基于线性回归的rcs
fit <- ols(meal.cal ~rcs(age,3)+sex,data=lung)
Kcal <- Predict(fit,age)
#画图
ggplot()+
geom_line(data=Kcal, aes(age,yhat),
linetype="solid",size=1,alpha = 0.7,colour="#0070b9")+
geom_ribbon(data=Kcal,
aes(age,ymin = lower, ymax = upper),
alpha = 0.1,fill="#0070b9")+
theme_classic()+
labs(title = "RCS", x="Age", y="Kcal")