Dans cet article, nous expliquons comment créer KG, l'analyser et créer des modèles d'intégration.

Créer un graphique de connaissances
Chargez nos données. Dans cet article, nous allons créer un simple KG à partir de zéro.
import pandas as pd
# Define the heads, relations, and tails
head = ['drugA', 'drugB', 'drugC', 'drugD', 'drugA', 'drugC', 'drugD', 'drugE', 'gene1', 'gene2','gene3', 'gene4', 'gene50', 'gene2', 'gene3', 'gene4']
relation = ['treats', 'treats', 'treats', 'treats', 'inhibits', 'inhibits', 'inhibits', 'inhibits', 'associated', 'associated', 'associated', 'associated', 'associated', 'interacts', 'interacts', 'interacts']
tail = ['fever', 'hepatitis', 'bleeding', 'pain', 'gene1', 'gene2', 'gene4', 'gene20', 'obesity', 'heart_attack', 'hepatitis', 'bleeding', 'cancer', 'gene1', 'gene20', 'gene50']
# Create a dataframe
df = pd.DataFrame({'head': head, 'relation': relation, 'tail': tail})
df

Ensuite, créez un graphique NetworkX (G) pour représenter KG. Chaque ligne du DataFrame (df) correspond à un triplet (tête, relation, queue) en KG. La fonction add_edge ajoute des arêtes entre les entités de tête et de queue, avec des relations comme étiquettes.
import networkx as nx
import matplotlib.pyplot as plt
# Create a knowledge graph
G = nx.Graph()
for _, row in df.iterrows():
G.add_edge(row['head'], row['tail'], label=row['relation'])
Ensuite, tracez les nœuds (entités) et les arêtes (relations) ainsi que leurs étiquettes.
# Visualize the knowledge graph
pos = nx.spring_layout(G, seed=42, k=0.9)
labels = nx.get_edge_attributes(G, 'label')
plt.figure(figsize=(12, 10))
nx.draw(G, pos, with_labels=True, font_size=10, node_size=700, node_color='lightblue', edge_color='gray', alpha=0.6)
nx.draw_networkx_edge_labels(G, pos, edge_labels=labels, font_size=8, label_pos=0.3, verticalalignment='baseline')
plt.title('Knowledge Graph')
plt.show()
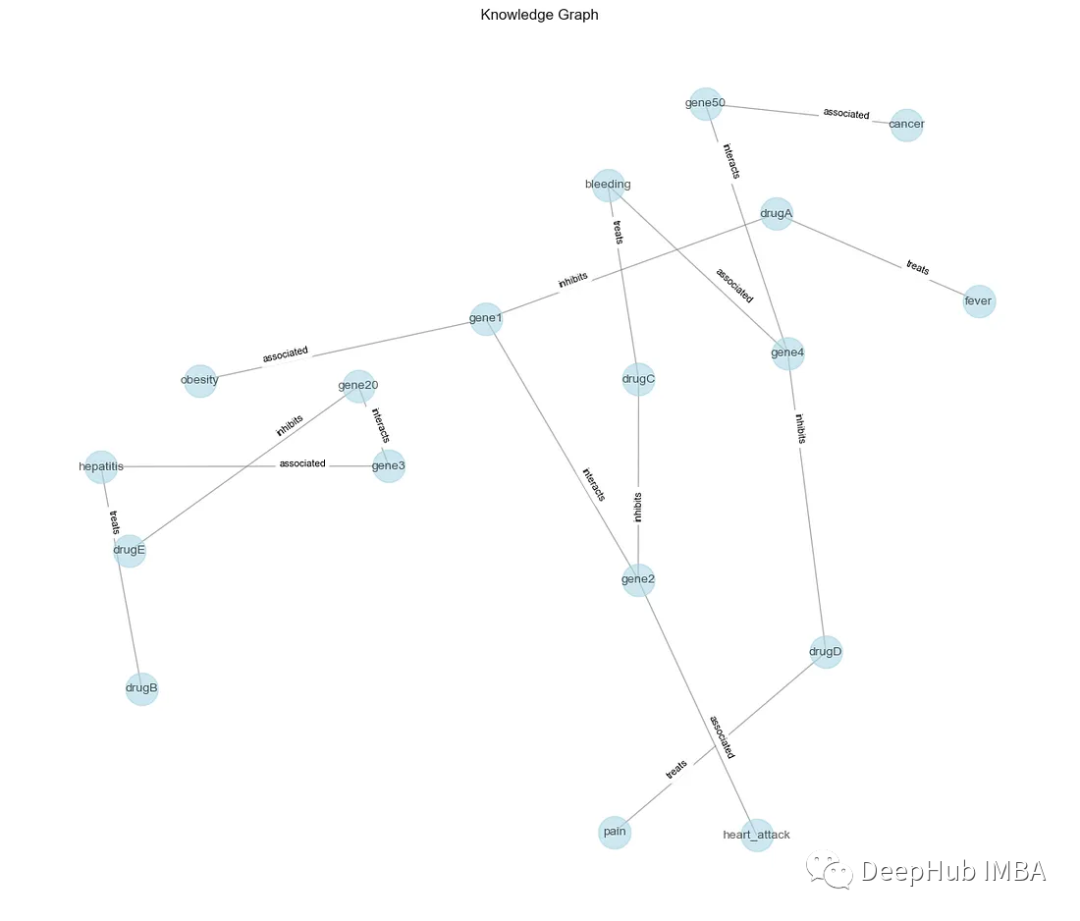
Nous pouvons maintenant faire une analyse.
analyser
Pour KG, la première chose que nous pouvons faire est de voir combien de nœuds et d’arêtes il possède, et d’analyser la relation entre eux.
num_nodes = G.number_of_nodes()
num_edges = G.number_of_edges()
print(f'Number of nodes: {num_nodes}')
print(f'Number of edges: {num_edges}')
print(f'Ratio edges to nodes: {round(num_edges / num_nodes, 2)}')

1. Analyse de la centralité des nœuds
La centralité des nœuds mesure l'importance ou l'influence des nœuds dans un graphique. Cela aide à identifier le nœud central de la structure du graphe. Certaines des mesures de centralité les plus courantes sont :
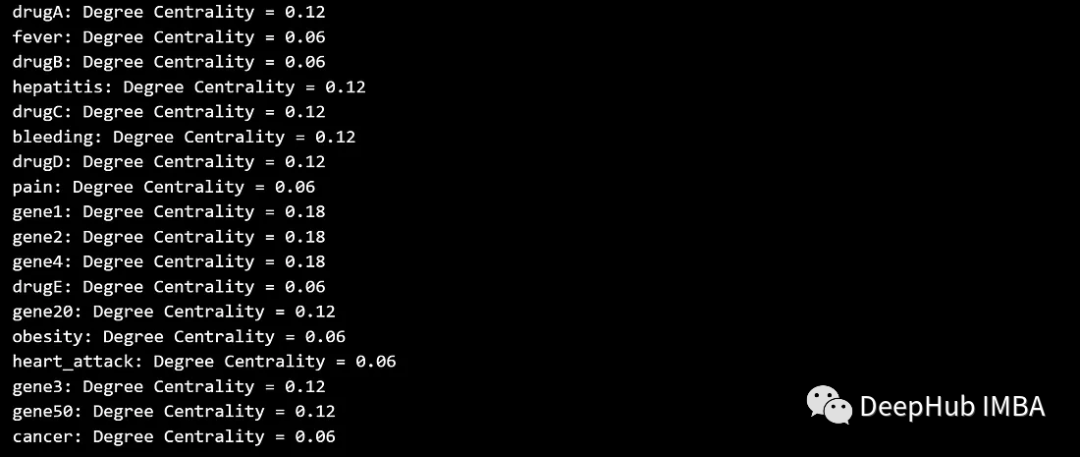
La centralité du degré compte le nombre d'arêtes associées sur un nœud. Les nœuds avec une centralité plus élevée sont plus étroitement connectés.
degree_centrality = nx.degree_centrality(G)
for node, centrality in degree_centrality.items():
print(f'{node}: Degree Centrality = {centrality:.2f}')
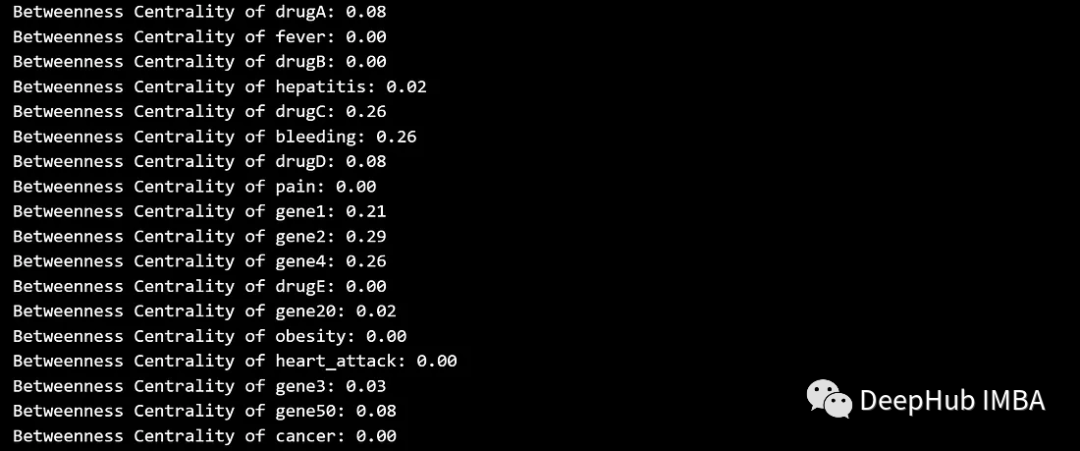
La centralité intermédiaire mesure la fréquence à laquelle un nœud se trouve sur le chemin le plus court entre d'autres nœuds, ou mesure l'influence d'un nœud sur le flux d'informations entre d'autres nœuds. Les nœuds avec un niveau élevé d’intermédiation peuvent servir de ponts entre différentes parties du graphique.
betweenness_centrality = nx.betweenness_centrality(G)
for node, centrality in betweenness_centrality.items():
print(f'Betweenness Centrality of {node}: {centrality:.2f}')
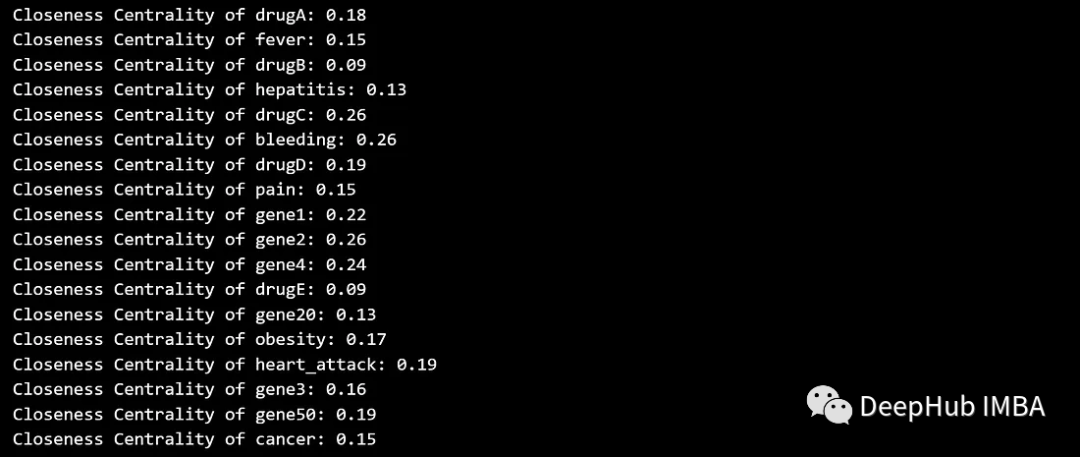
La centralité de proximité quantifie la rapidité avec laquelle un nœud atteint tous les autres nœuds du graphique. Les nœuds ayant une centralité de proximité plus élevée sont considérés comme plus centraux car ils peuvent communiquer plus efficacement avec d’autres nœuds.
closeness_centrality = nx.closeness_centrality(G)
for node, centrality in closeness_centrality.items():
print(f'Closeness Centrality of {node}: {centrality:.2f}')
visualisation
# Calculate centrality measures
degree_centrality = nx.degree_centrality(G)
betweenness_centrality = nx.betweenness_centrality(G)
closeness_centrality = nx.closeness_centrality(G)
# Visualize centrality measures
plt.figure(figsize=(15, 10))
# Degree centrality
plt.subplot(131)
nx.draw(G, pos, with_labels=True, font_size=10, node_size=[v * 3000 for v in degree_centrality.values()], node_color=list(degree_centrality.values()), cmap=plt.cm.Blues, edge_color='gray', alpha=0.6)
plt.title('Degree Centrality')
# Betweenness centrality
plt.subplot(132)
nx.draw(G, pos, with_labels=True, font_size=10, node_size=[v * 3000 for v in betweenness_centrality.values()], node_color=list(betweenness_centrality.values()), cmap=plt.cm.Oranges, edge_color='gray', alpha=0.6)
plt.title('Betweenness Centrality')
# Closeness centrality
plt.subplot(133)
nx.draw(G, pos, with_labels=True, font_size=10, node_size=[v * 3000 for v in closeness_centrality.values()], node_color=list(closeness_centrality.values()), cmap=plt.cm.Greens, edge_color='gray', alpha=0.6)
plt.title('Closeness Centrality')
plt.tight_layout()
plt.show()

2. Analyse du chemin le plus court
L’objectif de l’analyse du chemin le plus court est de trouver le chemin le plus court entre deux nœuds du graphique. Cela peut aider à comprendre la connectivité entre les différentes entités et le nombre minimum de relations requises pour les connecter. Par exemple, supposons que vous souhaitiez trouver le chemin le plus court entre les nœuds « gène2 » et « cancer » :
source_node = 'gene2'
target_node = 'cancer'
# Find the shortest path
shortest_path = nx.shortest_path(G, source=source_node, target=target_node)
# Visualize the shortest path
plt.figure(figsize=(10, 8))
path_edges = [(shortest_path[i], shortest_path[i + 1]) for i in range(len(shortest_path) — 1)]
nx.draw(G, pos, with_labels=True, font_size=10, node_size=700, node_color='lightblue', edge_color='gray', alpha=0.6)
nx.draw_networkx_edges(G, pos, edgelist=path_edges, edge_color='red', width=2)
plt.title(f'Shortest Path from {source_node} to {target_node}')
plt.show()
print('Shortest Path:', shortest_path)

Le chemin le plus court entre le nœud source « gène2 » et le nœud cible « cancer » est surligné en rouge, et les nœuds et les bords de l'ensemble du graphique sont également affichés. Cela peut aider à comprendre le chemin le plus direct entre deux entités et les relations le long de ce chemin.
intégration de graphiques
Les incorporations de graphiques sont des représentations mathématiques de nœuds ou d'arêtes dans un graphique dans un espace vectoriel continu. Ces intégrations capturent les informations structurelles et relationnelles du graphe, nous permettant d'effectuer diverses analyses telles que le calcul de similarité de nœuds et la visualisation dans des espaces de faible dimension.
Nous utiliserons l'algorithme node2vec, qui apprend les plongements en effectuant des marches aléatoires sur le graphe et en optimisant pour préserver la structure de voisinage local des nœuds.
from node2vec import Node2Vec
# Generate node embeddings using node2vec
node2vec = Node2Vec(G, dimensions=64, walk_length=30, num_walks=200, workers=4) # You can adjust these parameters
model = node2vec.fit(window=10, min_count=1, batch_words=4) # Training the model
# Visualize node embeddings using t-SNE
from sklearn.manifold import TSNE
import numpy as np
# Get embeddings for all nodes
embeddings = np.array([model.wv[node] for node in G.nodes()])
# Reduce dimensionality using t-SNE
tsne = TSNE(n_components=2, perplexity=10, n_iter=400)
embeddings_2d = tsne.fit_transform(embeddings)
# Visualize embeddings in 2D space with node labels
plt.figure(figsize=(12, 10))
plt.scatter(embeddings_2d[:, 0], embeddings_2d[:, 1], c='blue', alpha=0.7)
# Add node labels
for i, node in enumerate(G.nodes()):
plt.text(embeddings_2d[i, 0], embeddings_2d[i, 1], node, fontsize=8)
plt.title('Node Embeddings Visualization')
plt.show()
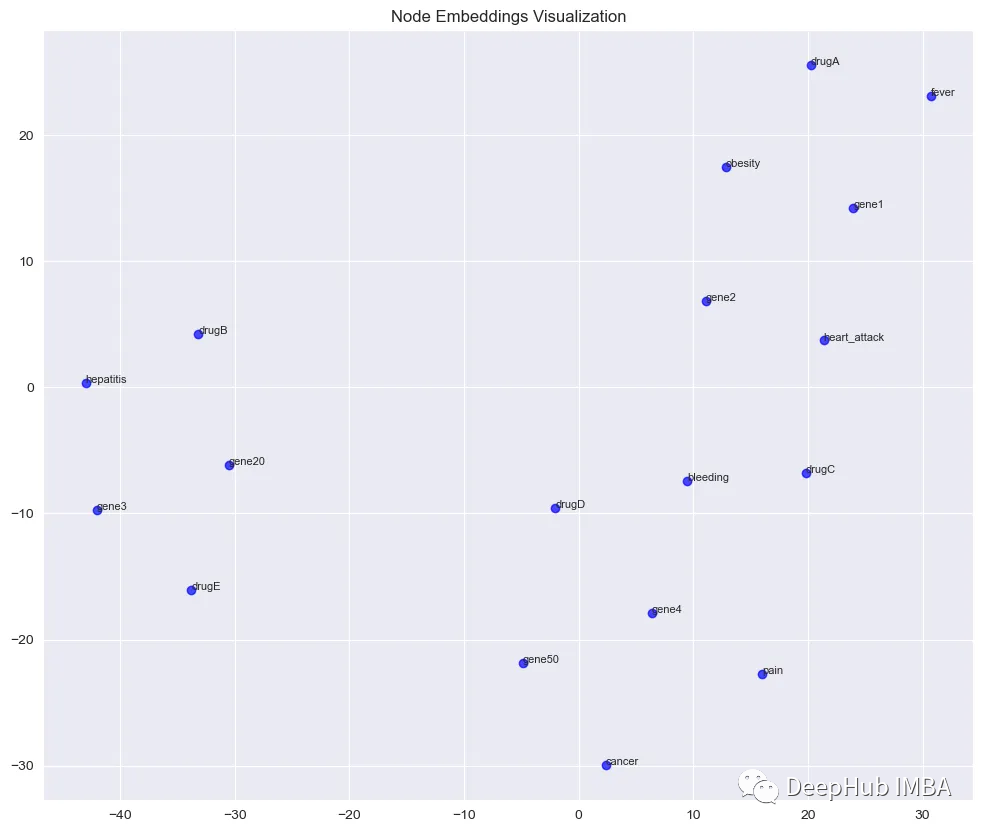
L'algorithme node2vec est utilisé pour apprendre les incorporations de nœuds à 64 dimensions dans KG. Les plongements sont ensuite réduits à 2 dimensions à l'aide de t-SNE. Et visualisez les résultats sous forme de nuage de points. Les sous-graphes déconnectés sont représentables individuellement dans l'espace vectorisé
regroupement
Le clustering est une technique permettant de rechercher des groupes d'observations présentant des caractéristiques similaires. Puisqu'il s'agit d'un algorithme non supervisé, vous n'avez pas besoin d'indiquer spécifiquement à l'algorithme comment regrouper ces observations, et l'algorithme jugera par lui-même, en fonction des données, que les observations (ou points de données) d'un groupe sont plus similaires que d'autres observations. dans un autre groupe.
1、K-signifie
K-means utilise une méthode de raffinement itérative pour générer des clusters finaux basés sur un nombre de clusters défini par l'utilisateur (indiqué par la variable K) et un ensemble de données.
Nous pouvons effectuer un clustering K-means sur l’espace d’intégration. Cela donne une image claire de la façon dont l'algorithme regroupe les nœuds en fonction des intégrations :
# Perform K-Means clustering on node embeddings
num_clusters = 3 # Adjust the number of clusters
kmeans = KMeans(n_clusters=num_clusters, random_state=42)
cluster_labels = kmeans.fit_predict(embeddings)
# Visualize K-Means clustering in the embedding space with node labels
plt.figure(figsize=(12, 10))
plt.scatter(embeddings_2d[:, 0], embeddings_2d[:, 1], c=cluster_labels, cmap=plt.cm.Set1, alpha=0.7)
# Add node labels
for i, node in enumerate(G.nodes()):
plt.text(embeddings_2d[i, 0], embeddings_2d[i, 1], node, fontsize=8)
plt.title('K-Means Clustering in Embedding Space with Node Labels')
plt.colorbar(label=”Cluster Label”)
plt.show()
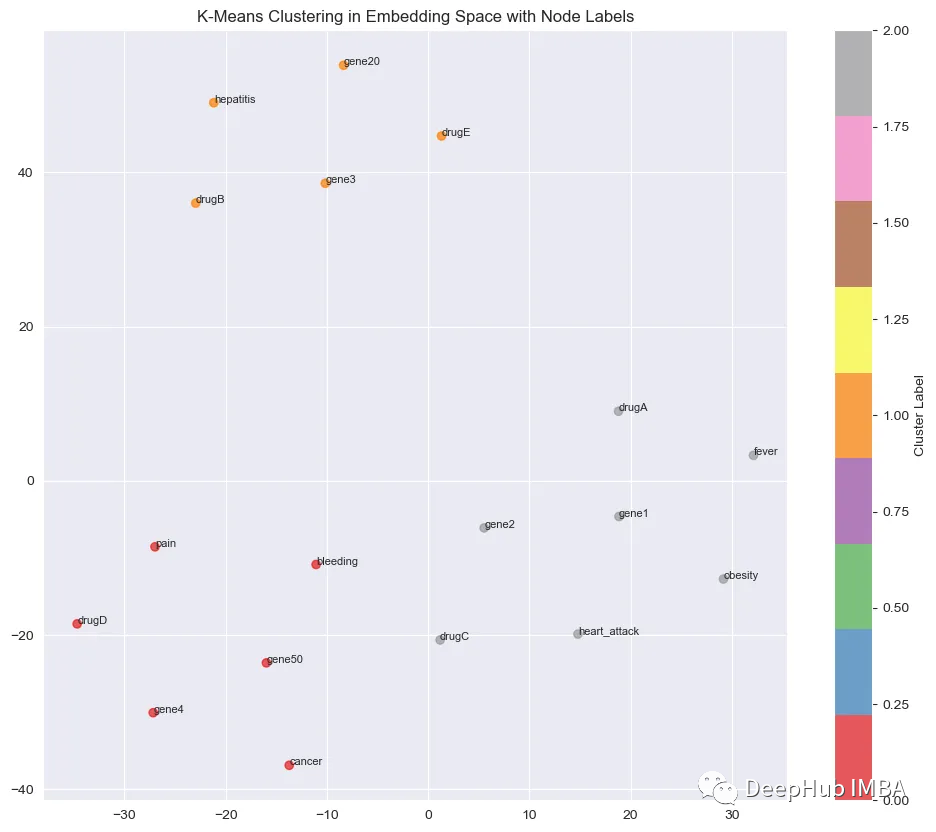
Chaque couleur représente un cluster différent. Revenons maintenant au graphique d'origine et interprétons ces informations dans l'espace d'origine :
from sklearn.cluster import KMeans
# Perform K-Means clustering on node embeddings
num_clusters = 3 # Adjust the number of clusters
kmeans = KMeans(n_clusters=num_clusters, random_state=42)
cluster_labels = kmeans.fit_predict(embeddings)
# Visualize clusters
plt.figure(figsize=(12, 10))
nx.draw(G, pos, with_labels=True, font_size=10, node_size=700, node_color=cluster_labels, cmap=plt.cm.Set1, edge_color=’gray’, alpha=0.6)
plt.title('Graph Clustering using K-Means')
plt.show()
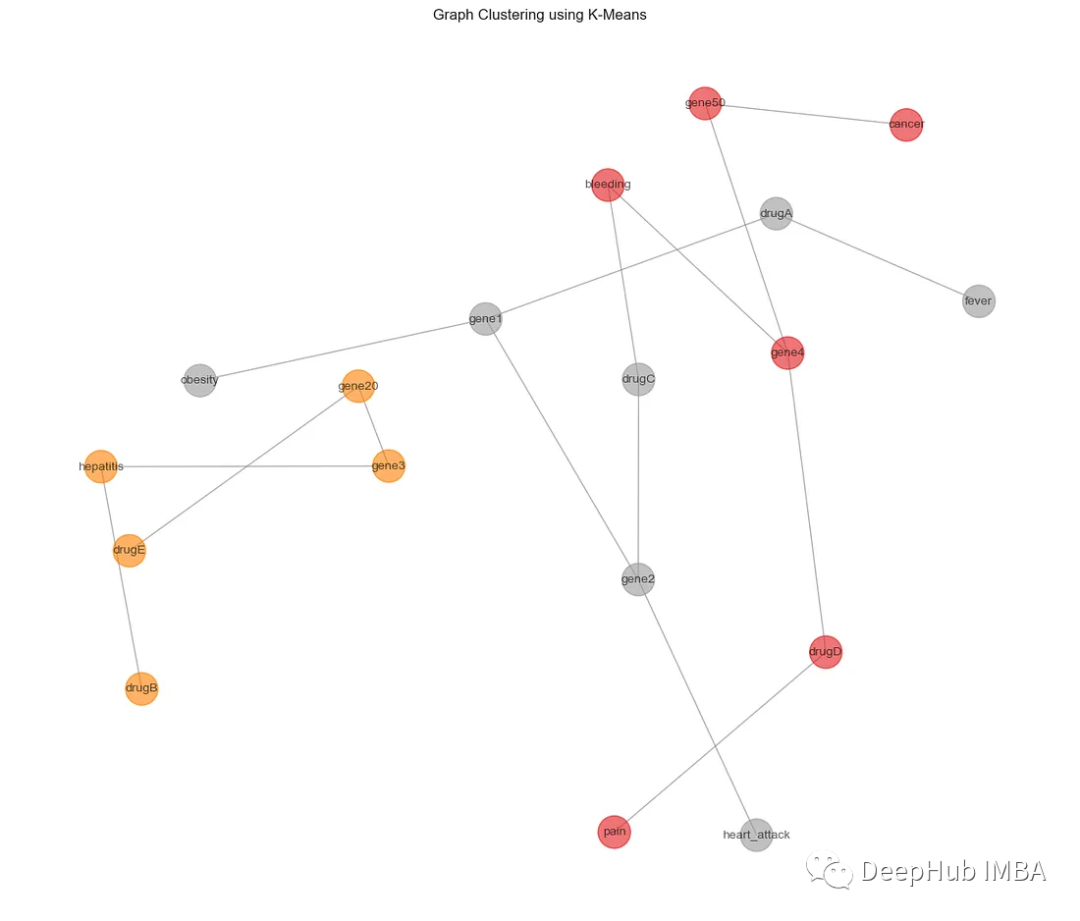
2、DBSCAN
DBSCAN est un algorithme de clustering basé sur la densité et ne nécessite pas un nombre prédéfini de clusters. Il peut également identifier les valeurs aberrantes comme étant du bruit. Vous trouverez ci-dessous un exemple d'utilisation de l'algorithme DBSCAN pour le clustering de graphiques, en se concentrant sur le clustering de nœuds basé sur les intégrations obtenues à partir de l'algorithme node2vec.
from sklearn.cluster import DBSCAN
# Perform DBSCAN clustering on node embeddings
dbscan = DBSCAN(eps=1.0, min_samples=2) # Adjust eps and min_samples
cluster_labels = dbscan.fit_predict(embeddings)
# Visualize clusters
plt.figure(figsize=(12, 10))
nx.draw(G, pos, with_labels=True, font_size=10, node_size=700, node_color=cluster_labels, cmap=plt.cm.Set1, edge_color='gray', alpha=0.6)
plt.title('Graph Clustering using DBSCAN')
plt.show()

Le paramètre eps ci-dessus définit la distance maximale entre deux échantillons et le paramètre min_samples détermine le nombre minimum d'échantillons dans un quartier considéré comme un point central. On peut voir que DBSCAN attribue des nœuds aux clusters et identifie les points bruyants qui n'appartiennent à aucun cluster.
Résumer
L’analyse des KG peut fournir des informations précieuses sur les relations et interactions complexes entre les entités. En combinant le prétraitement des données, les techniques d'analyse, l'intégration et l'analyse de clustering, des modèles cachés peuvent être découverts et des informations plus approfondies sur les structures de données sous-jacentes peuvent être obtenues.
La méthode présentée dans cet article permet de visualiser et d'explorer efficacement les KG, ce qui constitue une connaissance introductive nécessaire à l'apprentissage des graphes de connaissances.
https://avoid.overfit.cn/post/7ec9eb11e66c4b44bd2270b8ad66d80d
Titre : Diego Lopez Yse