algoritmo de agrupamiento k-medias
1. Propósito experimental
- Familiarícese con el algoritmo de agrupamiento de k-medias.
- Escriba un programa para la agrupación en clústeres de k-medias en el conjunto de muestras de entrenamiento, ejecute el algoritmo de agrupación en clústeres de k-medias en datos relacionados con la tarea y depure el experimento.
- Dominar los métodos de cálculo de distancias y los criterios de evaluación de agrupaciones.
- Escribe un informe de laboratorio.
2. Principios experimentales
1. agrupación de k-medias
La agrupación de K-medias es una técnica de partición basada en centroides. Los pasos de cálculo iterativos específicos son los siguientes:
- Las coordenadas del centroide K se generan aleatoriamente en el espacio vectorial de atributos.
- Calcule cada objeto de datos T i ( 1 ≤ i ≤ n ) T_i (1\leq i\leq n) en el conjunto de datos D por separadotyo( 1≤i≤n ) a todoskkMedida de distancia D ist ( i , j ) ( 1 ≤ i ≤ n , 1 ≤ j ≤ k ) de k centroides Dist (i,j) (1\leq i\leq n, 1\leq j\leq k)D i t ( i , _j ) ( 1≤i≤norte ,1≤j≤k ) y convertir el objeto de datosT i T_ityoReúnanse en el grupo con la métrica de distancia mínima. Es decir, T i ∈ CJ T_i\in C_Jtyo∈Cj, representa el objeto de datos T i T_ityoReunidos a JJEn el grupo J. dondeJ = arg min ( D ist ( i , j ) ) J=\argmin(Dist(i,j))j=ar g _min (dist(i, _ _ ___j ) ) , indicandoJJJ es tal queD ist (i, j) Dist(i,j)D i t ( i , _j ) toma el mínimojjj .
- Calcule las coordenadas del centroide de cada grupo de acuerdo con la definición de centroide para formar la próxima generación kkk coordenadas del centroide.
- Si no se cumple la condición de terminación, vaya a 2) para continuar con la iteración; de lo contrario, finalice.
Entre ellos, el centroide del grupo puede tener diferentes definiciones, por ejemplo, puede ser el valor medio del vector de atributos de los objetos de datos en el grupo (es decir, el centro de gravedad), o puede ser el punto central. , etc.; la medida de distancia también puede tener diferentes definiciones, y las comúnmente utilizadas son distancia euclidiana, distancia de Manhattan (o manzana, manzana), distancia de Minkowski, etc.; la condición de terminación puede ser cuando la reasignación de objetos ya no ocurre, la iteración del programa finaliza.
2. Condiciones de rescisión
La condición de terminación puede ser cualquiera de las siguientes:
- Ningún objeto (o un número mínimo de) se reasigna a diferentes clústeres.
- Ya no hay cambios (o el número mínimo de) centros de cluster.
- La suma de errores al cuadrado es un mínimo local.
3. Contenido y procedimientos experimentales.
1. Contenido experimental
- De acuerdo con los pasos de cálculo del algoritmo de agrupamiento de k-medias, dibuje el diagrama de flujo del programa cuando k = 3;
- El algoritmo de agrupamiento de k-means se implementa mediante la programación del diagrama de flujo del programa k-means;
- Muestre una serie de capturas de pantalla del proceso de agrupación de k-medias en el informe del experimento, que indican la evolución gradual de cada grupo
; - En el informe, señale y explique la selección del centroide inicial, la selección de la condición de terminación y
la selección de la métrica de distancia en el código experimental.
2. Pasos experimentales
La programación implementa las siguientes funciones:
- Primero, los vectores de atributos en el conjunto de datos D = {D1, D2, D3} se ingresan como datos experimentales;
- El algoritmo de agrupamiento de k-medias se programa mediante el diagrama de flujo del programa k-medias y se ejecuta con datos experimentales;
- Durante el proceso de ejecución, se detiene en el álgebra de iteración apropiada y muestra los resultados de la iteración en tiempo real, como la posición del centro del clúster
, los resultados de la agrupación por distancia al vecino más cercano, etc.;
3. Diagrama de bloques del programa
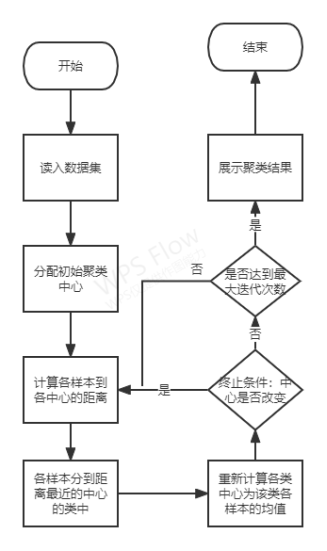
Diagrama de bloques
4. Muestras experimentales
datos.txt
5. Código experimental
#!/usr/bin/env python
# -*- coding: utf-8 -*-
#
# Copyright (C) 2021 #
# @Time : 2022/5/30 21:29
# @Author : Yang Haoyuan
# @Email : [email protected]
# @File : Exp5.py
# @Software: PyCharm
import math
import random
import argparse
import numpy as np
import matplotlib.pyplot as plt
from sklearn.metrics import calinski_harabasz_score
parser = argparse.ArgumentParser(description="Exp5")
parser.add_argument("--epochs", type=int, default=100)
parser.add_argument("--k", type=int, default=3)
parser.add_argument("--n", type=int, default=2)
parser.add_argument("--dataset", type=str, default="data.txt")
parser.set_defaults(augment=True)
args = parser.parse_args()
print(args)
# 读取数据集
def loadDataset(filename):
dataSet = []
with open(filename, 'r') as file_to_read:
while True:
lines = file_to_read.readline() # 整行读取数据
if not lines:
break
p_tmp = [str(i) for i in lines.split(sep="\t")]
p_tmp[len(p_tmp) - 1] = p_tmp[len(p_tmp) - 1].strip("\n")
for i in range(len(p_tmp)):
p_tmp[i] = float(p_tmp[i])
dataSet.append(p_tmp)
return dataSet
# 计算n维数据间的欧式距离
def euclid(p1, p2, n):
distance = 0
for i in range(n):
distance = distance + (p1[i] - p2[i]) ** 2
return math.sqrt(distance)
# 初始化聚类中心
def init_centroids(dataSet, k, n):
_min = dataSet.min(axis=0)
_max = dataSet.max(axis=0)
centre = np.empty((k, n))
for i in range(k):
for j in range(n):
centre[i][j] = random.uniform(_min[j], _max[j])
return centre
# 计算每个数据到每个中心点的欧式距离
def cal_distance(dataSet, centroids, k, n):
dis = np.empty((len(dataSet), k))
for i in range(len(dataSet)):
for j in range(k):
dis[i][j] = euclid(dataSet[i], centroids[j], n)
return dis
# K-Means聚类
def KMeans_Cluster(dataSet, k, n, epochs):
epoch = 0
# 初始化聚类中心
centroids = init_centroids(dataSet, k, n)
# 迭代最多epochs
while epoch < epochs:
# 计算欧式距离
distance = cal_distance(dataSet, centroids, k, n)
classify = []
for i in range(k):
classify.append([])
# 比较距离并分类
for i in range(len(dataSet)):
List = distance[i].tolist()
# 因为初始中心的选取完全随机,所以存在第一次分类,类的数量不足k的情况
# 这里作为异常捕获,也就是distance[i]=nan的时候,证明类的数量不足
# 则再次递归聚类,直到正常为止,返回聚类标签和中心点
try:
index = List.index(distance[i].min())
except:
labels, centroids = KMeans_Cluster(dataSet=np.array(data_set), k=args.k, n=args.n, epochs=args.epochs)
return labels, centroids
classify[index].append(i)
# 构造新的中心点
new_centroids = np.empty((k, n))
for i in range(len(classify)):
for j in range(n):
new_centroids[i][j] = np.sum(dataSet[classify[i]][:, j:j + 1]) / len(classify[i])
# 比较新的中心点和旧的中心点是否一样
if (new_centroids == centroids).all():
# 中心点一样,停止迭代
label_pred = np.empty(len(data_set))
# 返回个样本聚类结果和中心点
for i in range(k):
label_pred[classify[i]] = i
return label_pred, centroids
else:
centroids = new_centroids
epoch = epoch + 1
# 聚类结果展示
def show(label_pred, X, centroids):
x = []
for i in range(args.k):
x.append([])
for k in range(args.k):
for i in range(len(label_pred)):
_l = int(label_pred[i])
x[_l].append(X[i])
for i in range(args.k):
plt.scatter(np.array(x[i])[:, 0], np.array(x[i])[:, 1], color=plt.cm.Set1(i % 8), label='label' + str(i))
plt.scatter(x=centroids[:, 0], y=centroids[:, 1], marker='*', label='pred_center')
plt.legend(loc=3)
plt.show()
if __name__ == "__main__":
# 读取数据
data_set = loadDataset(args.dataset)
# 原始数据展示
plt.scatter(np.array(data_set)[:, :1], np.array(data_set)[:, 1:])
plt.show()
# 获取聚类结果
labels, centroids = KMeans_Cluster(dataSet=np.array(data_set), k=args.k, n=args.n, epochs=args.epochs)
print("Classes: ", labels)
print("Centers: ", centroids)
# 使用Calinski-Harabaz标准评价聚类结果
scores = calinski_harabasz_score(data_set, labels)
print("Scores: ", round(scores, 2))
# 展示聚类结果
show(X=np.array(data_set), label_pred=labels, centroids=centroids)
4. Resultados experimentales
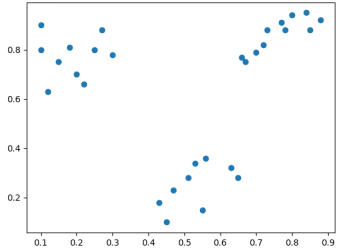
Diagrama de dispersión de distribución de datos original
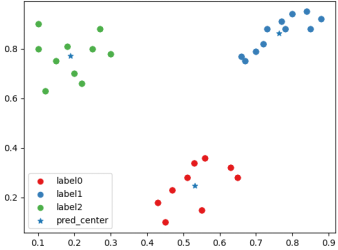
Un diagrama de dispersión de clasificación después de una agrupación "buena" y centros de agrupación.

Un diagrama de dispersión de etiquetas de agrupación, centros de agrupación y puntuaciones del índice CH después de una agrupación "buena"
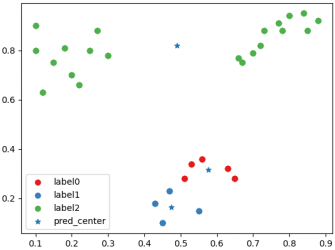
. Un diagrama de dispersión de clasificación después de una agrupación "mala" y centro de agrupación.

A " "malo" etiqueta de agrupación posterior al grupo, centro del grupo y situación de puntuación del índice CH
5. Análisis experimental
Este experimento trata principalmente sobre la implementación del algoritmo K-Means.
Para la distancia entre muestras, adopto la métrica de distancia euclidiana, que es una métrica de similitud y disimilitud que ha demostrado ser muy eficaz en el algoritmo clásico K-Means.
Configuré el centro del clúster para que ya no cambie o el número de iteraciones para alcanzar el límite superior como condición de terminación, dejando de lado algunas situaciones de no convergencia que hacen que el programa se ejecute durante demasiado tiempo.
Dado que la selección inicial del centro de agrupación es completamente aleatoria, puede suceder que el número de clases de agrupación por primera vez sea menor que el k especificado. En este caso, realizo directamente la recursividad en el manejo de excepciones, reinicio la agrupación y volver al agrupamiento recursivo resultado de la clase. Al mismo tiempo, debido a la aleatoriedad de los centros de los conglomerados, incluso para el mismo conjunto de datos, con los mismos parámetros, puede haber enormes lagunas en los resultados de la agrupación. Para juzgar si los resultados de la agrupación son "buenos" o "malos", utilizo Calinski − H arabaz ( CH ) Calinski-Harabaz(CH)C a l i n s k i−El criterio H a r a b a z ( C H ) evalúa los resultados de la agrupación. Este es un algoritmo clásico que se utiliza para evaluar la covarianza entre clases y dentro de ellas, y luego evaluar los resultados de la agrupación. En comparación con el índice Silhouettes, el cálculo del índice CH es más rápido. La fórmula de cálculo es la siguiente:
CH = BGSS k − 1 / WGSS n − k CH=\frac{BGSS}{k-1} / \frac{WGSS}{nk}CH _=k−1B G S S/norte−kW G S S
BGSS se refiere a la covarianza entre clases y WGSS se refiere a la covarianza intraclase. Una buena agrupación debe tener una covarianza interclase grande y una covarianza intraclase pequeña, de modo que el índice CH alcance un máximo local o global. Se puede ver en los resultados experimentales que los resultados de agrupamiento empíricamente mejores tienen puntuaciones de CH más altas.