앞에 쓰다
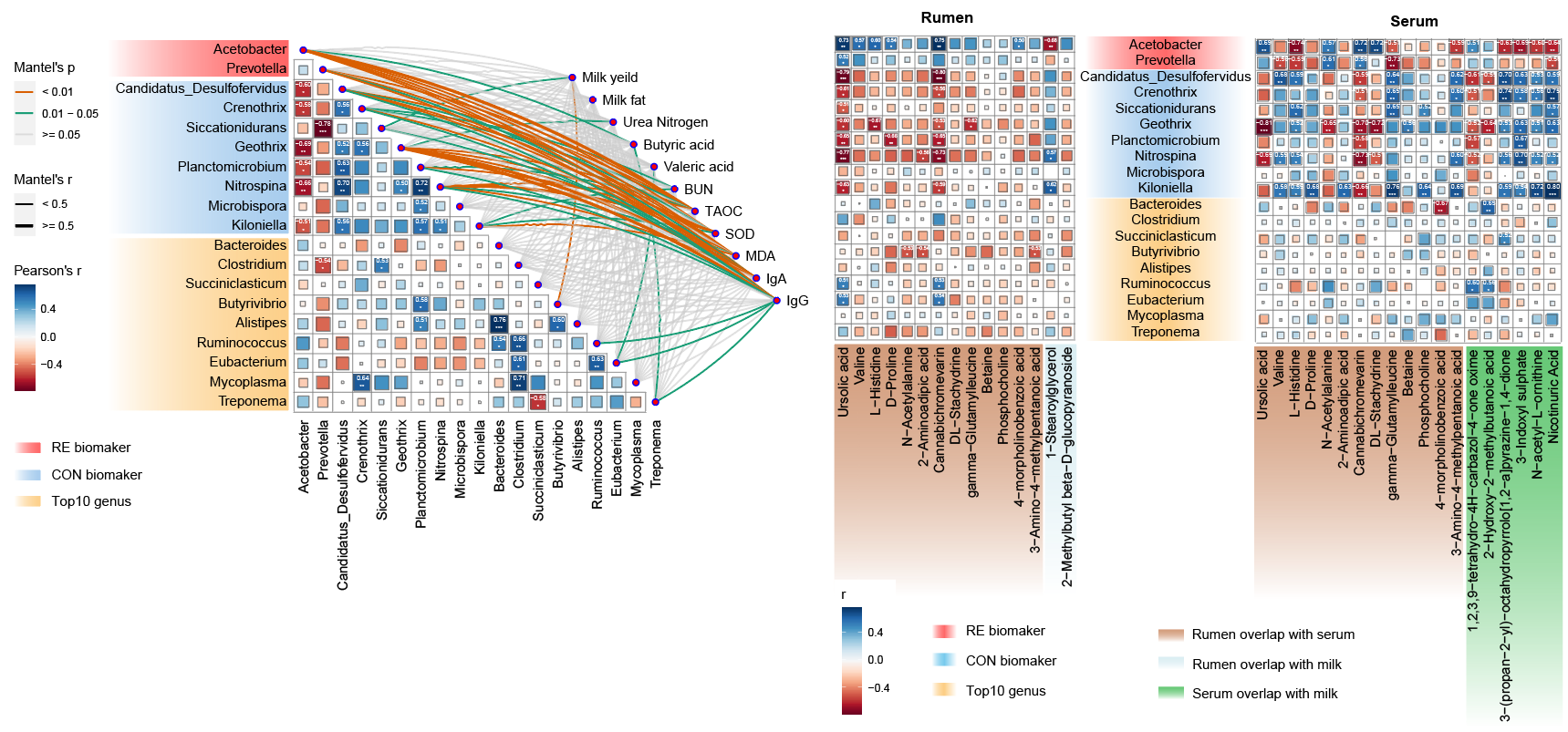
요구사항은 반추위 메타게놈 결과에서 확인된 차등 균주와 겉보기 지표인 반추위 대사체, 혈청 대사체 및 우유 대사체의 서로 다른 부분 간의 상관 분석을 수행하는 것입니다. 렌더링은 다음과 같습니다.
데이터 준비
쉼표로 구분된 csv 형식 파일, 두 개의 테이블, 하나는 각 샘플에 해당하는 명백한 지표 데이터이고 다른 하나는 각 샘플에 해당하는 박테리아 풍부도입니다. 여기서는 속 수준을 사용합니다.
- 연관시켜야 할 모양 데이터
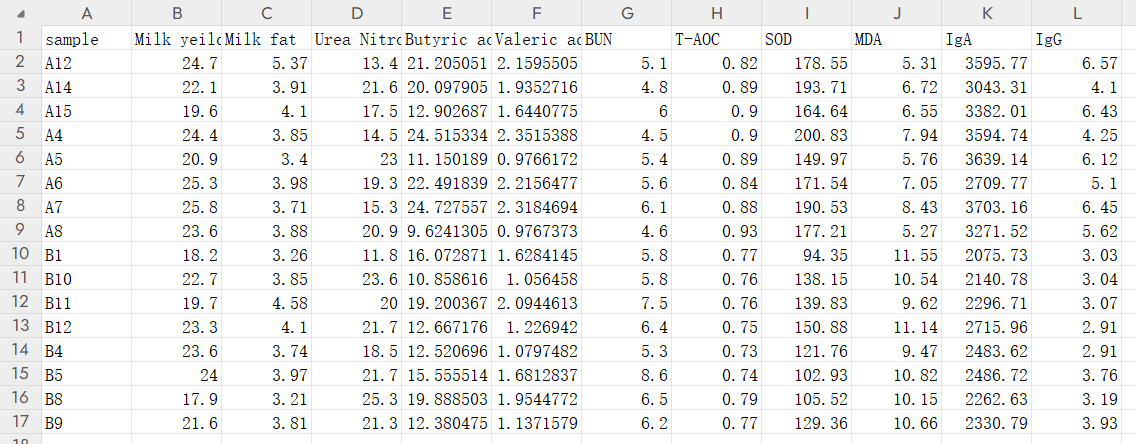
rumen.csv
- 다양한 샘플의 세균 풍부도
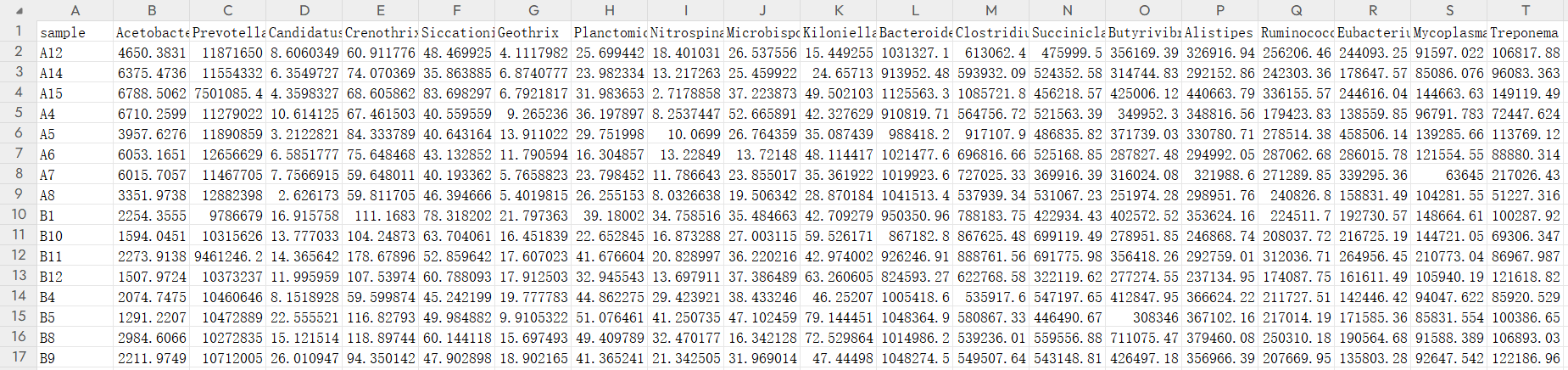
genus.csv
R 패키지 linkET 시각화
- 포장
install.pakages("linkET")
library(linkET)
R 버전에 문제가 있어 설치할 수 없다는 오류가 보고되면(이 오류는 R 4.3.1 버전에서 발생함) 다음을 시도해 보십시오.
install.packages("devtools")
devtools::install_github("Hy4m/linkET", force = TRUE)
packageVersion("linkET")
- 데이터 읽기
library(ggplot2)
rumen <- read.csv("rumen.csv",sep=",",row.name=1,stringsAsFactors = FALSE,check.names = FALSE)
genus <- read.csv("genus.csv",sep=",",row.name=1,stringsAsFactors = FALSE,check.names = FALSE)
#如果报错row.names重复错误请检查数据格式是否为csv
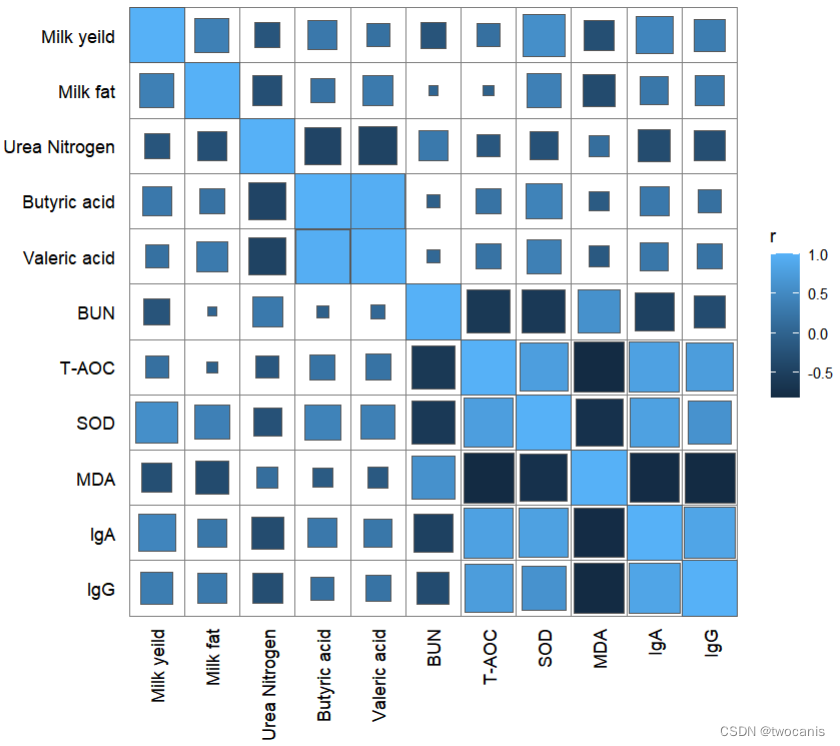
rumen.csv클래스 내 상관 계수
matrix_data(list(rumen = rumen)) %>%
as_md_tbl()
correlate(rumen) %>%
as_matrix_data()
correlate(rumen) %>%
as_md_tbl()
correlate(rumen) %>%
as_md_tbl() %>%
qcorrplot() +
geom_square()
#如果对“%>%”功能报错,装具有此功能的包即可,比如dplyr
library(vegan)
correlate(rumen, genus, method = "spearman") %>%
qcorrplot() +
geom_square() +
geom_mark(sep = '\n',size = 3, sig_level = c(0.05, 0.01, 0.001),
sig_thres = 0.05, color = 'white') + #添加显著性和相关性值
scale_fill_gradientn(colours = RColorBrewer::brewer.pal(11, "RdBu"))
- 두 테이블은 유의성 표시가 있는 상관 행렬 다이어그램을 생성하기 위해 연결됩니다.
library(vegan)
correlate(rumen, genus, method = "spearman") %>%
qcorrplot() +
geom_square() +
geom_mark(sep = '\n',size = 3, sig_level = c(0.05, 0.01, 0.001),
sig_thres = 0.05, color = 'white') + #添加显著性和相关性值
scale_fill_gradientn(colours = RColorBrewer::brewer.pal(11, "RdBu"))
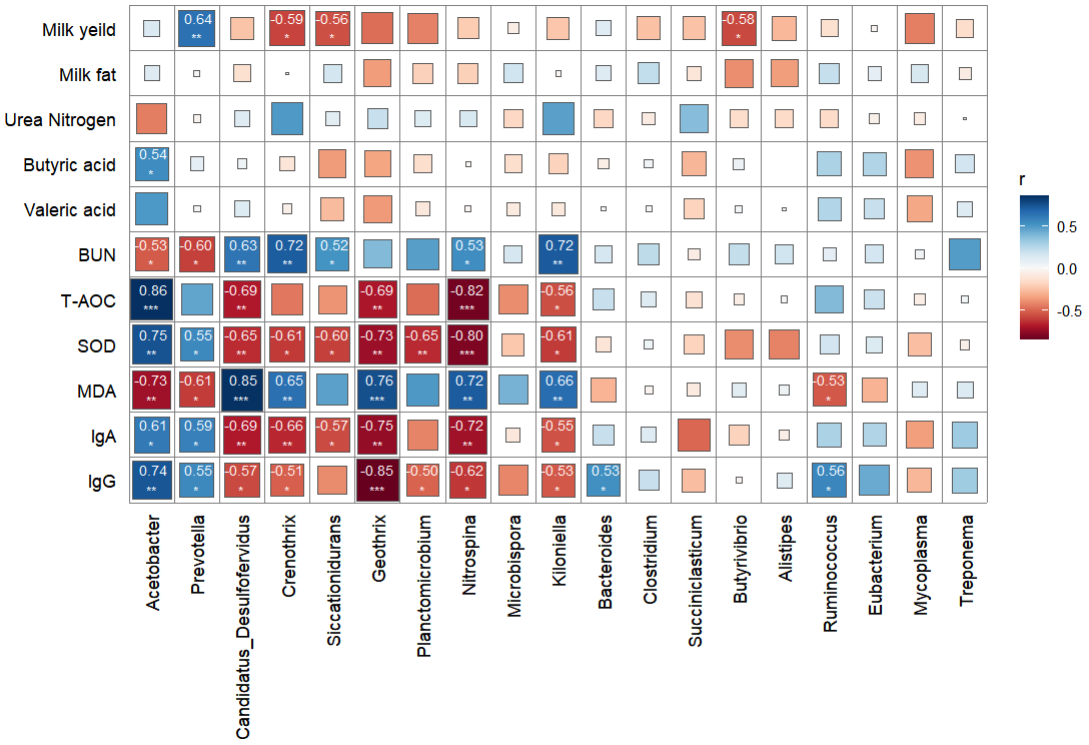
- 시각화 처리
library(dplyr)
mantel <- mantel_test(rumen, genus,
spec_select = list(Milk_yeild=1,Milk_fat=2,Urea_Nitrogen=3,Butyric_acid=4,Valeric_acid=5,BUN=6,
T_AOC=7,SOD=8,MDA=9,IgA=10,IgG=11))%>%
mutate(rd = cut(r, breaks = c(-Inf, 0.5, Inf),
labels = c("< 0.5", ">= 0.5")),
pd = cut(p, breaks = c(-Inf, 0.01, 0.05, Inf),
labels = c("< 0.01", "0.01 - 0.05", ">= 0.05")))
qcorrplot(correlate(genus), type = "lower", diag = FALSE) +
geom_square() +geom_mark(sep = '\n',size = 1.8, sig_level = c(0.05, 0.01, 0.001),
sig_thres = 0.05,color="white") +
geom_couple(aes(colour = pd, size = rd),
data = mantel,
curvature = nice_curvature()) +
scale_fill_gradientn(colours = RColorBrewer::brewer.pal(11, "RdBu")) +
scale_size_manual(values = c(0.5, 1, 2)) +
scale_colour_manual(values = color_pal(3)) +
guides(size = guide_legend(title = "Mantel's r",
override.aes = list(color = "black"),
order = 2),
colour = guide_legend(title = "Mantel's p",
override.aes = list(size = 3),
order = 1),
fill = guide_colorbar(title = "Pearson's r", order = 3))
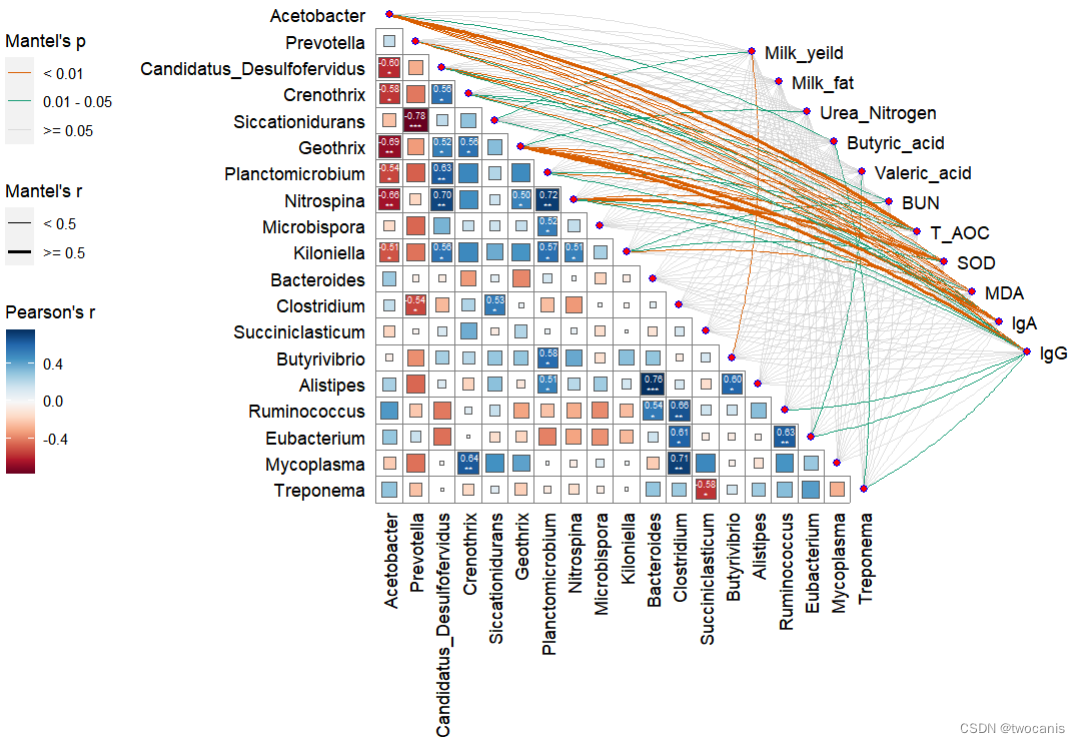
- 눈에 띄지 않는 회색 연결선도 제거하면 사진이 더 깔끔해집니다. 나머지 세부사항은 AI가 처리할 수 있다.