Yuyang proviene de Aofeisi
Qubit | Cuenta pública QbitAI
Cómo las mutaciones genéticas afectan la salud humana sigue siendo en gran medida un misterio.
Pero ahora, los humanos pueden usar el poder de la IA para estudiar este asunto:
Basado en AlphaFold, Google DeepMind entrenó a AlphaMissense específicamente para predecir la patogenicidad de mutaciones sin sentido en el genoma humano .
El artículo fue publicado hoy en Science.

Una "mutación sin sentido" es una sustitución no sinónima en una secuencia de ADN. En pocas palabras, se reemplazan las letras originales (pares de bases) del ADN.
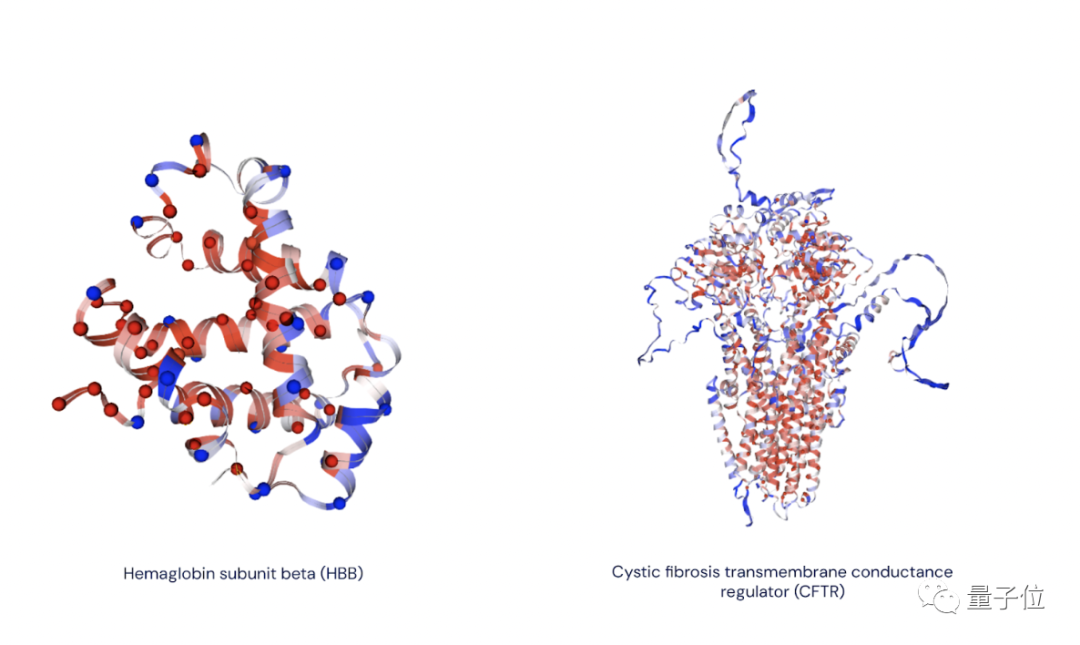
Esto significa que los aminoácidos de las proteínas producidas correspondientes también sufrirán disimilación, lo que puede provocar que la proteína pierda su función original y provoque enfermedades.
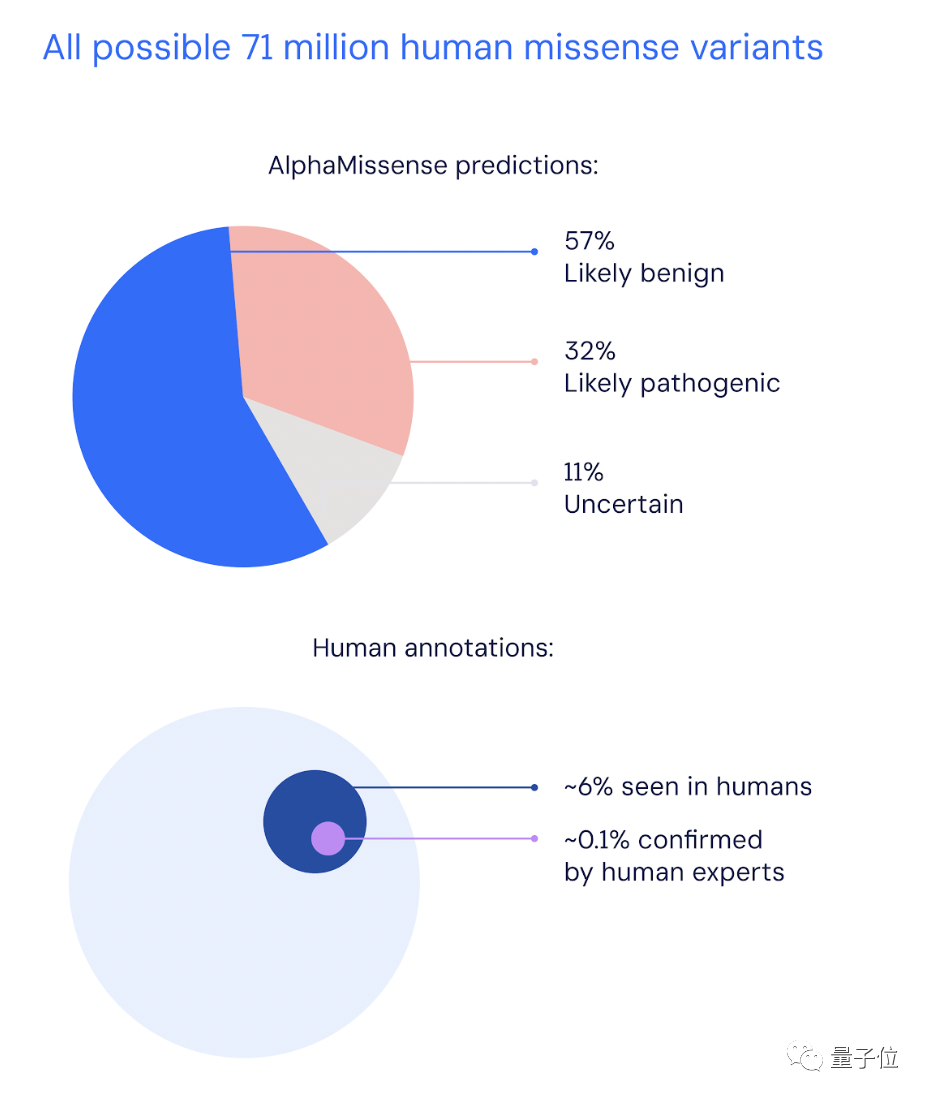
El primer paso de AlphaMissense fue clasificar los 71 millones de posibles mutaciones sin sentido.
Como resultado, la IA clasificó con éxito el 89% de estas variantes como "probablemente patógenas" o "probablemente benignas". En comparación, los expertos humanos alcanzan actualmente el 0,1%.
Uso de IA para predecir la patogenicidad de mutaciones genéticas
En una frase, la principal capacidad de AlphaMissense es predecir si todas las posibles mutaciones sin sentido en el genoma humano son patógenas o benignas.
Cómo hacerlo--
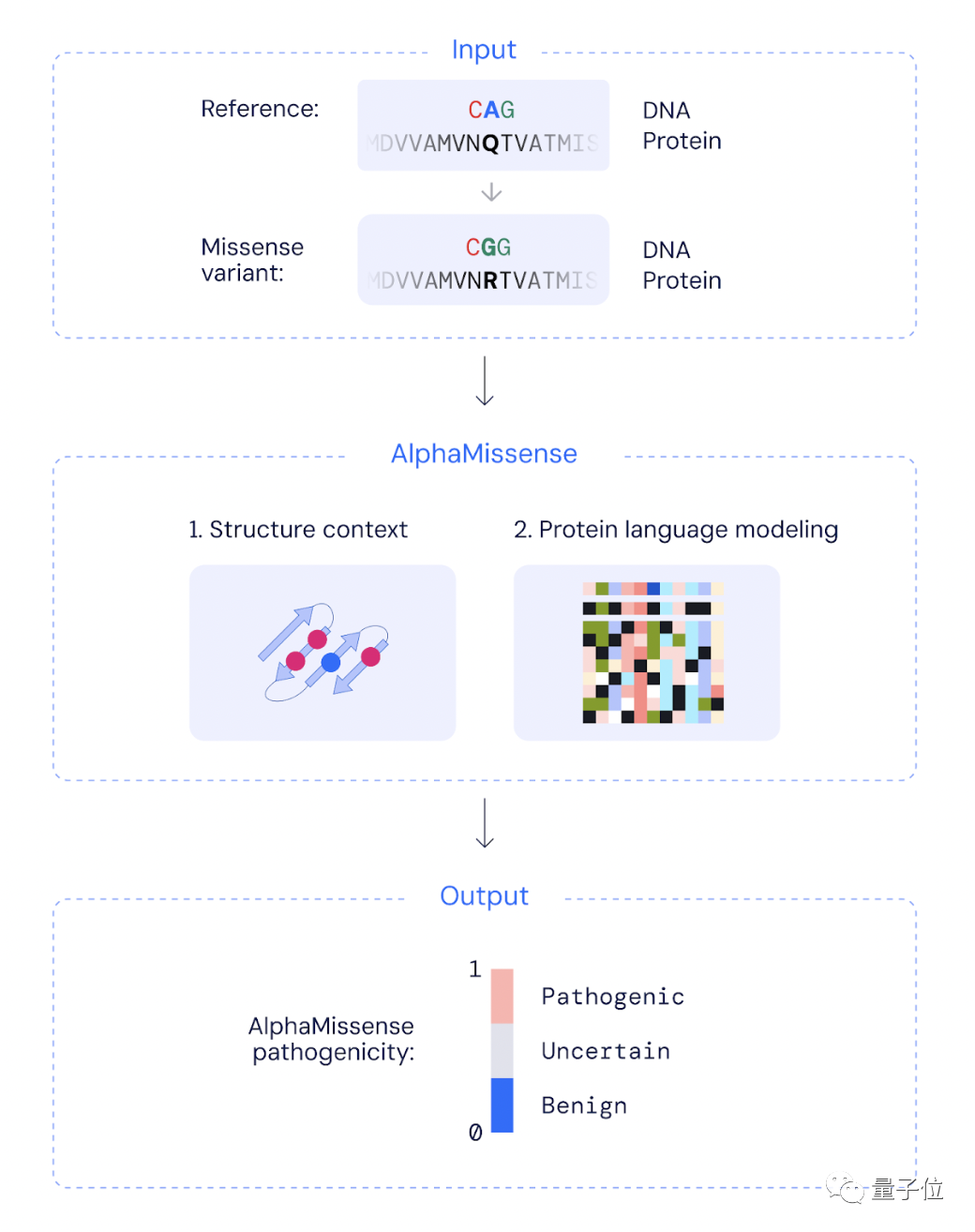
AlphaMissense se basa en el modelo de predicción de la estructura de proteínas AlphaFold de DeepMind.
Los investigadores perfeccionaron AlphaFold utilizando bases de datos de frecuencias de mutaciones humanas y de primates. Específicamente, las variantes comunes en la naturaleza pueden considerarse variantes inofensivas, mientras que las variantes que nunca han aparecido en la base de datos pueden considerarse datos de entrenamiento de "variantes patógenas".
Esta estrategia de formación puede evitar el sesgo causado por la anotación manual.
Vale la pena mencionar que AlphaMissense no puede predecir cambios en la estructura de las proteínas después de mutaciones y otros efectos de las mutaciones sobre la estabilidad de las proteínas.
Después de ingresar una mutación sin sentido, AlphaMissense combinará el contexto de la estructura de la proteína y el modelo de lenguaje de la proteína para darle a la mutación una puntuación de 0 a 1 para determinar aproximadamente si la mutación causará una enfermedad.
△ Efecto AlphaMissense+AlphaFold
Entonces la pregunta es: ¿es realmente confiable la clasificación de AlphaMissense?
Los investigadores lo comprobaron experimentalmente.
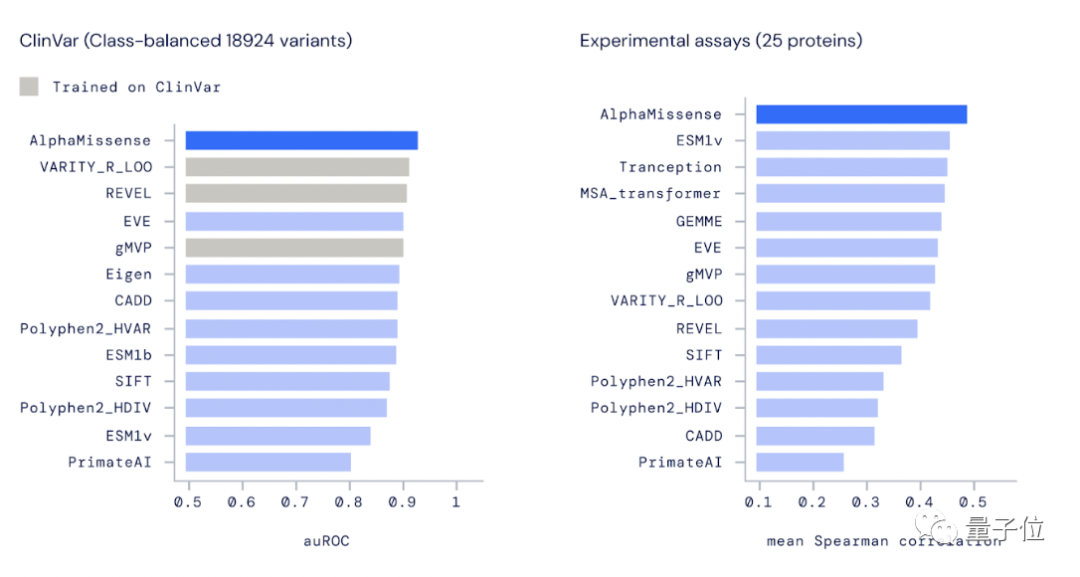
En ClinVar, la base de datos genética autorizada, AlphaMissense ha demostrado un rendimiento de clasificación más potente que otros métodos informáticos.
Entre los 18.924 datos de variantes, el área bajo la curva ROC (auROC) de AlphaMissense alcanzó 0,94. Cuanto más cerca esté este número de 1, con mayor precisión podrá el modelo distinguir entre muestras positivas y negativas.
Vale la pena señalar que en la figura anterior, el método de cálculo que se muestra en gris está entrenado en ClinVar y puede haber un sobreajuste.
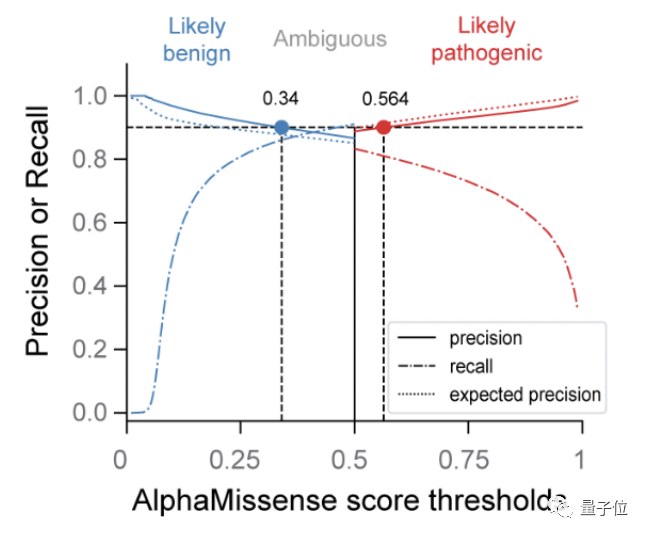
En términos de precisión de predicción, AlphaMissense también alcanzó SOTA. Al ajustar el umbral de clasificación, AlphaMissense puede clasificar "posiblemente patógeno" y "posiblemente benigno" con una precisión esperada del 90%.
DeepMind dijo:
Esperamos que AlphaMissense ayude a resolver preguntas sin respuesta en genómica y ciencias biológicas.
Con este fin, han hecho que los resultados de predicción y el código del modelo de AlphaMissense sean de código abierto.
Además, DeepMind también compartió un conjunto de datos de predicción de todos los posibles 216 millones de sustituciones de secuencias de aminoácidos individuales en más de 19.000 proteínas humanas.
Enlaces de referencia:
[1] Dirección del artículo: https://www.science.org/doi/10.1126/science.adg7492
[2] https://www.deepmind.com/blog/alphamissense-catalogue-of-genetic-mutations -para-ayudar-a-identificar-la-causa-de-las-enfermedades
[3] https://github.com/deepmind/alphamissense