En este artículo explicamos cómo construir KG, analizarlo y crear modelos integrados.

Construir gráfico de conocimiento
Cargue nuestros datos. En este artículo crearemos un KG sencillo desde cero.
import pandas as pd
# Define the heads, relations, and tails
head = ['drugA', 'drugB', 'drugC', 'drugD', 'drugA', 'drugC', 'drugD', 'drugE', 'gene1', 'gene2','gene3', 'gene4', 'gene50', 'gene2', 'gene3', 'gene4']
relation = ['treats', 'treats', 'treats', 'treats', 'inhibits', 'inhibits', 'inhibits', 'inhibits', 'associated', 'associated', 'associated', 'associated', 'associated', 'interacts', 'interacts', 'interacts']
tail = ['fever', 'hepatitis', 'bleeding', 'pain', 'gene1', 'gene2', 'gene4', 'gene20', 'obesity', 'heart_attack', 'hepatitis', 'bleeding', 'cancer', 'gene1', 'gene20', 'gene50']
# Create a dataframe
df = pd.DataFrame({'head': head, 'relation': relation, 'tail': tail})
df

A continuación, cree un gráfico NetworkX (G) para representar KG. Cada fila en DataFrame (df) corresponde a un triplete (cabeza, relación, cola) en KG. La función add_edge agrega bordes entre entidades de cabeza y cola, con relaciones como etiquetas.
import networkx as nx
import matplotlib.pyplot as plt
# Create a knowledge graph
G = nx.Graph()
for _, row in df.iterrows():
G.add_edge(row['head'], row['tail'], label=row['relation'])
Luego, traza los nodos (entidades) y los bordes (relaciones) junto con sus etiquetas.
# Visualize the knowledge graph
pos = nx.spring_layout(G, seed=42, k=0.9)
labels = nx.get_edge_attributes(G, 'label')
plt.figure(figsize=(12, 10))
nx.draw(G, pos, with_labels=True, font_size=10, node_size=700, node_color='lightblue', edge_color='gray', alpha=0.6)
nx.draw_networkx_edge_labels(G, pos, edge_labels=labels, font_size=8, label_pos=0.3, verticalalignment='baseline')
plt.title('Knowledge Graph')
plt.show()
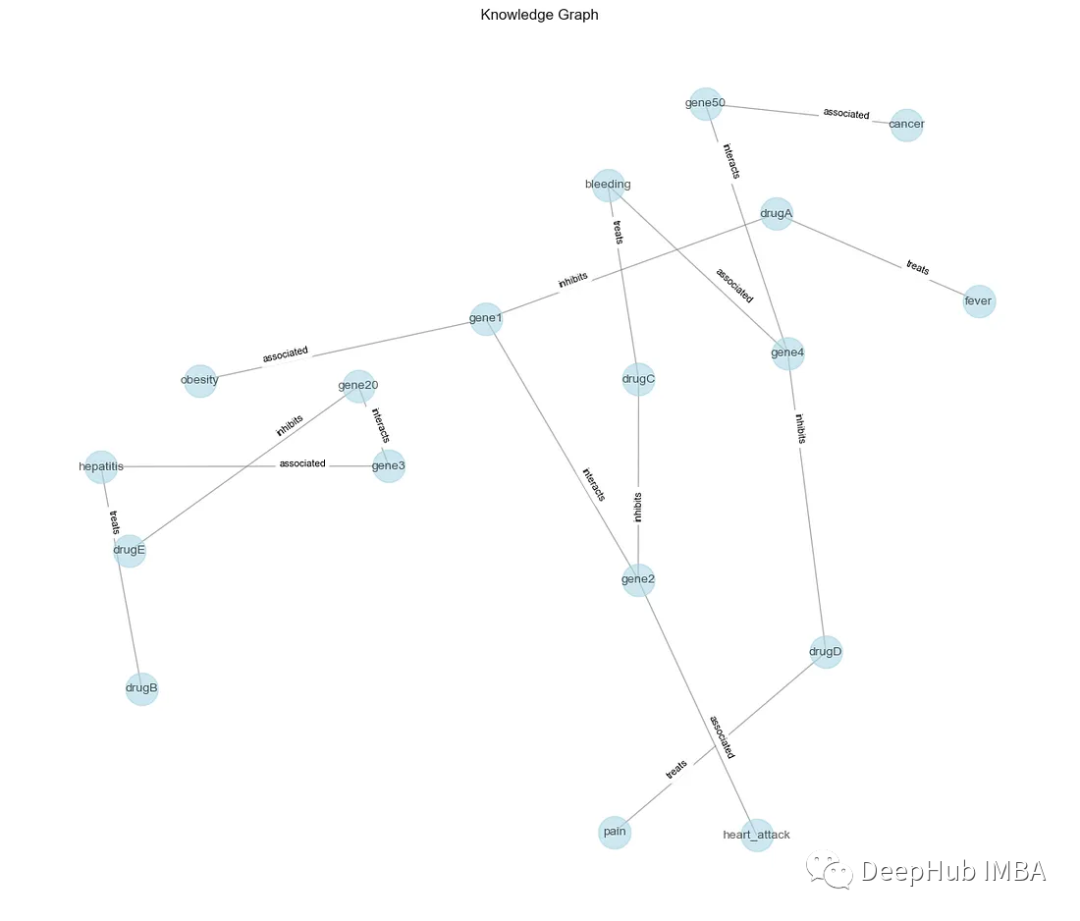
Ahora podemos hacer algunos análisis.
analizar
Para KG, lo primero que podemos hacer es ver cuántos nodos y aristas tiene y analizar la relación entre ellos.
num_nodes = G.number_of_nodes()
num_edges = G.number_of_edges()
print(f'Number of nodes: {num_nodes}')
print(f'Number of edges: {num_edges}')
print(f'Ratio edges to nodes: {round(num_edges / num_nodes, 2)}')

1. Análisis de centralidad de nodos
La centralidad de los nodos mide la importancia o influencia de los nodos en un gráfico. Ayuda a identificar el nodo central de la estructura del gráfico. Algunas de las medidas de centralidad más comunes son:
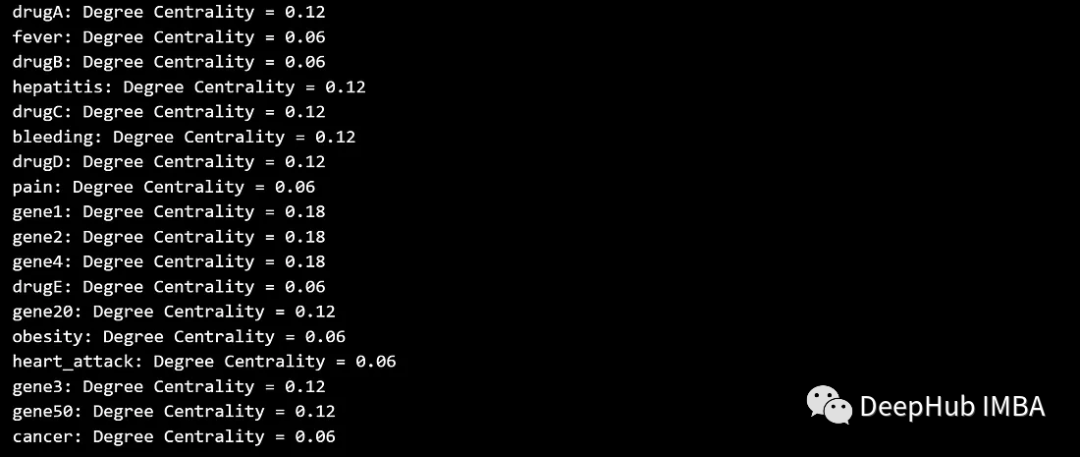
La centralidad de grado cuenta el número de aristas asociadas en un nodo. Los nodos con mayor centralidad están más estrechamente conectados.
degree_centrality = nx.degree_centrality(G)
for node, centrality in degree_centrality.items():
print(f'{node}: Degree Centrality = {centrality:.2f}')
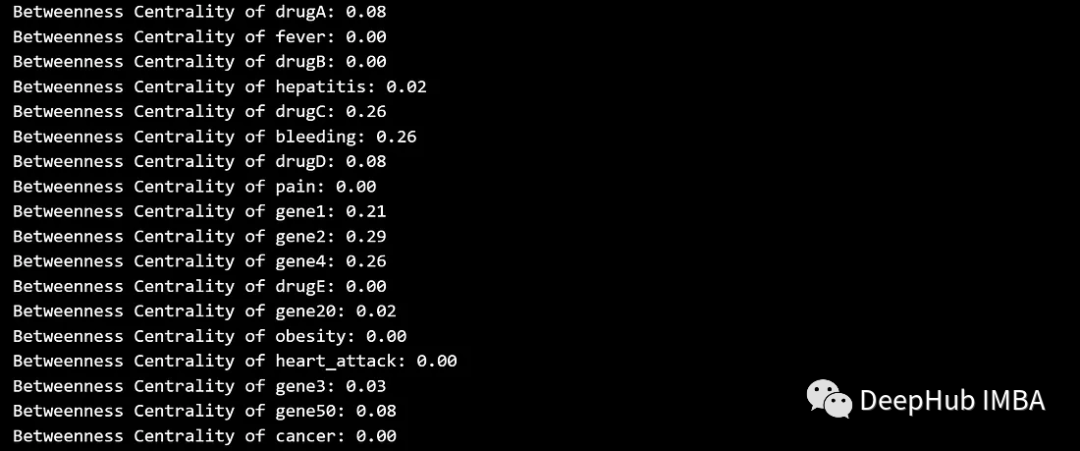
La centralidad de intermediación mide la frecuencia con la que un nodo se ubica en el camino más corto entre otros nodos, o mide la influencia de un nodo en el flujo de información entre otros nodos. Los nodos con alta intermediación pueden actuar como puentes entre diferentes partes del gráfico.
betweenness_centrality = nx.betweenness_centrality(G)
for node, centrality in betweenness_centrality.items():
print(f'Betweenness Centrality of {node}: {centrality:.2f}')
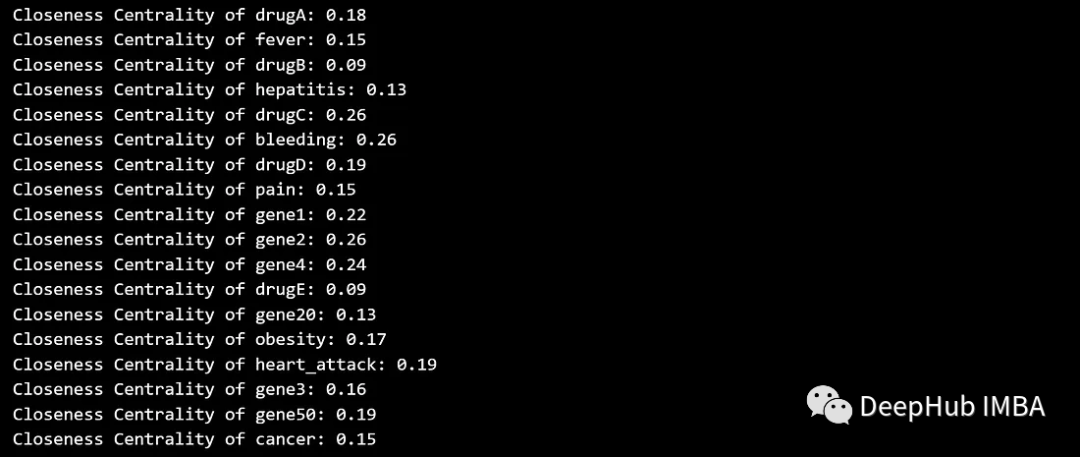
La centralidad de cercanía cuantifica la rapidez con la que un nodo llega a todos los demás nodos del gráfico. Los nodos con mayor centralidad de proximidad se consideran más centrales porque pueden comunicarse con otros nodos de manera más eficiente.
closeness_centrality = nx.closeness_centrality(G)
for node, centrality in closeness_centrality.items():
print(f'Closeness Centrality of {node}: {centrality:.2f}')
visualización
# Calculate centrality measures
degree_centrality = nx.degree_centrality(G)
betweenness_centrality = nx.betweenness_centrality(G)
closeness_centrality = nx.closeness_centrality(G)
# Visualize centrality measures
plt.figure(figsize=(15, 10))
# Degree centrality
plt.subplot(131)
nx.draw(G, pos, with_labels=True, font_size=10, node_size=[v * 3000 for v in degree_centrality.values()], node_color=list(degree_centrality.values()), cmap=plt.cm.Blues, edge_color='gray', alpha=0.6)
plt.title('Degree Centrality')
# Betweenness centrality
plt.subplot(132)
nx.draw(G, pos, with_labels=True, font_size=10, node_size=[v * 3000 for v in betweenness_centrality.values()], node_color=list(betweenness_centrality.values()), cmap=plt.cm.Oranges, edge_color='gray', alpha=0.6)
plt.title('Betweenness Centrality')
# Closeness centrality
plt.subplot(133)
nx.draw(G, pos, with_labels=True, font_size=10, node_size=[v * 3000 for v in closeness_centrality.values()], node_color=list(closeness_centrality.values()), cmap=plt.cm.Greens, edge_color='gray', alpha=0.6)
plt.title('Closeness Centrality')
plt.tight_layout()
plt.show()

2. Análisis del camino más corto
El objetivo del análisis del camino más corto es encontrar el camino más corto entre dos nodos en el gráfico. Esto puede ayudar a comprender la conectividad entre diferentes entidades y la cantidad mínima de relaciones necesarias para conectarlas. Por ejemplo, supongamos que desea encontrar el camino más corto entre los nodos "gene2" y "cancer":
source_node = 'gene2'
target_node = 'cancer'
# Find the shortest path
shortest_path = nx.shortest_path(G, source=source_node, target=target_node)
# Visualize the shortest path
plt.figure(figsize=(10, 8))
path_edges = [(shortest_path[i], shortest_path[i + 1]) for i in range(len(shortest_path) — 1)]
nx.draw(G, pos, with_labels=True, font_size=10, node_size=700, node_color='lightblue', edge_color='gray', alpha=0.6)
nx.draw_networkx_edges(G, pos, edgelist=path_edges, edge_color='red', width=2)
plt.title(f'Shortest Path from {source_node} to {target_node}')
plt.show()
print('Shortest Path:', shortest_path)

La ruta más corta entre el nodo de origen "gen2" y el nodo de destino "cáncer" está resaltada en rojo, y también se muestran los nodos y bordes de todo el gráfico. Esto puede ayudar a comprender el camino más directo entre dos entidades y las relaciones a lo largo de ese camino.
incrustación de gráficos
Las incrustaciones de gráficos son representaciones matemáticas de nodos o aristas en un gráfico en un espacio vectorial continuo. Estas incrustaciones capturan la información estructural y relacional del gráfico, lo que nos permite realizar varios análisis, como el cálculo de similitud de nodos y la visualización en espacios de baja dimensión.
Usaremos el algoritmo node2vec, que aprende incrustaciones realizando recorridos aleatorios en el gráfico y optimizando para preservar la estructura de vecindad local de los nodos.
from node2vec import Node2Vec
# Generate node embeddings using node2vec
node2vec = Node2Vec(G, dimensions=64, walk_length=30, num_walks=200, workers=4) # You can adjust these parameters
model = node2vec.fit(window=10, min_count=1, batch_words=4) # Training the model
# Visualize node embeddings using t-SNE
from sklearn.manifold import TSNE
import numpy as np
# Get embeddings for all nodes
embeddings = np.array([model.wv[node] for node in G.nodes()])
# Reduce dimensionality using t-SNE
tsne = TSNE(n_components=2, perplexity=10, n_iter=400)
embeddings_2d = tsne.fit_transform(embeddings)
# Visualize embeddings in 2D space with node labels
plt.figure(figsize=(12, 10))
plt.scatter(embeddings_2d[:, 0], embeddings_2d[:, 1], c='blue', alpha=0.7)
# Add node labels
for i, node in enumerate(G.nodes()):
plt.text(embeddings_2d[i, 0], embeddings_2d[i, 1], node, fontsize=8)
plt.title('Node Embeddings Visualization')
plt.show()
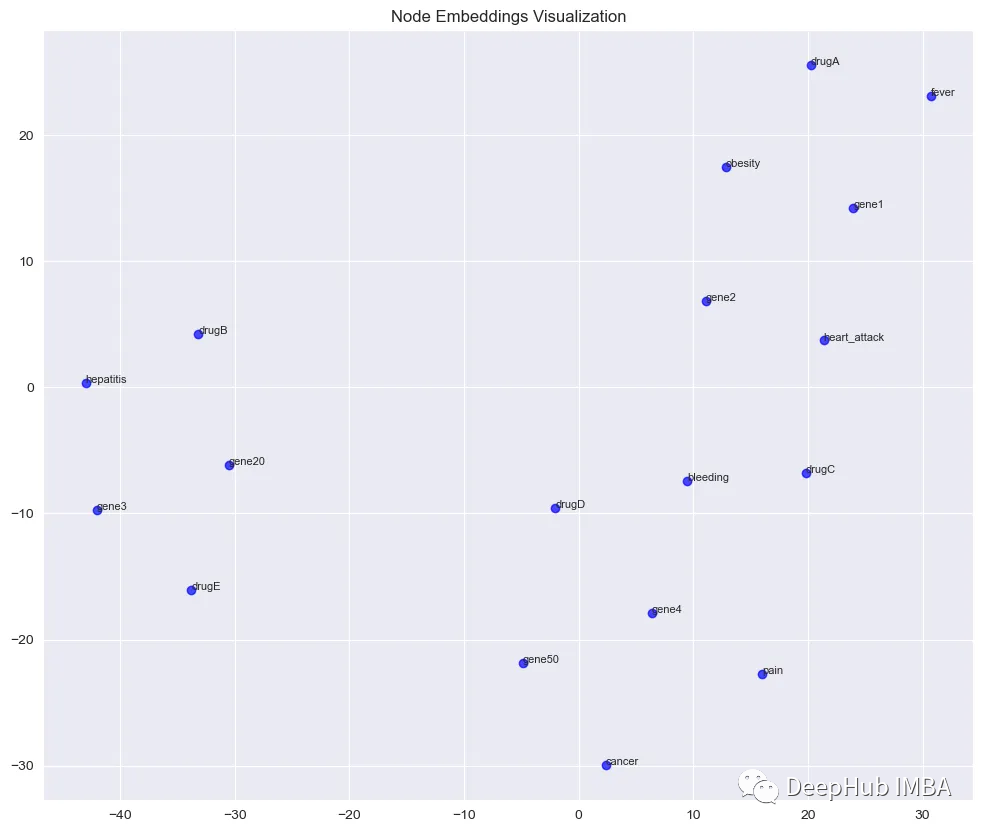
El algoritmo node2vec se utiliza para aprender incrustaciones de nodos de 64 dimensiones en KG. Luego, las incrustaciones se reducen a 2 dimensiones utilizando t-SNE. Y visualice los resultados como un diagrama de dispersión. Los subgrafos desconectados se pueden representar individualmente en el espacio vectorizado.
agrupamiento
La agrupación es una técnica para encontrar grupos de observaciones con características similares. Debido a que es un algoritmo no supervisado, no es necesario decirle al algoritmo específicamente cómo agrupar estas observaciones, y el algoritmo juzgará por sí mismo, basándose en los datos, que las observaciones (o puntos de datos) en un grupo son más similares que otras observaciones. en otro grupo.
1、K-significa
K-means utiliza un método de refinamiento iterativo para generar grupos finales en función del número de grupos definido por el usuario (indicado por la variable K) y el conjunto de datos.
Podemos realizar agrupaciones de K-medias en el espacio de incrustación. Esto proporciona una imagen clara de cómo el algoritmo agrupa los nodos según las incrustaciones:
# Perform K-Means clustering on node embeddings
num_clusters = 3 # Adjust the number of clusters
kmeans = KMeans(n_clusters=num_clusters, random_state=42)
cluster_labels = kmeans.fit_predict(embeddings)
# Visualize K-Means clustering in the embedding space with node labels
plt.figure(figsize=(12, 10))
plt.scatter(embeddings_2d[:, 0], embeddings_2d[:, 1], c=cluster_labels, cmap=plt.cm.Set1, alpha=0.7)
# Add node labels
for i, node in enumerate(G.nodes()):
plt.text(embeddings_2d[i, 0], embeddings_2d[i, 1], node, fontsize=8)
plt.title('K-Means Clustering in Embedding Space with Node Labels')
plt.colorbar(label=”Cluster Label”)
plt.show()
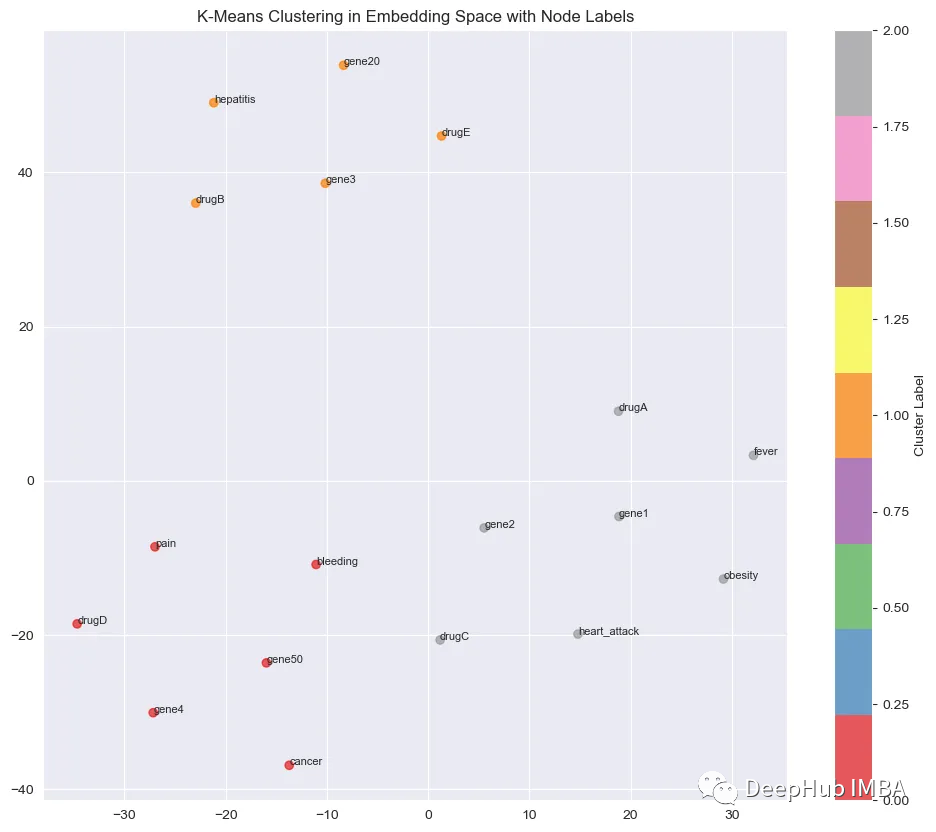
Cada color representa un grupo diferente. Ahora volvemos al gráfico original e interpretamos esta información en el espacio original:
from sklearn.cluster import KMeans
# Perform K-Means clustering on node embeddings
num_clusters = 3 # Adjust the number of clusters
kmeans = KMeans(n_clusters=num_clusters, random_state=42)
cluster_labels = kmeans.fit_predict(embeddings)
# Visualize clusters
plt.figure(figsize=(12, 10))
nx.draw(G, pos, with_labels=True, font_size=10, node_size=700, node_color=cluster_labels, cmap=plt.cm.Set1, edge_color=’gray’, alpha=0.6)
plt.title('Graph Clustering using K-Means')
plt.show()
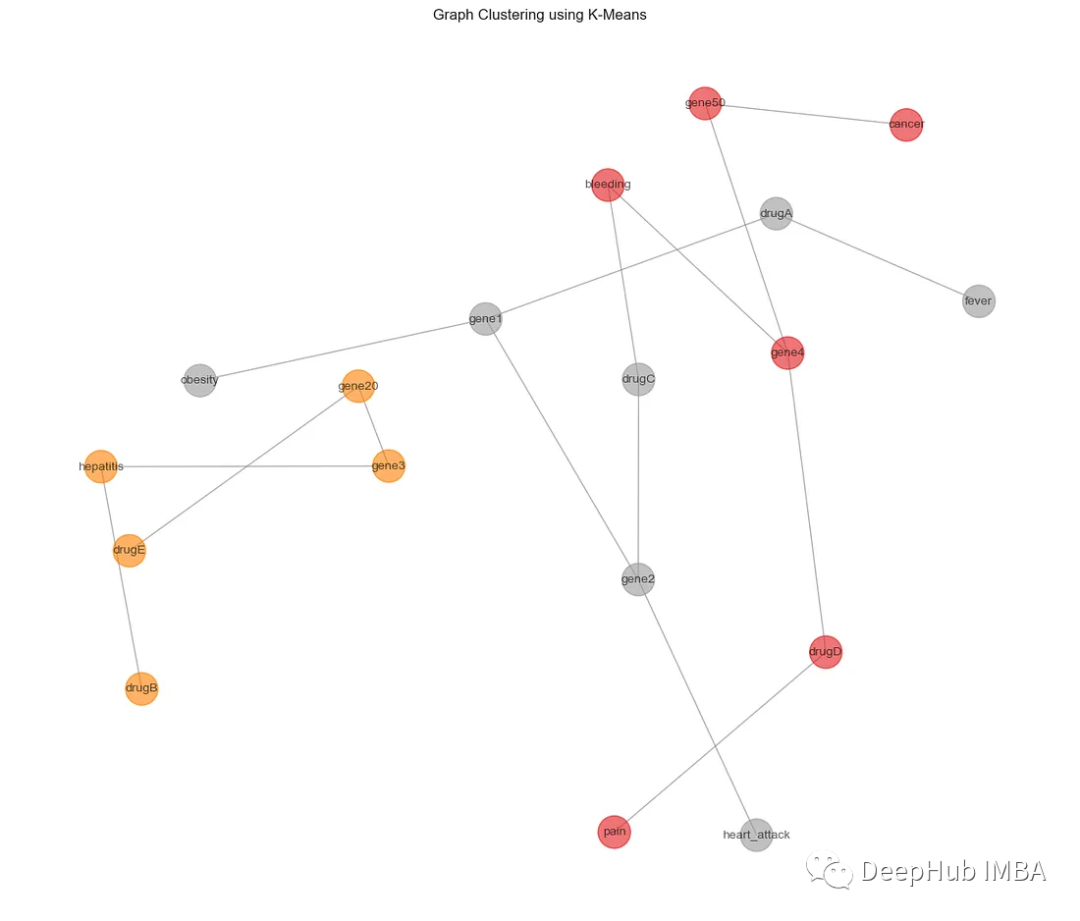
2、DBSCAN
DBSCAN es un algoritmo de agrupación basado en densidad y no requiere un número preestablecido de agrupaciones. También puede identificar valores atípicos como ruido. A continuación se muestra un ejemplo de cómo utilizar el algoritmo DBSCAN para la agrupación de gráficos, centrándose en la agrupación de nodos en función de las incrustaciones obtenidas del algoritmo node2vec.
from sklearn.cluster import DBSCAN
# Perform DBSCAN clustering on node embeddings
dbscan = DBSCAN(eps=1.0, min_samples=2) # Adjust eps and min_samples
cluster_labels = dbscan.fit_predict(embeddings)
# Visualize clusters
plt.figure(figsize=(12, 10))
nx.draw(G, pos, with_labels=True, font_size=10, node_size=700, node_color=cluster_labels, cmap=plt.cm.Set1, edge_color='gray', alpha=0.6)
plt.title('Graph Clustering using DBSCAN')
plt.show()

El parámetro eps anterior define la distancia máxima entre dos muestras y el parámetro min_samples determina la cantidad mínima de muestras dentro de un vecindario que se considera un punto central. Se puede ver que DBSCAN asigna nodos a grupos e identifica puntos ruidosos que no pertenecen a ningún grupo.
Resumir
El análisis de los KG puede proporcionar información valiosa sobre relaciones e interacciones complejas entre entidades. Al combinar el preprocesamiento de datos, técnicas de análisis, incrustación y análisis de agrupación, se pueden descubrir patrones ocultos y se puede obtener una comprensión más profunda de la estructura de datos subyacente.
El método de este artículo puede visualizar y explorar eficazmente los KG, lo cual es un conocimiento introductorio necesario en el aprendizaje de gráficos de conocimiento.
https://avoid.overfit.cn/post/7ec9eb11e66c4b44bd2270b8ad66d80d
Ejemplo: Diego López Ysé.