
개인적인 느낌: 저자 Gaurav Bhardwaj는 David Baker 연구 그룹의 고리형 펩타이드 연구 전문가입니다.그는 기본적으로 대칭형 고리형 펩티드이든 막 투과성 디자인이든 상관없이 로제타 고리형 펩티드의 구조 예측 및 설계 모듈에 참여합니다.매우 좋은. 시각적으로 이 기사는 Cell/Nature와 같은 저널에도 준비되어야 합니다. 부분 컴퓨팅 로제타에서 AI 기반 알파폴드로 전환하는 것은 놀라운 일이다. 여기에 사용된 Rosetta 에너지 풍경과 환각 루프 펩티드 디자인은 모두 이전 작업입니다.이 기사는 이전 작업 재단의 좋은 모음이며 정말 부티크입니다.
题目:AlphaFold를 이용한 순환 펩타이드 구조 예측 및 설계
문서 출처: https://doi.org/10.1101/2023.02.25.529956
代码:구조 예측, 시퀀스 설계 및 환각을 위한 예제 스크립트는 https://github.com/sokrypton/ColabDesign/blob/main/af/examples/af_cyc_design.ipynb에서 다운로드할 수 있습니다. Rosetta 소프트웨어 제품군은 https:// www.rosettacommons.org
콘텐츠:
1. 배경 소개
Alphafold, RoseTTAFold 및 기타 딥 러닝 모델은 최근 몇 년 동안 단백질 예측 분야에서 빛을 발했습니다. 그러나 이러한 모델은 천연 아미노산 구성의 더 큰 아미노산에 대해서만 작동하는 경향이 있습니다. 작은 펩타이드와 펩타이드-단백질 복합체의 구조를 예측하는 데 적용하는 것은 상대적으로 제한적이지만 저자는 이러한 알고리즘을 사용하여 고리형 펩타이드의 구조를 예측해야 한다고 생각합니다. 거대 고리화는 여러 가지 구조적, 안정성 및 투과성 이점을 부여하기 때문에 생체 활성 천연 제품 및 치료 펩타이드 발견 운동에서 일반적입니다. 펩타이드에 자유 말단이 없기 때문에 외부 프로테아제 및 펩티다아제에 대한 내성이 더 강해집니다. 이 종류의 화합물에는 규칙적인 2차 구조가 없지만 고리에는 작은 펩타이드를 안정적으로 접힌 구조로 고정시키는 제약이 있습니다. Rosetta는 오랫동안 고리형 펩타이드의 설계 및 구조 예측에 사용되어 왔지만 골격 샘플링의 높은 계산 비용으로 인해 정확한 고리형 펩티드 구조 예측을 위해서는 계산 비용이 더 낮은 몇 가지 방법을 찾는 것이 필요합니다.
고리형 펩타이드의 고해상도 구조는 덜 구조화되어 있기 때문에 처음부터 고리형 펩타이드에 대한 딥러닝 모델을 훈련시키는 것은 매우 어렵습니다. 대안으로 Rosetta 및 MD(Molecular Dynamics Method)와 같은 방법을 기반으로 모델을 훈련할 수 있습니다. 그러나 이러한 모델의 정확도와 성능은 교육 데이터를 생성하는 데 사용되는 방법의 정확도에 따라 제한됩니다. 실제로 AlphaFold 및 RoseTTAFold와 같은 사전 훈련된 네트워크는 루프 구조를 인식하도록 수정하고 루프 단백질 및 펩타이드 구조를 예측할 때 정확도를 결정하기 위해 벤치마킹할 수 있습니다. 저자는 이전에 KIC 방법(백본 샘플링 방법)을 사용했으며, 루프 펩타이드가 주로 천연 아미노산 모티프와 회전 유형으로 구성되었으며, 이는 더 큰 단백질의 루프 영역에서도 일반적이라는 점에 주목했습니다. 큰 단백질의 결정 구조에서 파생된 Rosetta 에너지 기능은 펩타이드 설계 과정에서 이러한 모티프를 정확하게 포착할 수 있습니다. 또한 최근의 벤치마크 연구에 따르면 AlphaFold는 아포지단백질에서 짧은 펩타이드 리간드의 구조와 결합 상태를 예측할 수 있습니다. 따라서 저자는 시퀀스에 해당하는 루프 구조의 위치 인코딩에 대한 루프 구조 제약 및 불변성을 적절하게 적용할 수 있다면 AlphaFold 네트워크에 인코딩된 정보만으로도 루프 구조를 정확하게 예측하고 설계할 수 있을 것이라고 추론했습니다. 여기에서 저자는 고리화 정보를 인코딩하고 위치를 AlphaFold에 대한 입력으로 인코딩하는 방법을 설명하고 PDB에서 사용할 수 있는 고리형 펩타이드 구조를 예측할 때 이러한 변화의 정확도를 테스트합니다. 다음으로 저자는 AlphaFold를 사용하여 매크로사이클의 백본 시퀀스를 재설계하여 설계된 구조로 접히는 경향을 개선하는 방법을 보고합니다. 마지막으로, 그들은 새로운 순환 펩타이드의 새로운 환각 설계 방법을 설명하고 이를 사용하여 7~13원 순환 펩타이드의 풍부한 다양성을 열거합니다.
2. 고리형 펩타이드 구조 예측
저자는 고리형 펩타이드의 구조 예측에 사용할 수 있도록 상대 위치 인코딩 입력을 수정하여 AlphaFold를 확장하기 시작했습니다. 선형 펩타이드의 경우, 상대 위치 코딩은 인접한 잔기에 대해 1의 서열 분리 및 펩타이드의 길이에 의해 분리된 N-말단 및 C-말단과 함께 아미노산 사이의 서열 분리를 정의합니다(그림 1A). 루프 제약 조건을 적용하기 위해 저자는 상대 위치 인코딩 고리화를 도입하고 길이가 N인 펩티드의 말단 잔기 사이의 서열 분리를 1로 변경하는 맞춤형 N x N 순환 오프셋 매트릭스를 적용합니다(그림 1B). 이 상대 위치 인코딩은 원-핫 인코딩 및 선형 프로젝션 후 AlphaFold 네트워크의 evoformer 모듈의 쌍별 기능에 추가됩니다. 이 인코딩이 없으면 Attention 계층은 순열 및 순서 불변입니다. 저자는 구조 예측 및 설계를 위해 AlphaFold를 지원하는 ColabDesign 프레임워크에서 이러한 변경 사항을 구현했습니다. 저자는 이 새로운 모델을 AfCycDesign이라고 명명했습니다. 저자는 먼저 PDB에서 무작위로 선택한 고리형 펩티드 서열로 이를 테스트했고 이러한 초기 테스트의 결과가 올바른 펩티드 결합 연결 및 말단 잔기 구조를 보여주었지만 모델의 예측이 나머지 펩티드 구조와 일치하지 않는다는 것을 부분적으로 소개합니다. 왜곡. 다음으로 저자는 시퀀스의 링 표현을 입력으로 치환하고 출력 예측이 변경되었는지 여부를 관찰했습니다. 결과는 모든 루프 순열의 출력 구조가 매우 유사하다는 것을 보여주었습니다.

그림 1 AfCycDesign을 사용한 천연 순환 펩타이드 구조의 예측. (A) 가상의 8원 고리형 펩티드에 대한 상대적 위치 인코딩의 예. AfDesign의 표준 코딩은 말단이 서로 가장 먼 선형 펩타이드의 잔기 위치 사이의 서열 분리를 보여줍니다. AfCycDesign에서 순환 오프셋을 적용하면 이 동작을 변경하고 펩티드 사슬의 두 끝을 서로 연결할 수 있습니다. (B) AfCycDesign은 단백질 데이터베이스에서 80개의 고리형 펩티드를 예측했습니다. 강조 표시된 영역은 pLDDT > 0.7 및 RMSD < 1.5Å을 사용하여 우수한 신뢰도와 정확한 예측 결과를 나타냅니다. (C) AfCycDysign의 올바르게 예측된 구조(RMSD<1.5Å, pLDDT>0.85). 실험적으로 결정된 구조는 회색으로 표시되고 AfCycDesign의 예측 구조는 자홍색, 주황색 또는 빨간색으로 표시됩니다. (D) 개별 시퀀스에 대한 순환 오프셋 및 MSA 예측 정확도(실험 구조를 비교하여 얻은 RMSDy)의 비교. (E) 단일 시퀀스에 대한 순환 오프셋이 있는 경우와 없는 경우의 예측 정확도 비교. (F) 순환 오프셋이 있는 경우와 없는 경우의 MSA 예측 정확도 비교.
다음으로 저자는 PDB에서 서로 다른 고리형 펩타이드의 구조를 예측하는 측면에서 AfCycDesign의 정확도를 평가했습니다. 그들은 PDB에서 40개 미만의 서열 길이를 가진 80개의 전형적인 고리형 펩타이드 구조를 수집했습니다. NMR 구조와 길이가 16개 아미노산 미만인 짧은 펩타이드를 제외하도록 모델이 훈련되었기 때문에 이러한 구조 중 어느 것도 AlphaFold 훈련 세트에 없었습니다. 저자가 선택한 구조는 다양한 크기, 2차 구조, 시퀀스 및 기능을 가진 광범위한 토폴로지를 포괄합니다. 특히, 시험 세트에서 상이한 식물 유래 고리형 펩티드 또는 고리형 데스민 접힘과 같은 많은 펩티드는 다수의 시스테인 잔기와 이황화 결합으로 구성된다. 다중 이황화 연결의 조합 - 4개의 시스테인에 대한 3개의 가능한 연결 및 6개의 시스테인에 대한 15개의 연결 - 이황화 연결이 잘 정의되어야 했기 때문에 이전 계산 방식에 대한 도전 과제를 제시했습니다.
AfCycDesign의 평가 지수: 주쇄 중원자 RMSD를 실험적으로 결정된 구조와 비교하고, 구조 예측 신뢰 지수-예측된 로컬 거리 차이(pLDDT). 전반적으로, AfCycDesign의 예측은 각각 0.88 및 1.13Å의 중앙값 pLDDT 및 1.13Å으로 실험적으로 결정된 구조와 밀접하게 일치했습니다(그림 1B). 80개의 테스트 케이스 중 50개의 예측된 구조는 실험 구조에 비해 좋은 신뢰도(pLDDT > 0.7)와 백본 RMSD가 1.5Å 미만이었습니다. 특히, AfCycDesign에 의해 신뢰도가 높은 예측 구조(plDDT > 0.85)로 판단된 49개의 예 중 73%(n = 36)는 백본 중원자 RMSD < 1.5 옹스트롬을 가지고 있어 pLDDT 점수를 예측 변수로 사용할 수 있음을 시사합니다. 고리형 펩타이드. 또한 정확하게 예측된 구조는 특정 펩타이드 또는 토폴로지로 제한되지 않으며 이황화물이 풍부한 루프 펩타이드, 작은 링 β-시트 및 매우 짧은 α-나선 모티프가 있는 펩타이드와 같은 다양한 크기 및 토폴로지를 포함할 수 있습니다(그림 1C). 14가지 경우에서 모델 예측 구조는 실험 구조에 매우 근접했지만(백본 RMSD < 1.5Å) AfCycDesign은 낮은 신뢰도(pLDDT < 0.85)로 이러한 예측 구조에 도달했습니다(그림 1B). 이러한 결과에 대한 낮은 점수는 NMR 데이터 세트에 유연한 루프 영역도 있다는 사실에서 비롯됩니다. 디설파이드 결합에 추가 제약이 부과되지는 않지만, 대부분의 경우 높은 신뢰도의 결합 연결이 데스민, 코-펩티드, 사이클릭 펩티드 및 다른 부류의 디설파이드-풍부 펩티드에 대해 발생한다는 것은 희소식이다. 예측 프로세스에 MSA(Multiple Sequence Alignment)를 추가하면 모델 예측의 정확도를 더욱 향상시킬 수 있으며, 80개 구조 중 58개 사례가 우수한 신뢰성(pLDDT > 0.7)으로 올바르게 예측됩니다(주쇄 중원자 RMSD < 1.5 옹스트롬). 대부분의 구조에서 예측 정확도가 향상되었습니다(그림 1D). 대조적으로, 단일 시퀀스 또는 MSA 기반 예측에서 순환 오프셋을 제거하면 올바른 구조를 예측하는 능력이 크게 감소했습니다(그림 1E-F). 요약하면, AfCycDesign 방법은 서로 다른 고리를 가진 펩타이드의 구조를 예측하는 데 좋은 능력을 가지고 있습니다.
3. 고리형 폴리펩티드의 서열 재설계
다음으로 저자는 AfCycDesign을 사용하여 고리의 상대적 위치 코딩을 사용하여 고리형 펩티드의 아미노산 서열을 디자인했습니다. 그들은 이 접근법이 천연 폴리펩티드에서 유래하거나 다른 백본 샘플링 방법을 사용하여 생성된 주어진 백본의 폴딩 성향을 개선하는 새로운 아미노산 서열의 식별을 용이하게 할 것이라고 추론했습니다. 이를 달성하기 위해 저자는 이전에 구현된 ColabDesign 방법에 순환 오프셋을 도입했습니다. 이 접근법의 목표는 원하는 백본으로 접힐 수 있는 AlphaFold에 의해 예측된 시퀀스를 찾는 것입니다. 이 방법은 먼저 AlphaFold 네트워크를 사용하여 무작위 시퀀스에서 분포 맵을 예측하고 각 후속 단계에서 시퀀스를 반복적으로 최적화하여 해당 단계에서 예측된 구조와 예상 백본 간의 차이를 최소화합니다. 시퀀스 최적화는 예측된 거리 맵(잔기의 각 쌍에 대한 거리 분포를 포함하는 텐서)과 원하는 구조에서 추출된 하나의 구조 사이의 차이(또는 범주형 교차 엔트로피)에 의해 안내됩니다. 이는 AlphaFold의 신뢰도를 극대화하고 예상 구조와 예상 구조 간의 차이를 최소화하는 좋은 방법임이 입증되었습니다.
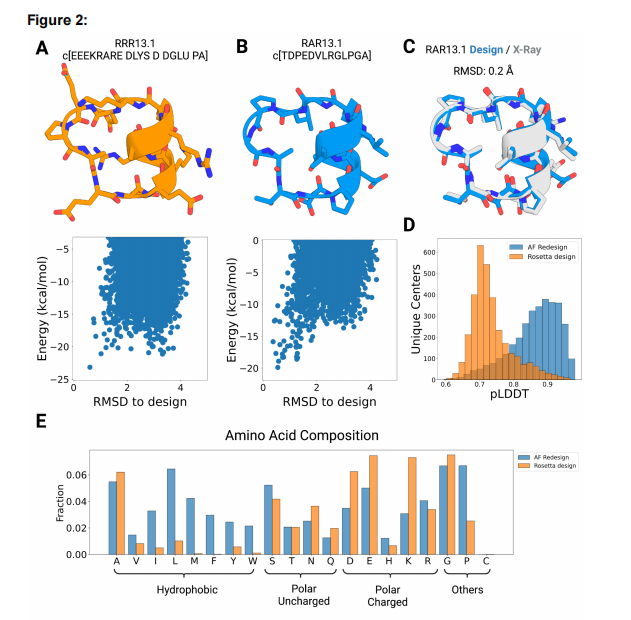
그림 2 AfCycDesign(A) 시퀀스를 사용하여 순환 펩타이드 백본 시퀀스를 설계하고 Rosetta가 설계한 13개 잔기 순환 펩타이드 RRR13.1을 설계합니다. 서열의 1-아미노산은 1문자 코드로 표시되는 반면, d-아미노산은 4문자 코드로 표시됩니다. Rosetta 순환 펩타이드 예측 방법으로 계산된 예측 에너지 환경은 다음과 같습니다. (B) AfCycDesign에 의해 설계된 13개 잔기 고리형 펩티드 RAR13.1의 서열 및 설계 모델. 서열의 1-아미노산은 한 글자 코드로 표시됩니다. Rosetta가 계산한 예상 에너지 환경은 아래 그림과 같습니다. (C) RAR13.1의 설계된 구조(파란색)와 고해상도 X선 결정 구조(회색)의 정렬은 Cα RMSD가 0.2Å인 매우 근접한 일치를 보여줍니다. (D) Rosetta(주황색) 및 AfCycDesign(파란색) 설계 시퀀스에 대한 pLDDT 점수 분포. 각 모집단은 3274개의 고유한 13원 순환 펩티드의 구조적 클러스터를 나타내는 동일한 백본에서 설계되었습니다. 13-루프 펩타이드 구조의 3274개 고유 클러스터 백본에서 Rosetta(주황색) 및 AfcycDesign(파란색)에 의해 디자인된 서열의 아미노산당 빈도.
저자는 단백질-단백질 인터페이스에서 일반적인 나선-나선 상호작용을 표적으로 삼기에 적합한 펩타이드 구조를 설계하기 시작했습니다. 그들은 로제타 고리형 펩티드 설계 방법을 사용하여 13개 고리형 펩티드의 457,615개 백본을 생성했으며, 모두 짧은 7개 아미노산 나선 구조를 포함했습니다. 이 대규모 실행에서 모든 고유한 모양을 식별하기 위해 트위스트 빈 접근 방식을 사용하여 결과 백본을 클러스터링했습니다. 구조를 나타내는 일련의 상자가 생성되고 각 아미노산은 ɸ, ψ 및 ω 트위스트 각도 상자 A와 B는 Ramachandran 그래프의 α 및 β 영역을 나타내고 상자 X와 Y는 그래프의 ɸ, ψ 영역의 미러 영역을 나타냅니다. 이것은 Rosetta 디자인에서 일반적으로 사용되는 방법이기도 합니다. 상자 문자열의 링 순열도 동일한 구조 클러스터로 그룹화됩니다. 저자는 고유한 박스 스트링이 있는 29,249개의 클러스터를 얻었고 Rosetta가 설계한 동일한 메인 체인 시퀀스가 에너지 환경에서 작은 에너지 갭(ΔE < 2 kcal/mol)을 가지고 있기 때문에 재설계를 위해 하나의 메인 체인 RRR13.1(박스 시퀀스: AAAAAAXBYBBAB)을 선택했습니다. ) (그림 2A). 사실, AfCycDesign이 설계한 서열은 Rosetta가 설계한 서열과 크게 다르며, 서열에 12개의 돌연변이가 있고 펩타이드 코어에서 단 하나의 알라닌만 유지되었습니다. 저자는 먼저 AlphaFold가 설계한 시퀀스 RAR13.1의 속성을 평가했습니다. 그들은 Rosetta의 순환 펩타이드 예측 방법을 사용하여 에너지 환경을 계산했습니다. AlphaFold 시퀀스는 설계된 구조와 대체 구조 사이에 더 큰 에너지 갭(ΔE ~ 6.0 kcal/mol)이 있는 가장 낮은 에너지 구조로 설계된 구조로 수렴했습니다(그림 2A,B). Alphafold가 설계한 시퀀스가 원하는 구조로 접힐 수 있는지 확인하기 위해 라세미 고해상도 X선 결정학으로 3차원 구조를 결정하고 이를 컴퓨터로 설계된 형태와 비교했습니다. X선 결정 구조는 Cα RMSD가 0.2Å이고 X선 결정 구조의 13개 측쇄 회전자 중 10개는 설계 모델과 일치했습니다(그림 2C).
Alphafold가 설계한 RAR13.1의 성공적인 구조적 검증을 고려하여 저자는 폴리알라닌(또는 d-알라닌)을 포함하는 대규모 백본 샘플링 실행에서 선택한 3274개의 고유 구조 클러스터에서 대표 펩타이드를 재설계하기로 결정했습니다. 주쇄 Rosetta 에너지는 0kcal/mol 미만. 동시에 저자는 선택한 백본을 Rosetta로 설계하고 이를 AfCycDesign에서 생성한 시퀀스와 비교했습니다. 예상대로 AfCycDesign이 디자인한 시퀀스는 동일한 백본에 대해 Rosetta가 디자인한 시퀀스보다 pLDDT 점수 분포가 더 우수했습니다(그림 2D). Rosetta가 설계한 시퀀스에서는 63개의 클래스만이 pLDDT > 0.9를 가졌고, AfCycDesign 시퀀스에는 pLDDT > 0.9를 가진 1145개의 클래스가 있었습니다(그림 2D). 그러나 저자는 Rosetta의 디자인 접근 방식을 일반적인 20개 아미노산(pLDDT 계산에 필요한 대로)으로 제한해야 했습니다. 이전에는 헤테로키랄 디자인을 수행할 수 있었습니다. 구조 예측 신뢰 메트릭을 비교하는 것 외에도 저자는 AfCycDesign 및 Rosetta가 설계한 서열의 아미노산 구성 및 화학적 특성의 차이를 조사했습니다. AfCycDesign으로 디자인된 시퀀스는 일반적으로 Rosetta가 디자인한 동일한 백본 시퀀스보다 더 소수성이고 더 많은 프롤린을 포함했습니다(그림 2E). 전반적으로 내 실험 결과는 AfCycDesign을 사용하여 원하는 구조로 접을 수 있는 고리형 펩타이드 백본 시퀀스를 설계할 수 있음을 보여줍니다. 보다 광범위하게 AfCycDesign 방법은 다른 펩타이드 백본 생성 방법을 보완하며 이러한 방법과 결합하여 다양한 토폴로지에서 올바르게 접힐 것으로 예상되는 시퀀스를 빠르게 찾을 수 있습니다.
4. 환각 순환 펩타이드의 De Novo 디자인
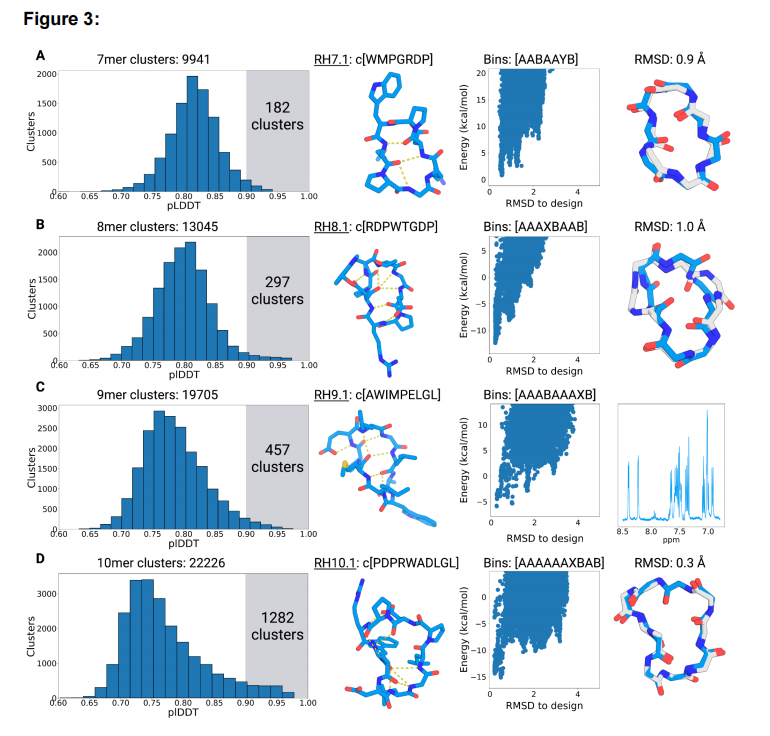
그림 3 AfCycDesign을 사용한 7-10원 환각 루프 펩타이드의 디자인. (A) 7원 고리, (B) 8원 고리, (C) 9원 고리 및 (D) 10-원 고리의 대규모 샘플링에서 구조 예측 신뢰 메트릭(pLDDT) 분포 및 후보 선택 검증 회원 반지. 각 행에 대해 첫 번째 열은 7-10개 잔기의 48000개 잔기의 모든 고유한 구조적 클러스터의 pLDDT 점수 분포를 설명합니다. 각 크기의 클러스터 수는 그래프에 표시됩니다. 각 그림에서 강조 표시된 영역은 pLDDT 점수 >0.9인 클러스터 수를 나타냅니다. 두 번째 열은 구조적 표현에 사용되는 모델의 환각 구조 및 순서를 보여줍니다. 수소 결합은 노란색 점선으로 표시됩니다. 세 번째 열은 선택한 환각 모델에 대해 Rosetta가 계산한 에너지 환경을 보여줍니다. 각 산란점(파란색)은 동일한 설계 시퀀스의 다른 형태를 나타냅니다. 선택한 설계 모델의 트위스트 박스 스트링이 플롯 상단에 표시됩니다. 네 번째 열은 환각 형태(파란색)와 X선 결정 구조(회색) 사이의 정렬을 보여줍니다. RH9.1의 1차원 NMR 스펙트럼을 나타낸다.
다음으로 저자들은 고리형 펩티드에 대한 환각 방법을 개발하여 서열과 구조를 모두 샘플링하고 이를 적용하여 이전의 재설계 방법으로 생성된 13개 펩티드 나선형 백본 외부에 없는 거대 고리형 펩티드를 열거했습니다. 그들은 매우 신뢰할 수 있는 구조적 순환 펩티드로 접힐 것으로 예측되는 서열을 생성하기 위해 3단계 환각 파이프라인을 구현했습니다. 손실에 따라 이 방법은 예측 신뢰 메트릭 pLDDT 및 예측 정렬 오류(PAE)와 분자 내 접촉 수를 개선하려고 시도합니다.
저자는 7~10개의 고리로 구성된 펩타이드로 시작하여 각 크기의 고리에 대해 48,000개의 환각 형태를 열거했습니다. 그런 다음 이러한 대규모 샘플링 작업의 구조를 이전에 설명한 꼬인 상자 기반 클러스터링을 사용하여 클러스터링하고 9941개의 7원 고리형 펩티드, 13405개의 8원 고리형 펩티드, 19705개의 9원 고리형 펩티드 및 22206개의 10원 고리형 펩티드를 확인했습니다. (그림 3A). 모든 독특한 구조 클러스터 중에서, 7-, 8-, 9- 및 10-원 고리형 펩티드는 각각 182, 297, 457 및 1282개의 클러스터를 가졌고, 적어도 하나의 구조가 설계된 구조로 접힐 것으로 예측되었습니다(pLDDT > 0.9 ) (그림 3A) . 기본 구조 예측 및 재설계의 결과를 고려할 때 저자는 0.9 엄격한 신뢰도 메트릭을 통과하는 펩타이드가 설계된 구조로 올바르게 접힐 것으로 예상합니다. 따라서 그들은 Rosetta 순환 펩티드 구조 예측 방법에 의한 추가 검증을 위해 이러한 서열을 선택했습니다. 이러한 시퀀스의 폴딩 성향을 평가하기 위해 Pnear 값(에너지 환경을 설명하는 척도)을 계산했습니다. Pnear 값의 범위는 0에서 1까지이며 값이 1이면 설계된 구조가 시퀀스의 단일 최저 에너지 형태임을 나타냅니다. 이러한 계산에서, 많은 환각 서열은 0.6보다 큰 Pnear 값을 나타내는 114개의 7원 고리형 펩티드, 186개의 8원 고리형 펩티드, 139개의 9원 고리형 펩티드 및 76개의 10원 고리형 펩티드와 함께 좋은 폴딩 성향을 보였다. 저자는 pLDDT > 0.9 및 Pnear > 0.9를 갖는 각 고리형 펩티드 크기에서 환각 유발 디자인 형태를 선택했습니다. 저자는 추가 실험 검증 및 구조적 특성화를 위해 이러한 설계된 순응자를 사용했습니다. 선택된 4개의 디자인된 형태 모두 규칙적인 2차 구조가 없지만 광범위한 분자 내 백본-주쇄 및 백본-측쇄 수소 결합에 의해 안정화됩니다. RH7.1, RH8.1, RH9.1 및 RH10.1의 구조는 각각 3, 5, 5 및 6개의 분자 내 수소 결합입니다(그림 3, 두 번째 열). 선택한 모델의 전체 모양은 알파, 베타 및 감마 회전의 일반적인 조합에 따라 결정됩니다. 구조 RH7.1은 프롤린 잔기에 의해 핵이 생성된 유형 I β 턴과 중첩되는 γ 및 α 턴으로 구성됩니다. 형태 RH8.1은 i+2의 위치 i에서 아스파르트산 잔기의 측쇄에서 주쇄로의 수소 결합에 의해 안정화된 2개의 유형 I β-회전을 포함합니다. 형태 RH9.1은 또한 메티오닌-4와 류신-9 사이의 한 쌍의 장거리 수소 결합에 의해 분리된 두 개의 유형 I β-턴을 포함합니다. 형태 RH9.1의 소수성이 확연히 드러나고 디자인의 유일한 극성 잔류물은 단일 글루탐산입니다. RH10. 1의 서열은 또한 다수의 노출된 비극성 측쇄, 및 분자내 수소 결합 상호작용이 거의 없는 분자를 안정화시키는 트립토판과 류신 사이의 소수성 스태킹을 갖는 특히 소수성이다. 저자는 또한 구조적 제약을 제공하는 프롤린과 Ramachandran 플롯의 X 및 Y 상자에 들어가는 글리신(파이각 >0도)과 함께 선택된 설계된 형태에서 다중 글리신과 프롤린을 관찰합니다.
저자는 네 가지 펩타이드 모두를 결정화하려고 시도했고 7, 8, 10원 고리형 펩타이드의 고해상도 X선 결정 구조를 얻었습니다(그림 3, 네 번째 열). RH7.1의 x-선 구조는 두 구조 사이의 Cα RMSD가 0.9Å인 환각 구조와 밀접하게 일치합니다. 디자인된 형태와 아스파라긴산 측쇄로부터 여분의 수소 결합을 갖는 β-선 결정 구조 사이에는 약간의 차이가 있어 디자인되지 않은 I형 턴을 안정화시킨다. 대조적으로, RH8.1 구조는 1.0Å의 Cα RMSD로 환각 형태에서 더 많이 벗어나고 설계된 형태에서 프롤린-3과 글리신-6의 비틀림은 x-선 결정 구조와 다릅니다. 이 차이는 시퀀스의 단일 글리신 위치에서 가장 두드러지며 ɸ 트위스트가 반전됩니다. RH10.1의 구조는 기본적으로 Cα RMSD가 0.3Å인 환각 형태와 동일합니다. 결정 구조의 측쇄 회전자도 디자인 모델과 매우 잘 일치했으며, 두 개의 류신과 아스파르테이트는 동일했습니다.
저자는 RH9.1의 X선 구조를 얻을 수 없었지만, 이 디자인의 1D NMR은 날카롭고 흩어진 피크를 보여서 접혀 있음을 시사합니다(그림 3C, 네 번째 열). 접힌 상태가 설계 모델과 일치하는지 여부를 결정하려면 추가 구조적 특성화가 필요합니다.

그림 4: AfCycDesign을 사용한 11-13원 환각 루프 펩타이드의 설계. (A) 11 (B) 12 및 (C) 13 사이클릭 펩타이드의 대규모 샘플링에서 선택된 후보의 구조 예측 신뢰 메트릭(pLDDT) 및 검증 분포. 각 행의 첫 번째 열은 48,000개의 11-13원 환각 루프 펩티드에서 식별된 모든 고유한 구조 클러스터에 대한 pLDDT 점수의 분포를 설명합니다. 각 크기에 대한 총 고유 클러스터 수는 그림 제목에 설명되어 있습니다. 각 그림에서 강조 표시된 영역은 pLDDT 점수 >0.9인 클러스터 수를 나타냅니다. 두 번째 열은 구조적 표현에 사용되는 모델의 환각 구조 및 순서를 보여줍니다. 세 번째 열은 선택한 환각 형태에 대한 Rosetta 계산 에너지 환경을 보여줍니다. 각 산란점은 동일한 디자인된 서열의 다른 형태를 나타냅니다. 선택한 설계 모델의 트위스트 박스 스트링이 플롯 상단에 표시됩니다. 네 번째 열은 환각 모델(보라색)과 X선 결정 구조(회색) 사이의 정렬을 보여줍니다.
다음으로, 저자들은 11-13개의 아미노산으로 구성된 거대고리 펩타이드의 환각 설계를 수행했습니다. 큰 구조의 고리형 펩타이드를 설계하는 것은 Rosetta 기반 접근 방식을 시도하는 데 있어서 상당한 도전이며, 이를 안정화하기 위해 추가적인 이황화 결합이 필요한 작업입니다. 저자는 AfCycDesign이 추가 결합 없이 이 크기 범위에서 거대고리 펩타이드를 생성할 수 있는지 궁금했습니다. 11-, 12- 및 13-고리 펩타이드의 경우 저자는 대규모 설계 계산에서 각각 28,457, 27,715 및 27,056개의 고유 구조 클러스터를 식별했습니다(그림 4, 첫 번째 열). 상당한 수의 클러스터가 얻어졌고 이러한 클러스터의 구조는 설계된 구조로 접힐 것으로 예측되었습니다. 11, 12 및 13개의 고리형 펩타이드는 각각 1810, 2855 및 3798개의 클러스터를 가지며 이러한 클러스터 형태의 AlphaFold pLDDT는 > 0.9. 저자는 실험 검증을 위해 pLDDT > 0.9 및 Pnear > 0.9인 시퀀스를 선택했습니다. 더 작은 7-10개 아미노산 설계 형태 대신 저자는 11-13개 아미노산의 3개 디자인에서 전형적인 2차 구조의 짧은 모티프를 선택했습니다(그림 4, 두 번째 열). 설계된 RH11.1은 8개의 잔기 α-나선 모티프를 포함하고 RH12.1 및 RH12.1은 더 짧은 확장된 β-시트를 가집니다. 특히, 서열 길이가 12 및 13인 순환 펩티드(RH12.1 및 RH13.1)는 둘 다 비정형 크기의 베타-루프 사슬을 가지고 있으며, 이 구조는 6, 10 및 14 순환 펩티드에 선호됩니다. 이러한 고리형 펩티드 구조에는 각각 11-, 12- 및 13-고리형 펩티드 디자인 구조에서 9, 7 및 9개의 수소 결합이 있는 광범위한 분자 내 수소 결합도 포함됩니다. RH11.1의 짧은 나선형 모티프는 4개 아미노산의 확장된 루프에 의해 고리화되고 트레오닌 잔기에 의해 매개되는 N-말단 나선형 캡핑 모티프를 가집니다(그림 4A, 두 번째 열). RH12.1은 한쪽 끝에서 가닥을 연결하는 전형적인 유형 II' β-턴과 다른 쪽 끝에서 α-턴이 있는 짧은 β-시트입니다. RH13.1에서는 아스파르트산 측쇄에서 백본 아미드 질소로의 수소 결합으로 인해 가닥 쌍 형성이 이동하여 양쪽 끝이 β 및 α 유형으로 고리화되는 꼬인 β-시트를 생성하고 비극성 사이의 소수성을 통과합니다. 측쇄 상호작용이 더욱 안정화됩니다(그림 4, 두 번째 열).
우리는 고상 화학 펩타이드 합성을 사용하여 RH11.1, RH12.1, RH13.1 및 이들의 미러 이미지를 합성하고 라세미 X선 결정학을 사용하여 세 가지 펩타이드의 구조를 결정했습니다. 이 세 가지 폴리펩티드 단편의 고해상도 결정 구조는 환각 형태와 매우 잘 일치했으며, Cα RMSD는 RH11.1, RH12.1 및 RH13.1에 대해 각각 0.3, 0.4 및 0.8이었습니다(그림 4, 네 번째 열). ). X선 결정 구조의 회전 유형과 수소 결합 패턴도 세 가지 펩타이드 모두의 설계된 형태와 거의 일치했습니다. RH13.1에서 관찰된 더 큰 RMSD는 RH7.1 및 RH8.1에서 이전에 관찰된 역형 또는 플립 아미드 변화보다는 펩타이드 사슬을 따라 전파되는 더 작은 비틀림 편향 때문이었습니다. 디자인의 주요 측쇄 상호작용의 대부분은 X선 구조에서도 관찰됩니다. 두 가지 가장 분명한 편차는 RH11.1의 트립토판이 180° 뒤집혀 있지만 루프는 여전히 디자인에서 생각한 대로 히스티딘으로 채워져 있다는 것입니다. 두 번째는 RH12.1의 티로신이 주쇄와 상호작용하는 대신 아르기닌과 양이온-π 상호작용을 형성한다는 것이다. 종합하면, 이 데이터는 고리형 펩타이드의 새로운 환각에서 AfCycDesign의 높은 정확도를 보여줍니다. 이 접근법은 11~13원 매크로사이클의 설계를 가능하게 하고 이전에 제안된 구조를 안정화하기 위해 추가 이황화 결합을 필요로 하지 않습니다. 보다 광범위하게 여기에 설명된 환각적 접근과 광범위한 구조적 샘플링은 몇 가지 새로운 골격을 제공합니다.
5개 토론
저자는 AlphaFold 네트워크에서 고리화의 상대적 위치를 암호화하고 이를 사용하여 고리형 펩티드 서열의 구조 예측, 자연의 아미노산 재설계 및 고리형 펩티드 백본의 디자인을 포함한 몇 가지 주요 응용 분야를 위한 계산 방법을 개발하는 방법을 보고합니다. 다른 시퀀스, 크기 및 토폴로지 Cyclic Peptide Illusion Design. 저자는 PDB의 고리형 펩티드에 대한 구조 예측을 테스트하여 오프셋이 있는 AlphaFold의 놀라운 정확도를 입증했습니다. 49개의 높은 신뢰도(pLDDT > 0.85) 예 중 73%는 대략 실험 구조와 유사할 수 있습니다(RMSD < 1.5). 따라서 이러한 방식으로 저자는 자연적인 고리형 펩타이드 구조를 관찰하고 올바르게 접힌 구조를 얻기 위해 설계된 펩타이드의 더 나은 필터링을 가능하게 하는 데 주의를 기울였습니다.
저자는 또한 로제타 방법에 의해 생성된 것보다 더 나은 plddt 및 폴딩 경향을 갖는 고리형 펩티드 백본 서열을 재설계하기 위한 계산 방법인 AfCycDesign에 대해 설명합니다. 저자는 AfCycDesign 시퀀스를 Rosetta가 디자인한 시퀀스와 비교하여 AfCycDesign에서 소수성 및 형태 제한 아미노산의 사용 증가를 포함하여 몇 가지 중요한 차이점을 발견했습니다. 저자는 AfCycDesign으로 환각 시퀀스 및 구조 설계를 추가로 수행하여 수만 개의 고유한 7-13원 순환 펩타이드 구조 클러스터 펩타이드를 열거했습니다. 여기에는 디자인된 구조(pLDDT > 0.9)로 접힐 가능성이 매우 높은 것으로 모델에서 간주된 10,681개의 고유한 클러스터가 포함되었습니다. 재설계된 환각 루프 펩타이드의 X선 결정 구조는 방법의 신뢰성을 보여줍니다. 7개의 모든 X선 결정 구조(재설계 1개 및 환각 설계 6개)는 설계된 형태(RMSD가 1.0 미만)에 매우 가깝습니다. 환각 방법이 11-13의 거대 고리 펩타이드를 설계할 수 있다는 점은 주목할 가치가 있습니다. 또한, 저자는 이전에 구조적 고리형 펩티드를 생성하는 데 있어 L- 및 D-아미노산 패턴의 중요성을 언급했지만, 여기에 설명된 환각 펩티드는 이러한 지침을 위반합니다 - 고리형 펩티드: L-아미노산만 있지만 잘 접혀 있습니다. 저자는 d-아미노산 및 기타 비정형 아미노산이 제공하는 프로테아제 및 대사 안정성 이점을 인정하고 이 작업이 디자인 프로세스에서 더 광범위한 화학적 다양성을 통합할 수 있는 심층 학습 네트워크의 향후 개발을 위한 기반을 제공한다고 믿습니다.
환각 펩타이드는 표적 결합 및 막관통과 같은 풍부한 기능을 가진 미러 이미지뿐만 아니라 설계된 구조(pLDDTs > 0.9)로 접힐 것으로 예측됩니다. 결합 상호작용은 단백질-단백질 상호작용의 접목된 모티프 또는 드노보 디자인을 통해 통합될 수 있습니다. 현재와 미래의 노력의 초점은 치료 표적에 대한 환각제 루프 펩티드 접합체에 대한 전산 접근법을 확장하는 것입니다. 지난 5년 동안 딥 러닝 방법은 치료용 단백질 설계에서 엄청난 발전을 가져왔습니다. 여기에 제시된 전산 접근법을 통해 유사한 발전이 높은 치료적 관심의 구조화된 순환 펩타이드의 맞춤형 설계로 확장될 수 있습니다.
-------------------------------------------
좋아요, 수집 및 전달에 오신 것을 환영합니다!
다음에 만나요!