Hace unos días, un compañero de clase preguntó sobre la implementación de un método en un artículo. Después de leer este artículo
qPCR, básicamente es pura letra, excepto la verificación. Hoy intentaré reproducirlo. Reaparece al azar. Si los datos de lectura no son buenos, puede darse por vencido. Espero que a todos les guste, miren y envíen apoyo.

文章标题: Investigación de un modelo de predicción y firma génica del microambiente relacionado con la inmunidad a la hipoxia para la fibrosis pulmonar idiopática.
doi: 10.3389 / fimmu.2021.629854
proceso
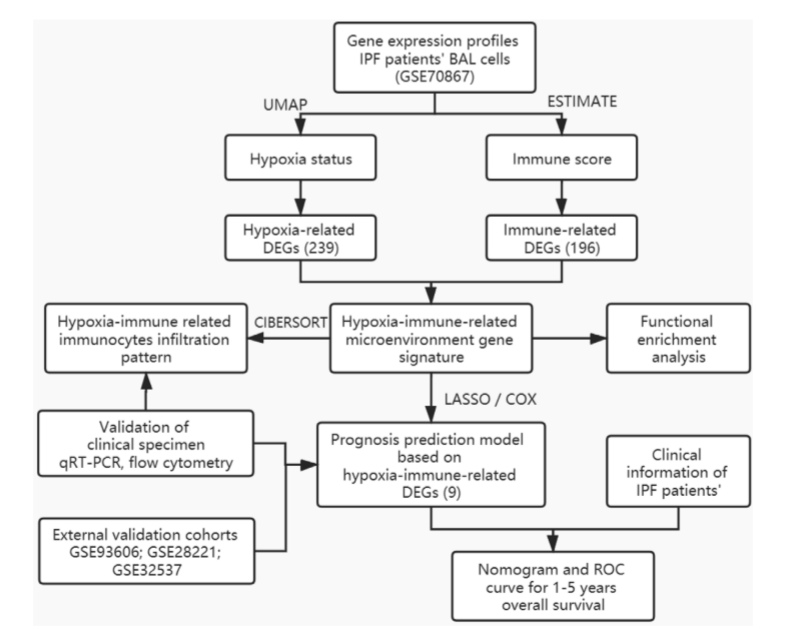
Muestra de datos y recogida de códigos
Dale me gusta, lee este artículo, compártelo con tu círculo de amigos 集赞10个y 保留30分钟envía una captura de pantalla a WeChat para mzbj0002recibirlo. Los miembros VIP de 2022 lo recibirán gratis .
Proyecto VIP Canoe Notes 2022
derechos e intereses:
Datos de muestra y código de todos los tweets en Canoe Notes en 2022 (incluida la mayor parte de 2021).

Canoa Notas Grupo de Intercambio de Investigación Científica .
Compra a mitad de precio
跟着Cell学作图系列合集(tutorial gratis + colección de códigos)|Sigue a Cell para aprender a dibujar una colección de series .
PEAJE:
99¥/persona . Puede agregar WeChat: mzbj0002transferir dinero o dar una recompensa directamente al final del artículo.

Descarga de datos GEO
GSE70866Hay dos GPL, que deben extraerse y anotarse por separado.
rm(list = ls())
BiocManager::install("GEOquery")
library(GEOquery)
eSet <- getGEO(GEO = 'GSE70866',
destdir = '.',
getGPL = F)
# 提取表达矩阵exp
exp1 <- exprs(eSet[[1]]) #GPL14550
exp2 <- exprs(eSet[[2]]) #GPL17077
exp1[1:4,1:4]
dim(exp1)
dim(exp2)
#exp = log2(exp+1)
# 提取芯片平台编号
gpl1 <- eSet[[1]]@annotation
gpl2 <- eSet[[2]]@annotation
gpl1
gpl2
## GPL注释
library(devtools)
install_github("jmzeng1314/idmap3")
## 下载后本地安装
## devtools::install_local("idmap3-master.zip")
library(idmap3)
ids_GPL14550=idmap3::get_pipe_IDs('GPL14550')
head(ids_GPL14550)
ids_GPL17077=idmap3::get_pipe_IDs('GPL17077')
head(ids_GPL17077)Las matrices de expresión génica se anotaron y fusionaron.
library(dplyr)
exp1 <- data.frame(exp1)
exp1$probe_id = row.names(exp1)
exp1 <- exp1 %>%
inner_join(ids_GPL14550,by="probe_id") %>% ##合并探针信息
dplyr::select(-probe_id) %>% ##去掉多余信息
dplyr::select(symbol, everything()) %>% #重新排列
mutate(rowMean =rowMeans(.[grep("GSM", names(.))])) %>% #求出平均数
arrange(desc(rowMean)) %>% #把表达量的平均值按从大到小排序
distinct(symbol,.keep_all = T) %>% # 留下第一个symbol
dplyr::select(-rowMean) #去除rowMean这一列
exp2 <- data.frame(exp2)
exp2$probe_id = row.names(exp2)
exp2 <- exp2 %>%
inner_join(ids_GPL17077,by="probe_id") %>%
dplyr::select(-probe_id) %>%
dplyr::select(symbol, everything()) %>%
mutate(rowMean =rowMeans(.[grep("GSM", names(.))]))
arrange(desc(rowMean)) %>%
distinct(symbol,.keep_all = T) %>%
dplyr::select(-rowMean)
exp = exp1 %>%
inner_join(exp2,by="symbol") %>% ##合并探针信息
tibble::column_to_rownames(colnames(.)[1]) # 把第一列变成行名并删除
# 先保存一下
save(exp, eSet, file = "GSE70866.Rdata")
# load('GSE70866.Rdata')
# install.packages("devtools")
# 提取临床信息
pd1 <- pData(eSet[[1]])
pd2 <- pData(eSet[[2]])
## 筛选诊断为IPF的样本
pd1 = subset(pd1,characteristics_ch1.1 == 'diagnosis: IPF')
pd2 = subset(pd2,characteristics_ch1.1 == 'diagnosis: IPF')
exp_idp = exp[,c(pd1$geo_accession,pd2$geo_accession)]corrección por lotes
## 批次校正
BiocManager::install("sva")
library('sva')
## 批次信息
batch = data.frame(sample = c(pd1$geo_accession,pd2$geo_accession),
batch = c(pd1$platform_id,pd2$platform_id))PCA antes de la corrección por lotes
#install.packages('FactoMineR')
#install.packages('factoextra')
library("FactoMineR")
library("factoextra")
pca.plot = function(dat,col){
df.pca <- PCA(t(dat), graph = FALSE)
fviz_pca_ind(df.pca,
geom.ind = "point",
col.ind = col ,
addEllipses = TRUE,
legend.title = "Groups"
)
}
pca.plot(exp_idp,factor(batch$batch))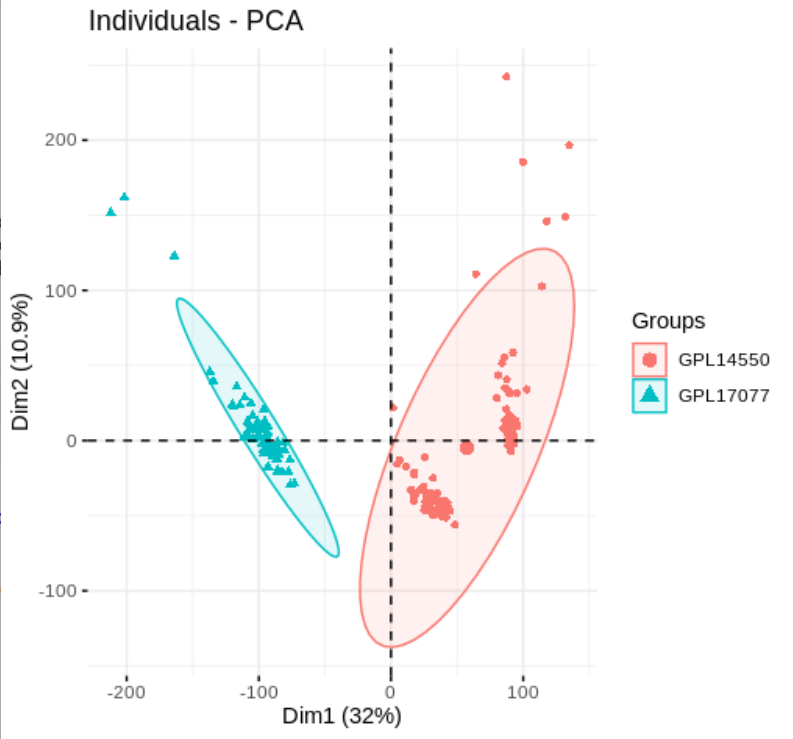
corrección por lotes
## sva 批次校正
combat_exp <- ComBat(dat = as.matrix(log2(exp_idp+1)),
batch = batch$batch)
pca.plot(combat_exp,factor(batch$batch))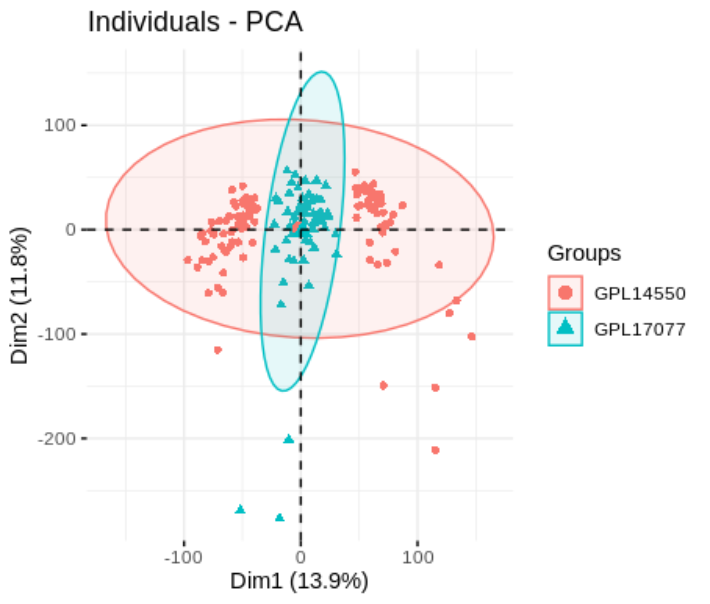
## 保存校正后的基因表达矩阵便于后续分析
save(combat_exp, eSet, file = "GSE70866.Rdata")Nota: Debido a que es solo una reproducción superficial, puede haber algunos errores o fallas en el proceso, espero que todos puedan criticarlo y corregirlo.
Contenido pasado
Sigue a Cell para aprender a mapear | Mapa de volcanes versión avanzada
(Tutorial gratuito + Colección de códigos)|Siga a Cell para aprender Colección de series de dibujos
Preguntas y respuestas | ¿Cómo dibujar bellas ilustraciones en papeles?
Sigue a Nat Commun para aprender a dibujar | 2. Gráfico de línea de tiempo
Sigue a Nat Commun para aprender a mapear | 3. Histograma de apilamiento de abundancia de especies
