1. Prinzip
Der eingeschränkte kubische Spline (RCS) ist eine der gebräuchlichsten Methoden zur Analyse nichtlinearer Beziehungen. RCS verwendet eine kubische Funktion, um die Kurven zwischen verschiedenen Knoten anzupassen und sie reibungslos zu verbinden, wodurch der Prozess der Anpassung der gesamten Kurve und des Testens ihrer Linearität erreicht wird. Wie Sie sich vorstellen können, ist die Anzahl der Knoten im RCS für die Anpassungsergebnisse sehr wichtig. Normalerweise werden 3 Knoten für kleine Stichproben mit weniger als 30 Stichproben und 5 Knoten für große Stichproben verwendet.
2.R-Implementierung
1.cox kehrt zurück
#Used for RCS(Restricted Cubic Spline)
#我们使用rms包
library(ggplot2)
library(rms)
library(survminer)
library(survival)Hier verwenden wir die Lungendaten im Überlebenspaket
#####基于cox回归
#这里用survival包里的lung数据集来做范例分析
head(lung)
# inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
# 3 306 2 74 1 1 90 100 1175 NA
# 3 455 2 68 1 0 90 90 1225 15
# 3 1010 1 56 1 0 90 90 NA 15
# 5 210 2 57 1 1 90 60 1150 11
# 1 883 2 60 1 0 100 90 NA 0
# 12 1022 1 74 1 1 50 80 513 0
#status: censoring status 1=censored, 2=dead
#sex: Male=1 Female=2
#ph.ecog:医生对患者的体能状态评级
#ph.karno:医生对患者的另一种体能状态评级karnofsky
#pat.karno:患者karnofsky自评
#meal.cal:摄入卡路里
#wt.loss:过去半年体重减轻
# 对数据进行打包,整理
dd <- datadist(lung) #为后续程序设定数据环境
options(datadist='dd') #为后续程序设定数据环境
#用AIC法计算不同节点数选择下的模型拟合度来决定最佳节点数
for (knot in 3:10) {
fit <- cph(Surv(time,status) ~ rcs(meal.cal,knot) + sex+age , x=TRUE, y=TRUE,data=lung)
tmp <- extractAIC(fit)
if(knot==3){AIC=tmp[2];nk=3}
if(tmp[2]<AIC){AIC=tmp[2];nk=knot}
}
nk #3
#cox回归中自变量对HR的rcs
fit <- cph(Surv(time,status) ~ rcs(meal.cal,3) + sex+age , x=TRUE, y=TRUE,data=lung)#大样本5节点,小样本(<30)3节点
#比例风险PH假设检验,p>0.05满足假设检验
cox.zph(fit,"rank")
#非线性检验,p<0.05为有非线性关系
anova(fit)
#这里的结果是
# Wald Statistics Response: Surv(time, status)
#
# Factor Chi-Square d.f. P
# meal.cal 0.42 2 0.8113
# Nonlinear 0.09 1 0.7643,呈线性
# sex 6.61 1 0.0101
# age 1.99 1 0.1582
# TOTAL 10.29 4 0.0358
#查看各meal.cal对应的HR值
HR<-Predict(fit, meal.cal,fun=exp)
head(HR)
#画图
ggplot()+
geom_line(data=HR, aes(meal.cal,yhat),
linetype="solid",size=1,alpha = 0.7,colour="#0070b9")+
geom_ribbon(data=HR,
aes(meal.cal,ymin = lower, ymax = upper),
alpha = 0.1,fill="#0070b9")+
theme_classic()+
geom_hline(yintercept=1, linetype=2,size=1)+
labs(title = "Lung Cancer Risk", x="Age", y="HR (95%CI)") Das Ergebnis wird wie in der Abbildung dargestellt dargestellt:
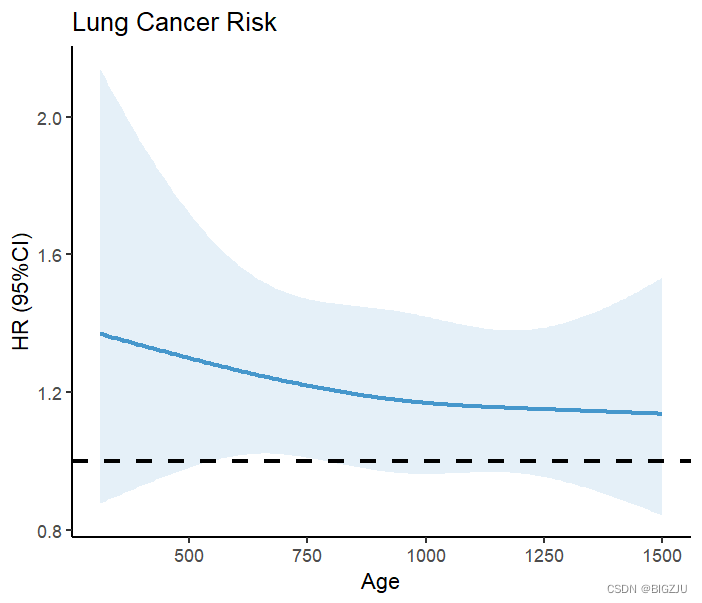
Die Ergebnisse von anova(fit) und der visuellen Präsentation zeigen einen linearen Zusammenhang zwischen der Energieaufnahme über die Nahrung und der Mortalität unter RCS.
Suchen wir ein weiteres nichtlineares Beispiel. Dieses Beispiel verwendet die Doppelpunktdaten des Survival-Pakets:
#结肠癌病人数据
Colon <- colon
Colon$sex <- as.factor(Colon$sex)#1 for male,0 for female
Colon$etype <- as.factor(Colon$etype-1)
dd <- datadist(Colon)
options(datadist='dd')
for (knot in 3:10) {
fit <- cph(Surv(time,etype==1) ~ rcs(age,knot) +sex , x=TRUE, y=TRUE,data=Colon)
tmp <- extractAIC(fit)
if(knot==3){AIC=tmp[2];nk1=3}
if(tmp[2]<AIC){AIC=tmp[2];nk1=knot}
}
nk1 #3
fit <- cph(Surv(time,etype==1) ~ rcs(age,3) +sex , x=TRUE, y=TRUE,data=Colon)
cox.zph(fit,"rank")
anova(fit)
# Wald Statistics Response: Surv(time, etype == 1)
#
# Factor Chi-Square d.f. P
# age 6.90 2 0.0317
# Nonlinear 6.32 1 0.0120,非线性
# sex 1.20 1 0.2732
# TOTAL 7.54 3 0.0565
HR<-Predict(fit, age,fun=exp)
head(HR)
ggplot()+
geom_line(data=HR, aes(age,yhat),
linetype="solid",size=1,alpha = 0.7,colour="#0070b9")+
geom_ribbon(data=HR,
aes(age,ymin = lower, ymax = upper),
alpha = 0.1,fill="#0070b9")+
theme_classic()+
geom_hline(yintercept=1, linetype=2,size=1)+
geom_vline(xintercept=47.35176,size=1,color = '#d40e8c')+#查表HR=1对应的age
geom_vline(xintercept=65.26131,size=1,color = '#d40e8c')+
labs(title = "Colon Cancer Risk", x="Age", y="HR (95%CI)") Das Ergebnis ist wie folgt: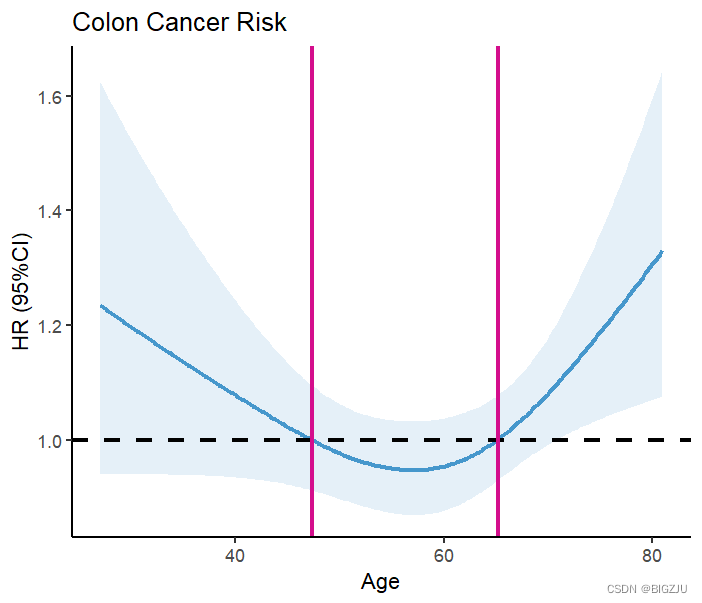
Die Ergebnisse von anova(fit) und die visuelle Darstellung zeigen, dass das Beispiel einen nichtlinearen Zusammenhang aufweist
Wir können auch gruppierte Recherchen und visuelle Präsentationen durchführen, indem wir einfach die Parameter in der Funktion Predict() ändern
HR1 <- Predict(fit, age, sex=c('0','1'),
fun=exp,type="predictions",
conf.int = 0.95,digits =2)
HR1
ggplot()+
geom_line(data=HR1, aes(age,yhat, color = sex),
linetype="solid",size=1,alpha = 0.7)+
geom_ribbon(data=HR1,
aes(age,ymin = lower, ymax = upper,fill = sex),
alpha = 0.1)+
scale_color_manual(values = c('#0070b9','#d40e8c'))+
scale_fill_manual(values = c('#0070b9','#d40e8c'))+
theme_classic()+
geom_hline(yintercept=1, linetype=2,size=1)+
labs(title = "Colon Cancer Risk", x="Age", y="HR (95%CI)")
Ergebnis:
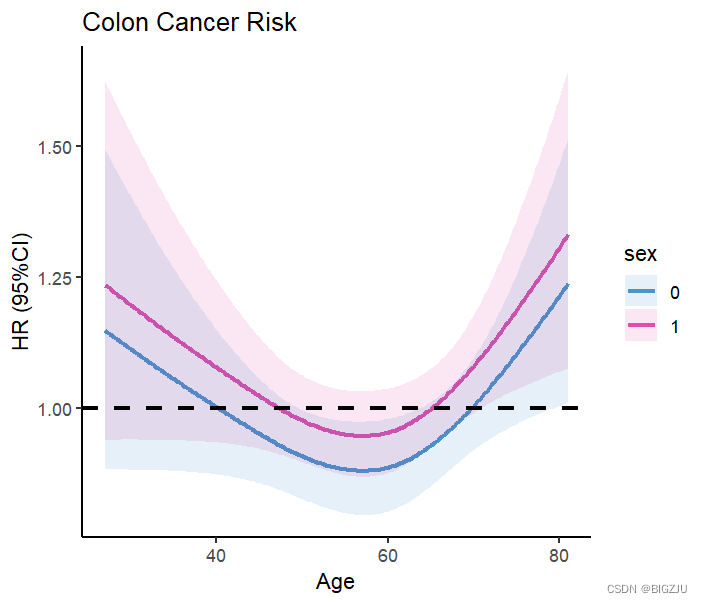
2.Logistische Regression
Wenn anstelle der Cox-Regression die logistische Regression verwendet wird, ist das Gesamtergebnis nahezu dasselbe. Sie müssen lediglich das Modell ersetzen.
#####基于logistic回归的rcs
#建模型
fit <-lrm(status ~ rcs(age, 3)+sex,data=lung)
OR <- Predict(fit, age,fun=exp)
#画图
ggplot()+
geom_line(data=OR, aes(age,yhat),
linetype="solid",size=1,alpha = 0.7,colour="#0070b9")+
geom_ribbon(data=OR,
aes(age,ymin = lower, ymax = upper),
alpha = 0.1,fill="#0070b9")+
theme_classic()+
geom_hline(yintercept=1, linetype=2,size=1)+
geom_vline(xintercept=38.93970,size=1,color = '#d40e8c')+ #查表OR=1对应的age
labs(title = "Lung Cancer Risk", x="Age", y="OR (95%CI)")3. Lineare Regression
Das Gleiche gilt für die lineare Regression
#####基于线性回归的rcs
fit <- ols(meal.cal ~rcs(age,3)+sex,data=lung)
Kcal <- Predict(fit,age)
#画图
ggplot()+
geom_line(data=Kcal, aes(age,yhat),
linetype="solid",size=1,alpha = 0.7,colour="#0070b9")+
geom_ribbon(data=Kcal,
aes(age,ymin = lower, ymax = upper),
alpha = 0.1,fill="#0070b9")+
theme_classic()+
labs(title = "RCS", x="Age", y="Kcal")