在SCI文章中,交互效应表格(通常是表五)能为文章锦上添花,增加文章的信服力,增加结果的可信程度,还能进行数据挖掘。什么是亚组,通常就是特殊类型人群,比如男女,种族等,就是说你的数据放入特殊人群中结果还可靠吗?如果在各个特殊人群中,你的结果很稳定,说明你的结论很可靠。如果亚组的结论和你的数据数据结论相反,你可以拿来做个新论题。还可以比较不同亚组之间有无区别,比如做了心脏支架和没做支架的区别,可以发现很多新思路,易于数据挖掘。

交互效应表我在既往文章《R语言手把手教你制作一个交互效应表》已经介绍怎么制作了,详细的可以去看一下。在既往文章《scitb5函数1.7版本(交互效应函数P for interaction)发布----用于一键生成交互效应表》中,我发布了本人编写的scitb5函数1.7版本,scitb5函数可以用于普通加权数据,但是不能用于复杂加权数据,因此我重新写了关于NHANES数据的亚组交互效应函数。
下面我来演示一下
导入一个我自己下载的NHANES数据,这里要注意一下,导入数据方法也是很重要的(很多人都是这里错的),因为我是基于R的基础数据框data.frame格式编程的,有些R包导入数据带有格式会报错(如haven包),比较稳妥的方法是像我这样,先使用EXCEL把数据另存为csv格式,然后按我下面方法导入。或者你使用as.data.frame()把数据强制转成数据框应该也可以。这里删除缺失值只是为了演示方便,不具有实际意义。
library(survey)
bc<-read.csv("E:/r/test/subtext.csv",sep=',',header=TRUE)
bc <- na.omit(bc)
str(bc)
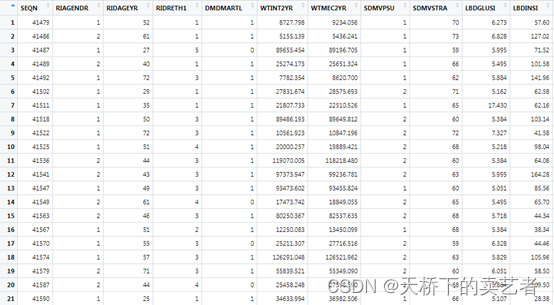 我介绍一下数据,SEQN:序列号,RIAGENDR, # 性别, RIDAGEYR, # 年龄,RIDRETH1, # 种族,DMDMARTL, # 婚姻状况,WTINT2YR,WTMEC2YR, # 权重,SDMVPSU, # psu,SDMVSTRA,# strata,LBDGLUSI, #血糖mmol表示,LBDINSI, #胰岛素( pmmol/L),PHAFSTHR #餐后血糖,LBXGH #糖化血红蛋白,SPXNFEV1, #FEV1:第一秒用力呼气量,SPXNFVC #FVC:用力肺活量,ml(估计肺容量),LBDGLTSI #餐后2小时血糖,factor.FVC是我把肺活量分为了2分类,方便用于测试。
我介绍一下数据,SEQN:序列号,RIAGENDR, # 性别, RIDAGEYR, # 年龄,RIDRETH1, # 种族,DMDMARTL, # 婚姻状况,WTINT2YR,WTMEC2YR, # 权重,SDMVPSU, # psu,SDMVSTRA,# strata,LBDGLUSI, #血糖mmol表示,LBDINSI, #胰岛素( pmmol/L),PHAFSTHR #餐后血糖,LBXGH #糖化血红蛋白,SPXNFEV1, #FEV1:第一秒用力呼气量,SPXNFVC #FVC:用力肺活量,ml(估计肺容量),LBDGLTSI #餐后2小时血糖,factor.FVC是我把肺活量分为了2分类,方便用于测试。
把分类变量转成因子
bc$DMDMARTL<-ifelse(bc$DMDMARTL==1,1,0)
bc$RIAGENDR<-as.factor(bc$RIAGENDR)
bc$RIDRETH1<-as.factor(bc$RIDRETH1)
bc$DMDMARTL<-as.factor(bc$DMDMARTL)
bc$factor.FVC<-as.factor(bc$factor.FVC)
建立抽样调查函数
bcSvy2<- svydesign(ids = ~ SDMVPSU, strata = ~ SDMVSTRA, weights = ~ WTMEC2YR,
nest=TRUE,data = bc)
设定协变量和交互变量,这里要注意一下cov1指协变量,Interaction为交互变量,交互变量
cov1<-c("DMDMARTL","RIDRETH1","RIDAGEYR","LBXGH")
Interaction<-c("DMDMARTL","RIDRETH1")
导入函数svyscitb5函数
source("E:/r/test/1.4svyscitb5.R")
成功导入后应该显示7个函数
函数的主体为
svy.scitb5 (data,x,y,Interaction,cov=NULL,time=NULL,family=NULL,method=NULL,
ids=NULL,strata=NULL,weights=NULL,svydstr=NULL)
我来解释一下data是数据,必须数据框形式,x是你研究的目标变量,y是你的结局变量,Interaction是你的分层变量,这个必须是分类变量并转成因子,cov是你的协变量,在我的设定中cov是要包含Interaction的,这也符合我们的习惯。统一使用family=“glm”,svydstr这里填入调查函数,目前支持逻辑回归和线性回归,cox回归目前还没支持。如果你使用过我写的scitb5函数,这个函数使用起来完全没有压力。
先来个分类变量的,假设我研究的变量x是种族,y变量是肺活量,想了解在这个分层的关系
out<-svy.scitb5(data=bc,x="RIAGENDR",y="factor.FVC",Interaction=Interaction,cov = cov1,family="svyglm",
svydstr=bcSvy2) #x分类,Y分类
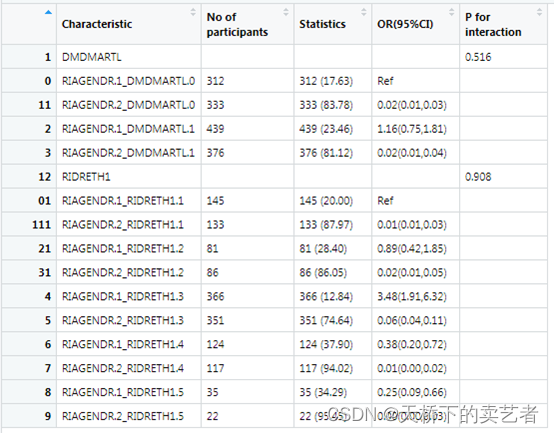
上图我要解释一下,做分类变量的时候需要设定一个参考,在DMDMARTL这个分层比较的时候,RIAGENDR.1_DMDMARTL.0是被认定做参考的,什么意思呢?就是当RIAGENDR等于1和DMDMARTL等于0这个亚组的人群是被默认为做参考比较的,其他组都是和它进行比较,分类变量进行亚组交互的时候,分类最好不要太多,要不数据会很大,而且有些层分不到数据就会显示数据缺失NA。
一键生成森林图
plotforest(out)
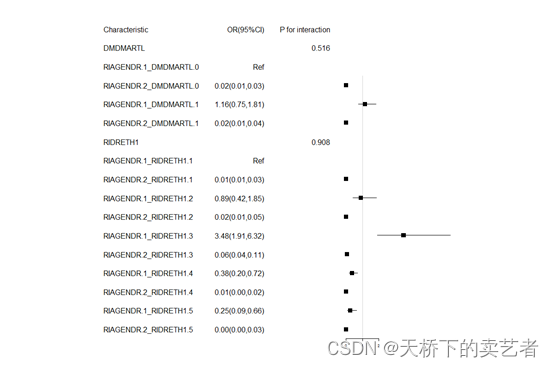
换个Y是连续变量的,这里生成的应该是β,我们可以看到,函数自己对数据类型进行了判断了
out<-svy.scitb5(data=bc,x="LBDINSI",y="SPXNFVC",Interaction=Interaction,cov = cov1,family="svyglm",
svydstr=bcSvy2) #x连续,Y连续
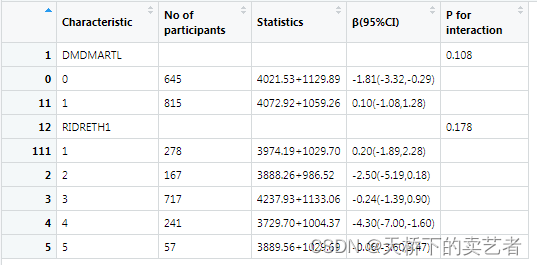
Y是连续变量是绘制不了森林图的,因为有负值,就算绘制出来也很怪,这里我就不弄了。
plotforest(out)

下面来个x连续,Y分类的
out<-svy.scitb5(data=bc,x="LBDINSI",y="factor.FVC",Interaction=Interaction,cov = cov1,family="svyglm"
,svydstr=bcSvy2) #x连续,Y分类
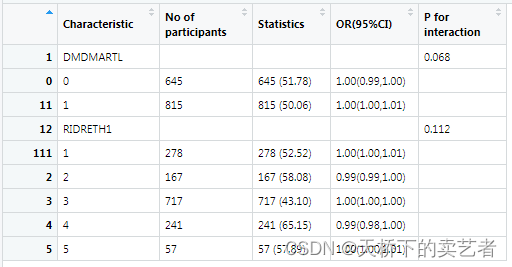
绘制森林图
plotforest(out)

下面我介绍一下svy.scitb5 函数的method参数,这个参数是用来计算P for interaction值的,它有(“LRT”, “Wald”)两个选项,,LRT是使用加权偏差法,Wald是Wald检验,对于嵌套模型使用LRT就可以了,所以平时默认的是LRT,我们可以试一下两个参数有什么不同。
out<-svy.scitb5(data=bc,x="LBDINSI",y="factor.FVC",Interaction=Interaction,cov = cov1,family="svyglm",method="Wald",
svydstr=bcSvy2) #x连续,Y分类
out<-svy.scitb5(data=bc,x="LBDINSI",y="factor.FVC",Interaction=Interaction,cov = cov1,family="svyglm",method="LRT",
svydstr=bcSvy2) #x连续,Y分类
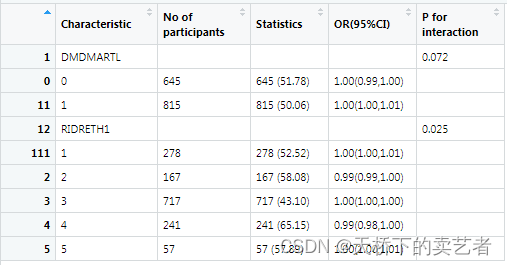
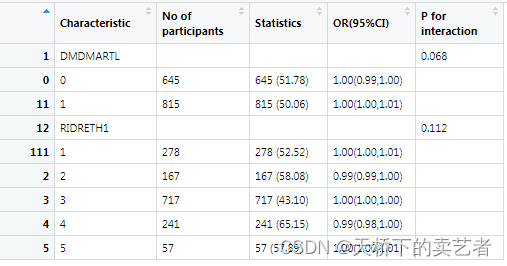
这个参数平时你不用理也不用填。为什么我要提这个东西呢?因为有时候你的模型数据极端值太小,使用LRT方法算不出来,然后咱们需要改一下,平时一般不用理。我们使用一个粉丝的数据来演示一下,这里就不解释数据意思了。
fenxi<-read.csv("E:/r/fensi/fenxiR.csv",sep=',',header=TRUE)
fenxi$Y<- as.factor(fenxi$Y)
fenxi$Y<- as.factor(fenxi$X3)
fenxi$Y<- as.factor(fenxi$X5)
fenxi$Y<- as.factor(fenxi$X6)
fenxi$Y<- as.factor(fenxi$X7)
fenxi$Y<- as.factor(fenxi$X8)
fenxi$Y<- as.factor(fenxi$X9)
fenxi$Y<- as.factor(fenxi$X10)
fenxi$Y<- as.factor(fenxi$X11)
fenxi$Y<- as.factor(fenxi$X12)
fenxi$Y<- as.factor(fenxi$X13)
fenxi$Y<- as.factor(fenxi$X1)
fenxi$Y<- as.factor(fenxi$X16)
fenxi$Y<- as.factor(fenxi$X17)
fenxi[,c("Y","X3", "X5", "X6", "X7", "X8",
"X9", "X10", "X11", "X12", "X13","X1","X16", "X17")] <- lapply(fenxi[,c("Y","X3", "X5", "X6", "X7", "X8",
"X9", "X10", "X11", "X12", "X13","X1","X16", "X17")], factor)
nhans<- svydesign(data=fenxi, ids=~SDMVPSU,strata = ~SDMVSTRA,
weights=~quanzhong,nest = TRUE)
cov1<-c("X2", "X3","X5", "X6", "X7", "X8",
"X9", "X10", "X11", "X12", "X13","X15","X16", "X17")
Interaction<-c("X3","X5","X6","X7", "X8", "X9", "X10", "X11", "X12", "X13","X16","X17")
咱们看一下常规方法
out<-svy.scitb5(fenxi,x="X",y="Y",Interaction=Interaction,cov = cov1,family="svyglm",
svydstr=nhans)

这个时候需要换一个方法
out<-svy.scitb5(fenxi,x="X",y="Y",Interaction=Interaction,cov = cov1,family="svyglm",method="Wald",
svydstr=nhans)
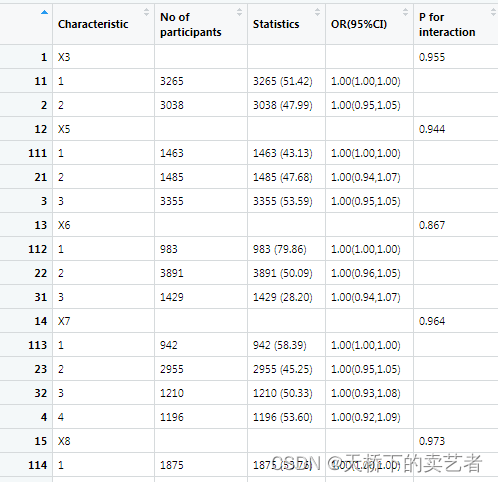
我这里要说一下,这两种方法都是survey包中的方法,不是我自己生成的,我只是调用而已,最后画图
plotforest(out)

最后我原创声明一下,本函数全套代码全部由我设计,手工编写,你可以随意使用和修改,但是用于商业用途必须征得我的同意授权。
获取svy.scitb5函数请看这篇文章
NHANES数据(复杂调查数据)亚组交互函数1.4尝鲜版(P for interaction)发布—用于一键生成交互效应表